Ce package fournit des modèles U-net formés pour la segmentation pulmonaire. Pour l'instant, quatre modèles sont disponibles :
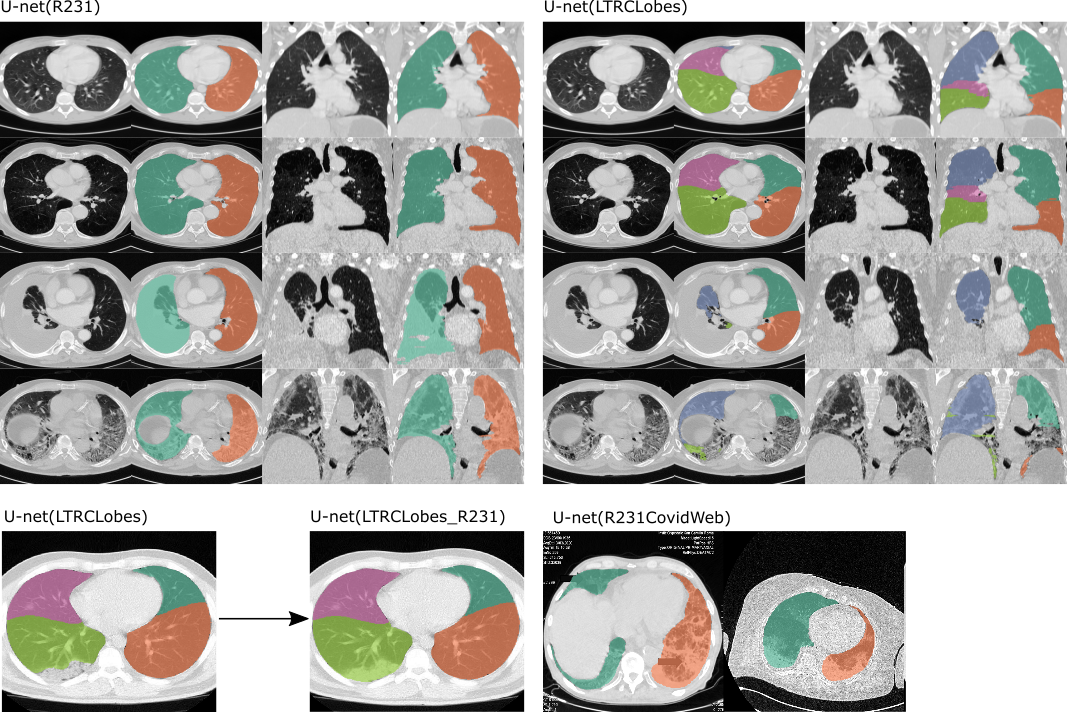
U-net (R231) : ce modèle a été formé sur un ensemble de données vaste et diversifié qui couvre un large éventail de variabilité visuelle. Le modèle effectue une segmentation sur des tranches individuelles, extrait séparément le poumon droit et gauche, y compris les poches d'air, les tumeurs et les épanchements. La trachée ne sera pas incluse dans la segmentation pulmonaire. https://doi.org/10.1186/s41747-020-00173-2
U-net (LTRCLobes) : ce modèle a été formé sur un sous-ensemble de l'ensemble de données LTRC. Le modèle effectue une segmentation des lobes pulmonaires individuels, mais donne des performances limitées lorsque des pathologies denses sont présentes ou lorsque les fissures ne sont pas visibles sur chaque tranche.
U-net (LTRCLobes_R231) : cela exécutera les modèles R231 et LTRCLobes et fusionnera les résultats. Les faux négatifs de LTRCLobes seront remplis par les prédictions R231 et mappés sur une étiquette voisine. Les faux positifs de LTRCLobes seront supprimés. Le processus de fusion nécessite beaucoup de calculs et peut, en fonction des données et des résultats, prendre jusqu'à plusieurs minutes par volume.
U-net(R231CovidWeb)
Exemples des deux modèles appliqués . Gauche : U-net (R231), fera la distinction entre les poumons gauche et droit et inclura les zones très denses telles que les épanchements (troisième rangée), les tumeurs ou les fibroses sévères (quatrième rangée). À droite : U-net (LTRLobes), fera la distinction entre les lobes pulmonaires mais n’inclura pas les zones très denses. LTRCLobes_R231 fusionnera les résultats LTRCLobes et R231. R231CovidWeb est formé avec des données COVID-19 supplémentaires.
Sémantique de sortie :
Deux modèles d'étiquettes (gauche-droite) :
1 = Poumon droit
2 = Poumon gauche
Cinq modèles d'étiquettes (Lobes pulmonaires) :
1 = Lobe supérieur gauche
2 = Lobe inférieur gauche
3 = Lobe supérieur droit
4 = Lobe médian droit
5 = Lobe inférieur droit
Pour des recherches plus passionnantes sur les données de tomodensitométrie pulmonaire, consultez le site Web de notre groupe de recherche : https://www.cir.meduniwien.ac.at/research/lung/
Si vous utilisez ce code ou l'un des modèles formés dans votre travail, veuillez vous référer à :
Hofmanninger, J., Prayer, F., Pan, J. et al. La segmentation automatique des poumons en imagerie de routine est avant tout un problème de diversité des données et non un problème de méthodologie. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Cet article contient une description détaillée de l'ensemble de données utilisé, une évaluation approfondie du modèle U-net (R231) et une comparaison avec les méthodes de référence.
pip install lungmask
ou
pip install git+https://github.com/JoHof/lungmask
Sous Windows, selon votre configuration, il peut être nécessaire d'installer torch au préalable : https://pytorch.org
Le temps d'exécution entre l'inférence prise en charge par le CPU uniquement et par le GPU varie considérablement. En utilisant le GPU, le traitement d'un volume ne prend que quelques secondes, l'utilisation du CPU uniquement prendra plusieurs minutes. Pour utiliser le GPU, assurez-vous que votre installation de torche prend en charge CUDA. En cas d'erreurs de mémoire insuffisante, réduisez la taille du lot à 1 avec l'argument facultatif --batchsize 1
lungmask INPUT OUTPUT
Si INPUT pointe vers un fichier, le fichier sera traité. Si INPUT pointe vers un répertoire, la série DICOM sera recherchée dans le répertoire. Le plus grand volume trouvé (en termes de nombre de voxels) sera utilisé pour calculer le masque pulmonaire. OUTPUT est le nom du fichier de sortie. Tous les formats ITK sont pris en charge.
Choisissez un modèle :
Le U-net (R231) sera utilisé par défaut. Cependant, vous pouvez spécifier un modèle alternatif tel que LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Pour des options supplémentaires, tapez :
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image doit être un objet SimpleITK.
Chargez un modèle alternatif comme ceci :
inferer = LMInferer(modelname="R231CovidWeb")
Pour utiliser la capacité de fusion de modèles pour (par exemple LTRCLobes_R231), utilisez :
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
Depuis la version 0.2.9, les tableaux numpy sont pris en charge comme volumes d'entrée. Ce mode suppose que le tableau numpy d'entrée a le format suivant pour chaque axe :
Le modèle fonctionne uniquement sur des tranches complètes. La coupe à traiter doit montrer le poumon entier et le poumon doit être entouré de tissu afin d'être segmenté. Cependant, le modèle est assez stable dans les cas où le champ de vision est coupé tant que le poumon est entouré de tissus.
lungmask INPUT OUTPUT --modelname R231CovidWeb
Le modèle U-net(R231) standard fonctionne très bien pour les tomodensitogrammes COVID-19. Cependant, les collections de tranches et de rapports de cas provenant du Web sont souvent recadrées, annotées ou codées dans des formats d'image classiques, de sorte que les valeurs originales de l'unité Hounsfield (HU) ne peuvent être estimées. Les données de formation du modèle U-net (R231CovidWeb) ont été complétées par des tranches de COVID-19 qui ont été cartographiées à partir de formats d'imagerie classiques vers HU. Les données ont été collectées et préparées par MedSeg (//medicalsegmentation.com/covid19/). Bien que le U-net standard (R231) ait montré de très bons résultats pour ces images, il peut y avoir des cas pour lesquels ce modèle produira des segmentations légèrement améliorées. Notez que vous devez mapper les images sur HU lorsque vous utilisez des images du Web. Cet article de blog décrit comment procéder. Vous pouvez également définir la balise --noHU . 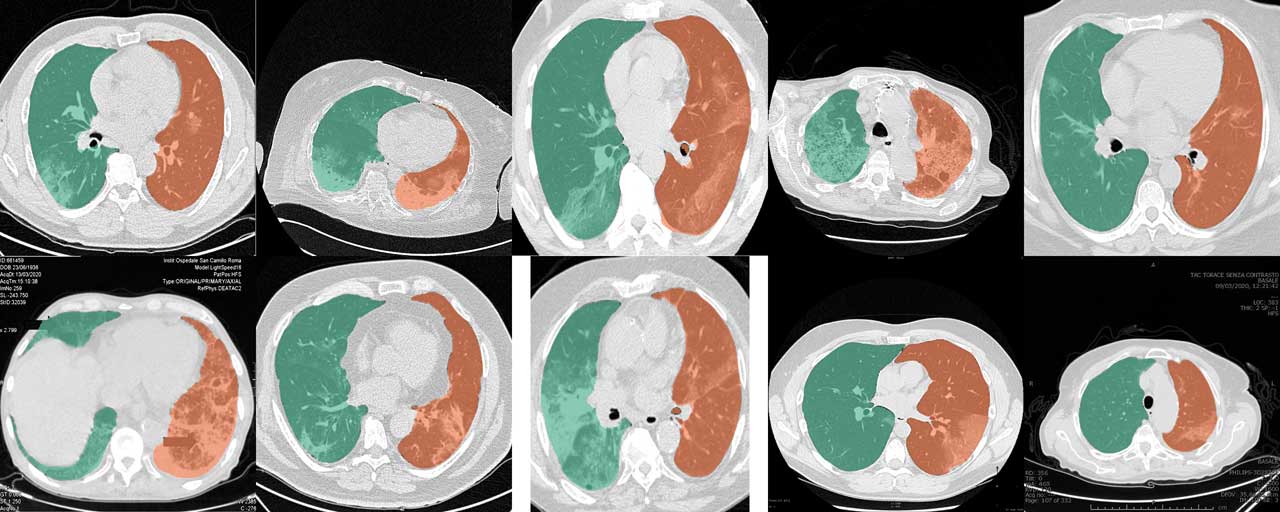
Cette fonctionnalité n'est disponible que dans les versions comprises entre 0.2.5 et 0.2.14. Depuis la version 0.2.5, ces images sont prises en charge. Utilisez la balise --noHU si vous traitez des images qui ne sont pas codées en HU. Gardez à l’esprit que les modèles ont été formés sur des tomodensitogrammes appropriés codés en HU. Les résultats sur des images recadrées, annotées, décalées de très haute et très faible intensité peuvent ne pas être très fiables. Lors de l'utilisation de la balise --noHU , seules des tranches uniques peuvent être traitées.