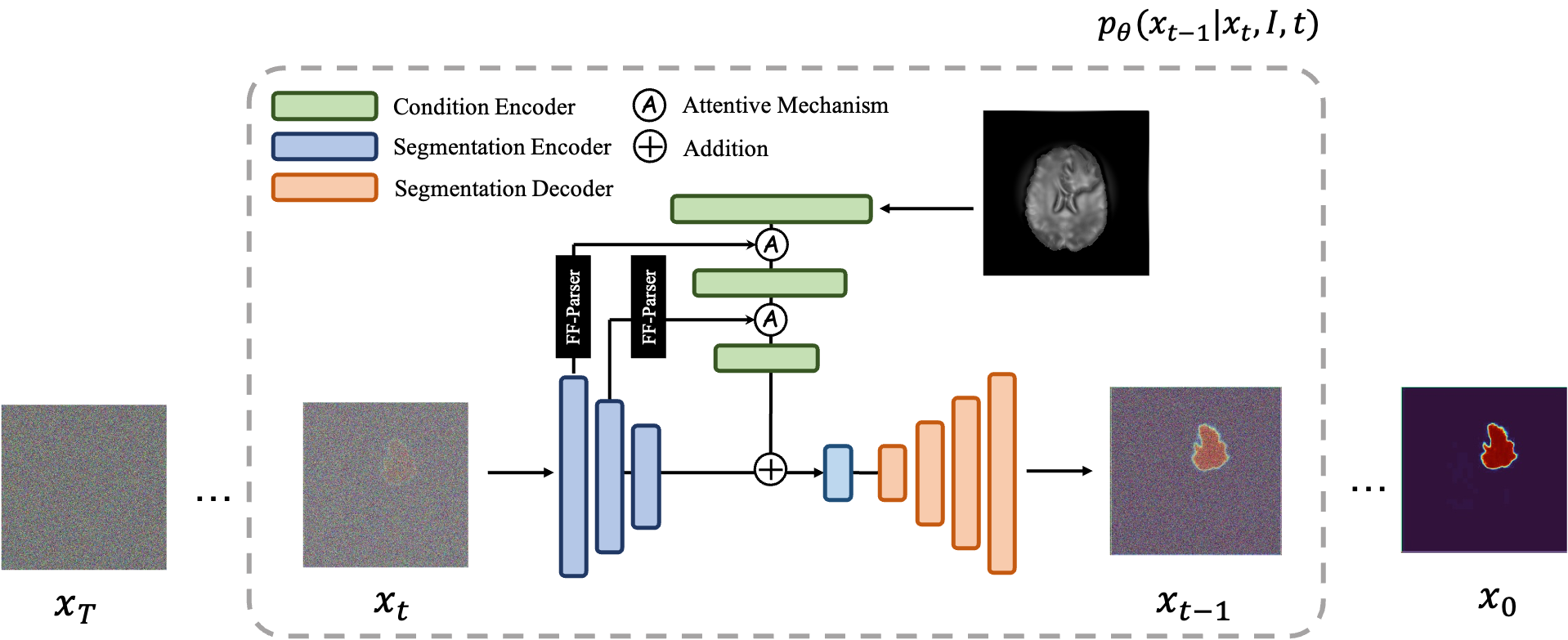
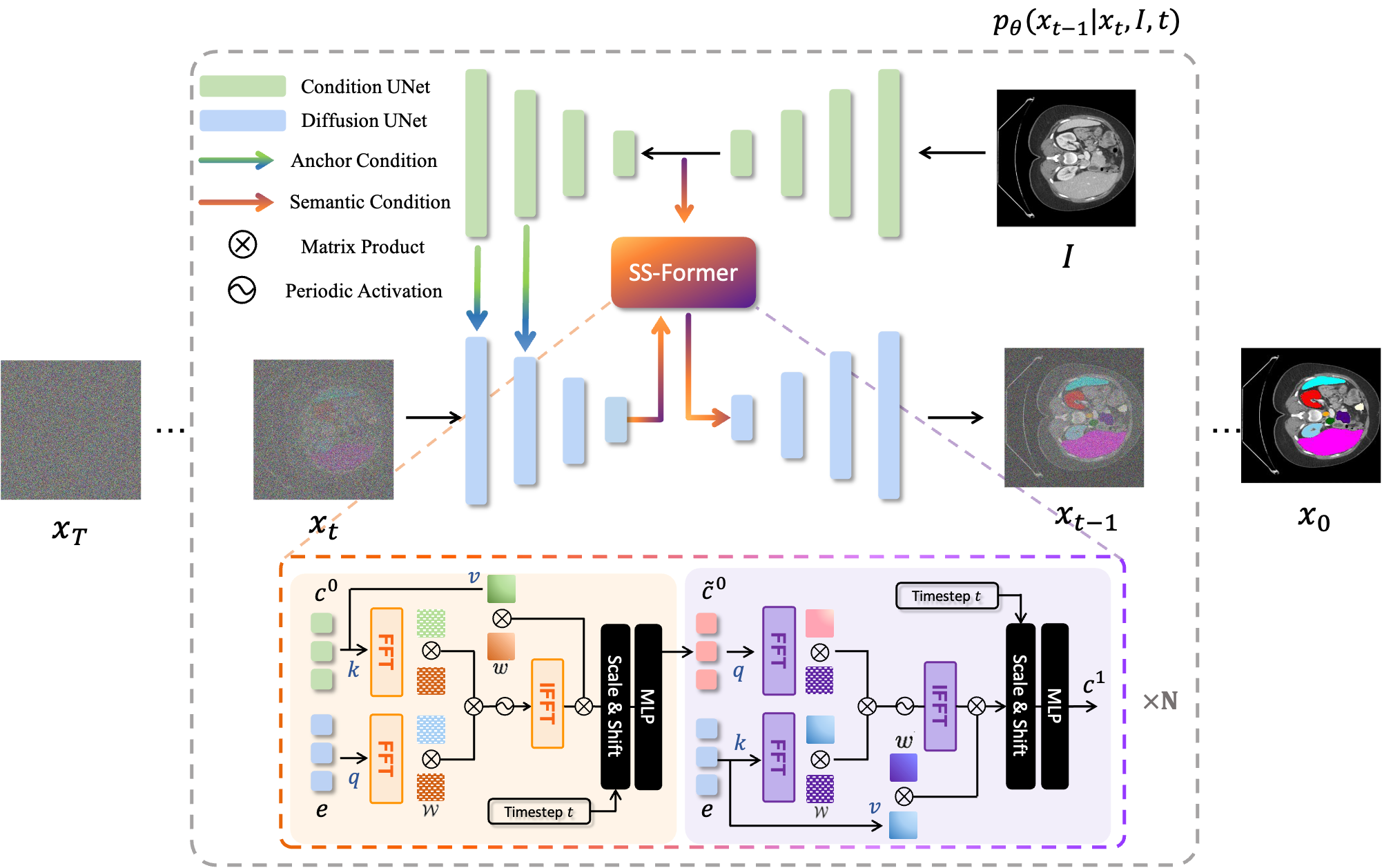
MedSegDiff, un cadre basé sur un modèle probabiliste de diffusion (DPM) pour la segmentation des images médicales. L'algorithme est élaboré sur notre article MedSegDiff : Segmentation d'images médicales avec modèle probabiliste de diffusion et MedSegDiff-V2 : Segmentation d'images médicales basée sur la diffusion avec transformateur.
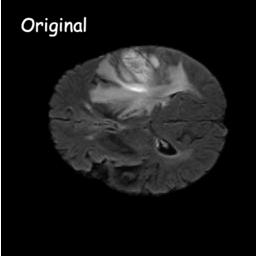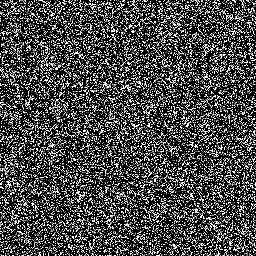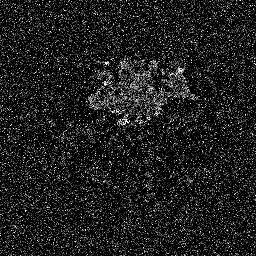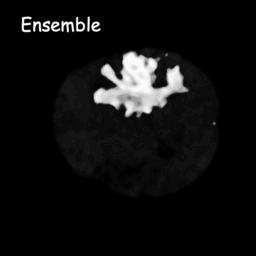 Les modèles de diffusion fonctionnent en détruisant les données d'entraînement par l'ajout successif de bruit gaussien, puis en apprenant à récupérer les données en inversant ce processus de bruit. Après la formation, nous pouvons utiliser le modèle de diffusion pour générer des données en faisant simplement passer un bruit échantillonné de manière aléatoire via le processus de débruitage appris. Dans ce projet, nous étendons cette idée à la segmentation d'images médicales. Nous utilisons l'image originale comme condition et générons plusieurs cartes de segmentation à partir de bruits aléatoires, puis effectuons un assemblage sur celles-ci pour obtenir le résultat final. Cette approche capture l’incertitude des images médicales et surpasse les méthodes précédentes sur plusieurs points de référence.
Les modèles de diffusion fonctionnent en détruisant les données d'entraînement par l'ajout successif de bruit gaussien, puis en apprenant à récupérer les données en inversant ce processus de bruit. Après la formation, nous pouvons utiliser le modèle de diffusion pour générer des données en faisant simplement passer un bruit échantillonné de manière aléatoire via le processus de débruitage appris. Dans ce projet, nous étendons cette idée à la segmentation d'images médicales. Nous utilisons l'image originale comme condition et générons plusieurs cartes de segmentation à partir de bruits aléatoires, puis effectuons un assemblage sur celles-ci pour obtenir le résultat final. Cette approche capture l’incertitude des images médicales et surpasse les méthodes précédentes sur plusieurs points de référence.
 |  |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Pour la formation, exécutez : python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Pour l'échantillonnage, exécutez : python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Pour l'évaluation, exécutez python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
Par défaut, les échantillons seront enregistrés dans ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Pour la formation, exécutez : python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Pour l'échantillonnage, exécutez : python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
Il est simple d’exécuter MedSegDiff sur les autres ensembles de données. Écrivez simplement un autre fichier de chargeur de données après ./guided_diffusion/isicloader.py ou ./guided_diffusion/bratsloader.py . Bienvenue pour ouvrir les problèmes si vous rencontrez un problème. Il serait apprécié que vous puissiez contribuer à vos extensions d'ensemble de données. Contrairement aux images naturelles, les images médicales varient beaucoup en fonction des différentes tâches. Étendre la généralisation d'une méthode nécessite les efforts de chacun.
Pour former un modèle fin, c'est-à-dire MedSegDiff-B dans l'article, définissez les hyperparamètres du modèle comme suit :
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
hyperparamètres de diffusion comme :
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Pour accélérer l’échantillonnage :
--diffusion_steps 50 --dpm_solver True
fonctionner sur plusieurs GPU :
--multi-gpu 0,1,2 (for example)
hyperparamètres de formation comme :
--lr 5e-5 --batch_size 8
et définissez --num_ensemble 5 dans l'échantillonnage.
L'exécution d'environ 100 000 étapes de formation sera convergée sur la plupart des ensembles de données. Notez que même si les pertes ne diminuent pas dans la plupart des étapes ultérieures, la qualité des résultats continue de s'améliorer. Un tel processus est également observé sur les autres applications DPM, comme la génération d'images. J'espère que quelqu'un d'intelligent pourra me dire pourquoi ?.
Je publierai bientôt ses performances sous une taille de lot plus petite (adaptée pour fonctionner sur un GPU de 24 Go) pour besoin de comparaison ?.
Un paramètre pour libérer tout son potentiel est (MedSegDiff++) :
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Entraînez-le ensuite avec la taille du lot --batch_size 64 et échantillonnez-le avec le numéro d'ensemble --num_ensemble 25 .
Bienvenue pour contribuer à MedSegDiff. Toute technique pouvant améliorer les performances ou accélérer l’algorithme est appréciée. J'écris MedSegDiff V2, destiné à une publication de type revues Nature/CVPR. Je suis heureux de lister les contributeurs comme mes co-auteurs ?.
Code copié beaucoup depuis openai/improved-diffusion, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion,guided-diffusion, bigmb/Unet-Segmentation-Pytorch-Nest -of-Unets, nnUnet, lucidrains/vit-pytorch
Veuillez citer
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu