med seg diff pytorch
0.3.3
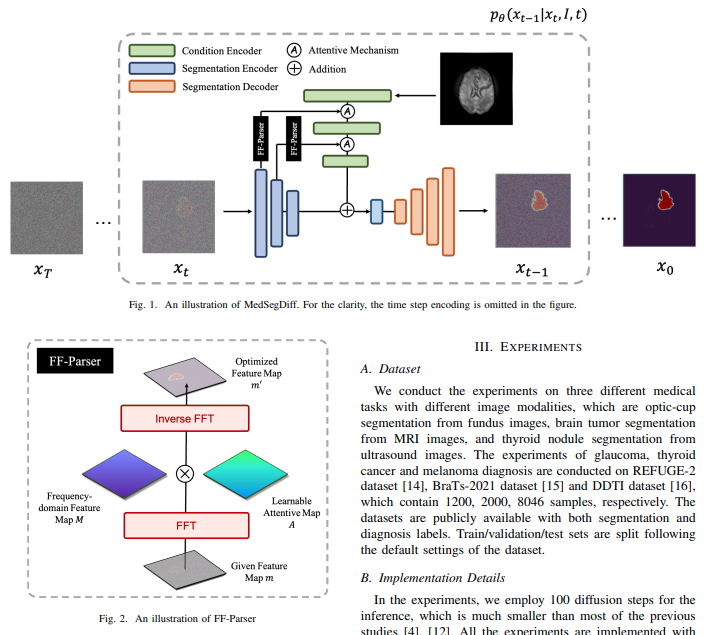
Implémentation de MedSegDiff dans Pytorch - Segmentation médicale SOTA hors Baidu à l'aide de DDPM et conditionnement amélioré au niveau des fonctionnalités, avec filtrage des fonctionnalités dans l'espace de Fourier.
StabilityAI pour son généreux parrainage, ainsi que mes autres sponsors
Isamu et Daniel pour avoir ajouté un script de formation pour un ensemble de données sur les lésions cutanées !
$ pip install med-seg-diff-pytorch import torch
from med_seg_diff_pytorch import Unet , MedSegDiff
model = Unet (
dim = 64 ,
image_size = 128 ,
mask_channels = 1 , # segmentation has 1 channel
input_img_channels = 3 , # input images have 3 channels
dim_mults = ( 1 , 2 , 4 , 8 )
)
diffusion = MedSegDiff (
model ,
timesteps = 1000
). cuda ()
segmented_imgs = torch . rand ( 8 , 1 , 128 , 128 ) # inputs are normalized from 0 to 1
input_imgs = torch . rand ( 8 , 3 , 128 , 128 )
loss = diffusion ( segmented_imgs , input_imgs )
loss . backward ()
# after a lot of training
pred = diffusion . sample ( input_imgs ) # pass in your unsegmented images
pred . shape # predicted segmented images - (8, 3, 128, 128) Commande à exécuter
accelerate launch driver.py --mask_channels=1 --input_img_channels=3 --image_size=64 --data_path= ' ./data ' --dim=64 --epochs=100 --batch_size=1 --scale_lr --gradient_accumulation_steps=4Si vous souhaitez ajouter une condition personnelle là où nous conditionnons avec le masque que nous avons jusqu'à présent, faites --self_condition
@article { Wu2022MedSegDiffMI ,
title = { MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model } ,
author = { Junde Wu and Huihui Fang and Yu Zhang and Yehui Yang and Yanwu Xu } ,
journal = { ArXiv } ,
year = { 2022 } ,
volume = { abs/2211.00611 }
} @inproceedings { Hoogeboom2023simpleDE ,
title = { simple diffusion: End-to-end diffusion for high resolution images } ,
author = { Emiel Hoogeboom and Jonathan Heek and Tim Salimans } ,
year = { 2023 }
}