
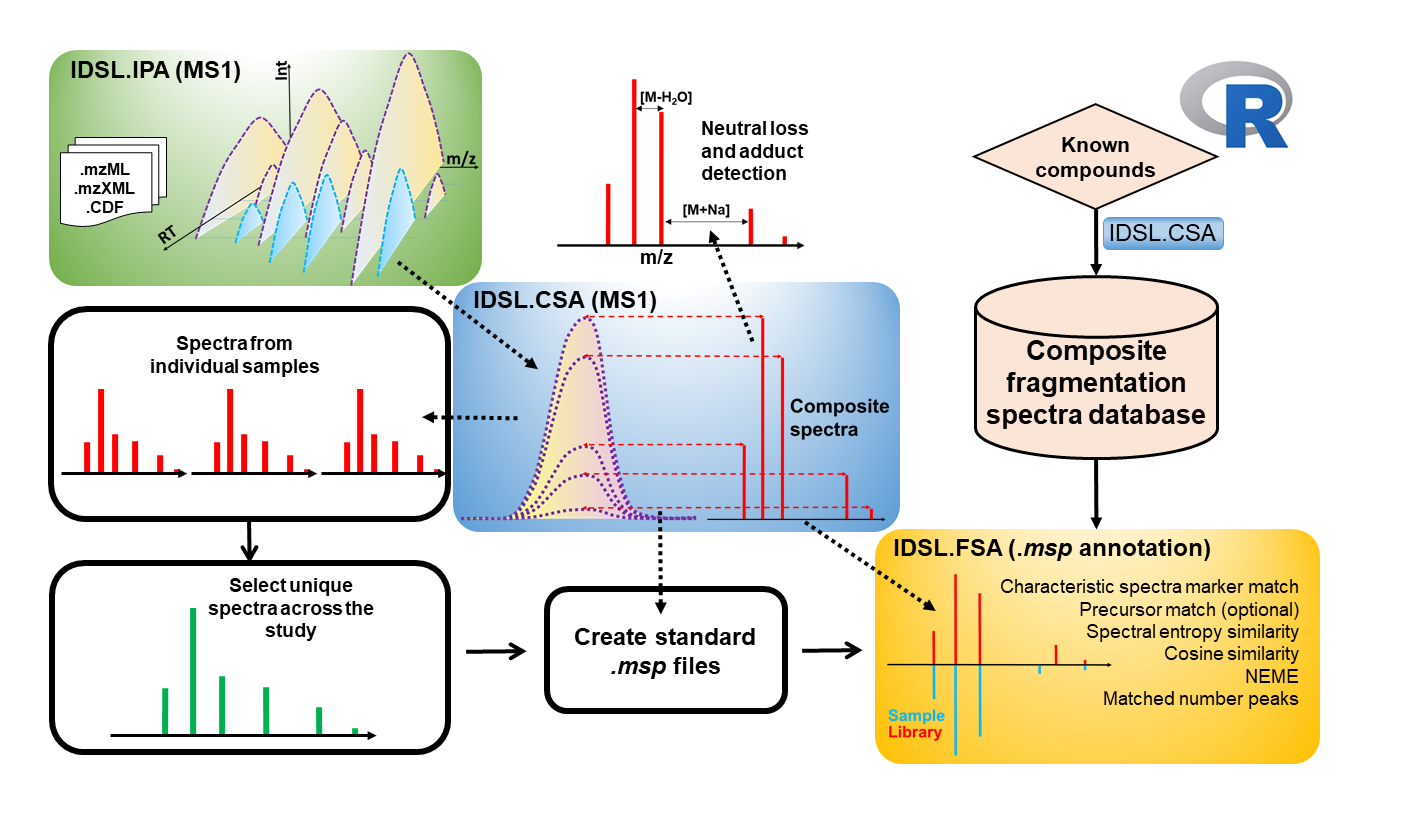
L' analyse des spectres composites (IDSL.CSA) R pour l'analyse des données de spectrométrie de masse a été développée par le Laboratoire de science des données intégré pour la métabolomique et l'exposomique (IDSL.ME) . Ce package peut être utilisé pour la déconvolution des spectres de fragmentation obtenus via diverses méthodes analytiques telles que l'analyse de déconvolution composite de déconvolution ( CSA) (CSA ), les méthodes ( DDA ) et une acquisition ( DIA ) indépendante des données (DDA) et diverses méthodes indépendantes des données, y compris MS E , Fragmentation All-ion (AIF) et analyses SWATH-MS. L'objectif du package IDSL.CSA est d'aider à rationaliser le processus d'analyse des données et à améliorer l'annotation globale de la structure chimique dans les domaines de la métabolomique et de l'exposomique.
Start dans la feuille de calcul du paramètre iDSL.CSA. install.packages("IDSL.CSA")
Avant de traiter vos données de spectrométrie de masse ( MZXML , MZML , NETCDF ) à l'aide du flux de travail IDSL.CSA, les données de spectrométrie de masse doivent être traitées à l'aide du flux de travail IDSL.IPA pour acquérir des informations chromatographiques des pics ( M / Z-RT ). Lorsque les informations chromatographiques sur les pics alignées individuelles et agrégées ont été générées à l'aide du flux de travail IDSL.IPA, téléchargez la feuille de calcul du paramètre idsl.csa et sélectionnez les paramètres en conséquence, puis utilisez cette feuille de calcul comme entrée pour le flux de travail IDSL.CSA:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Suivez ces étapes pour une étude de cas rapide (n = 33) ST002263 qui possède des données Orbrid Orbitrap hybrides HF Thermo Q perçues dans les modes Hilic-ESI-POS / NEG.
Traiter les données de spectrométrie de masse brutes et les informations chromatographiques en utilisant la méthode décrite pour idsl.ipa
L' analyse des spectres composites nécessite 39 paramètres distribués dans 5 sections distinctes pour une analyse à grande échelle. Pour cette étude, utilisez les valeurs de paramètre par défaut présentées dans la feuille de calcul du paramètre iDSL.CSA. Ensuite, fournissez des informations pour
2.1. Sélectionnez oui pour param0001 dans l'onglet Start pour traiter uniquement le flux de travail CSA .
2.2. CSA0005 pour l'adresse d'emplacement des données HRMS (données MS1 Niveau HRMS)
2.3. CSA0008 pour l'adresse du répertoire peaklists généré par le flux de travail idsl.ipa
2.4. CSA0009 pour l'adresse du répertoire peak_alignment généré par le flux de travail idsl.ipa
2.5. CSA0011 pour l'emplacement de la sortie (fichiers et eics.
2.6. Vous pouvez également augmenter le nombre de threads de traitement à l'aide de CSA0004 en fonction de votre puissance de calcul
Exécutez cette commande dans la console ou le terminal R / rstudio:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Vous pouvez analyser les résultats à l'adresse que vous avez fournie pour CSA0011 .
4.1. CSA_MSP inclut le fichier .msp
4.2. CSA_ADDUCT_Annotation comprend des pics de pics avec des informations d'addition potentielles
4.3. Peak_alignment_subset comprend des sous-ensembles de tables de pointe alignées pour les principaux ions dans chaque cluster CSA
4.4. Aligned_Spectra_Table comprend des informations pour l'agrégation CSA sur le tableau aligné
[1] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL.CSA: Analyse des spectres composites pour l'annotation chimique des ensembles de données de métabolomique non ciblés. Analytical Chemistry , 2023 , 95 (25) , 9480–9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, DK Idsl. L'IPA caractérise l'espace chimique organique dans les ensembles de données LC / HRMS non ciblés. Journal of Proteome Research , 2022 , 21 (6) , 1485-1494.