Nous sommes heureux d'annoncer que la taille de l'index sur le disque est en baisse de 8 fois et en mémoire de 4 fois en raison du passage à un seul type d'index FM (2Bit.64 au lieu de 2BIT.64 et 8BIT.32) et de compression 8X du tableau de suffixes. Par exemple, pour le génome humain, la taille de l'indice sur le disque est tombée à ~ 10 Go de ~ 80 Go et l'empreinte mémoire est jusqu'à ~ 10 Go de ~ 40 Go. Il y a une réduction substantielle du temps d'IO indexe en raison de la réduction et pratiquement aucun impact sur les performances sur la cartographie de lecture. En raison de cette modification de la structure de l'indice (dans Commit # 4B59796, 10 octobre 2020), vous devrez reconstruire l'index.
Ajout du drapeau MC dans le fichier SAM de sortie dans Commit A591E22. La sortie doit correspondre à BWA-MEM Version 0.7.17 d'origine.
En tant que Commit E0AC59E, nous avons un sous-module Git Safestriblib. Pour l'obtenir, utilisez - réécursif lors du clonage ou utilisez "Git Submodule Init" et "Git Subdodule Update" dans un référentiel déjà cloné (voir ci-dessous pour plus de détails).
# Utilisez des binaires précompilés (recommandés) curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2 | TAR JXF - BWA-MEM2-2.2.1_X64-LINUX / BWA-MEM2 INDEX REF.FA BWA-MEM2-2.2.1_X64-LINUX / BWA-MEM2 MEM REF.FA LEAD1.FQ LEAD2.FQ> OUT.SAM # Compile à partir de Source (non recommandée pour les utilisateurs généraux) # Obtenez le clone SourceGit - Recursif Https: // github.com/bwa-mem2/bwa-mem2cd bwa-mem2 # orgit clone https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 sous-module git init Mise à jour du sous-module GIT # compilation et runmake ./bwa-mem2
L'outil BWA-MEM2 est la prochaine version de l'algorithme BWA-MEM dans BWA. Il produit un alignement identique à BWA et est ~ 1,3-3.1x plus rapidement en fonction du cas d'utilisation, de l'ensemble de données et de la machine en cours d'exécution.
La BWA d'origine a été développée par Heng Li (@ LH3). L'amélioration des performances dans BWA-MEM2 a été principalement effectuée par Vasimuddin MD (@ yuk12) et Sanchit Misra (@ Sanchit-Misra) de Parallel Computing Lab, Intel. BWA-MEM2 est distribué sous la licence du MIT.
Pour les utilisateurs généraux, il est recommandé d'utiliser les binaires précompilés de la page de version. Ces binaires ont été compilés avec le compilateur Intel et fonctionnent plus rapidement que les binaires compilés GCC. Les binaires précompilés soutiennent également indirectement la répartition du processeur. Le binaire bwa-mem2 peut choisir automatiquement l'implémentation la plus efficace en fonction de l'ensemble d'instructions SIMD disponible sur la machine en cours d'exécution. Des binaires précompilés ont été générés sur une machine CentOS7 en utilisant la ligne de commande suivante:
faire cxx = icpc multi
L'utilisation est exactement la même que l'outil BWA MEM d'origine. Voici une brève synopsys. Exécutez ./bwa-mem2 pour les commandes disponibles.
# Indexation de la séquence de référence (nécessite une mémoire 28N GB où n est la taille de la séquence de référence) ../ index bwa-mem2 [-p préfixe] <in.fasta> où <in.fasta> est le fichier Fasta de séquence de référence de la trajectoire et <fréfix> est le préfixe des noms des fichiers qui stockent l'index résultant. La valeur par défaut est dans.fasta. # Mapping # run "./bwa-mem2 mem" pour obtenir toutes Où <préfix> est le préfixe spécifié lors de la création de l'index ou du chemin vers le fichier Fasta de référence au cas où aucun préfixe n'a été fourni.
Ensembles de données:
Genome de référence: humain_g1k_v37.fasta
| Alias | Source de l'ensemble de données | Nombre de lectures | Longueur de lecture |
|---|---|---|---|
| D1 | Grand institut | 2 x 2,5 m BP | 151BP |
| D2 | SRA: SRR7733443 | 2 x 2,5 m BP | 151BP |
| D3 | SRA: SRR9932168 | 2 x 2,5 m BP | 151BP |
| D4 | SRA: SRX6999918 | 2 x 2,5 m BP | 151BP |
Détails de la machine:
Processeur: Intel (R) Xeon (R) 8280 CPU @ 2,70 GHz
OS: Centos Linux version 7.6.1810
Mémoire: 100 Go
Nous avons suivi les étapes ci-dessous pour collecter les résultats des performances:
A. Étapes de téléchargement des données:
Téléchargez la boîte à outils SRA depuis https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global
Tar xfzv Sratoolkit.2.10.5-centos_linux64.tar.gz
Télécharger D2: Sratoolkit.2.10.5-centos_linux64 / bin / fastq-dump - Split-files srr7733443
Télécharger D3: Sratoolkit.2.10.5-centos_linux64 / bin / fastq-dump - Split-files srr9932168
Télécharger D4: Sratoolkit.2.10.5-centos_linux64 / bin / fastq-dump - Split-files srx6999918
B. Étapes d'alignement:
git clone https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (en utilisant le compilateur Intel C / C ++)
ou make (en utilisant le compilateur GCC)
./bwa-mem2 Index <Ref.fa>
./bwa-mem2 mem [-t <#Threads>] <Ref.fa> <in_1.fastq> [<in_22
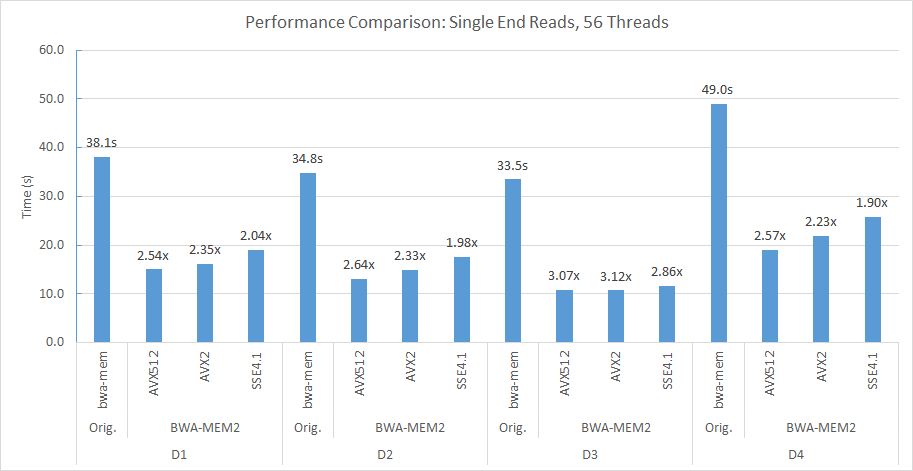
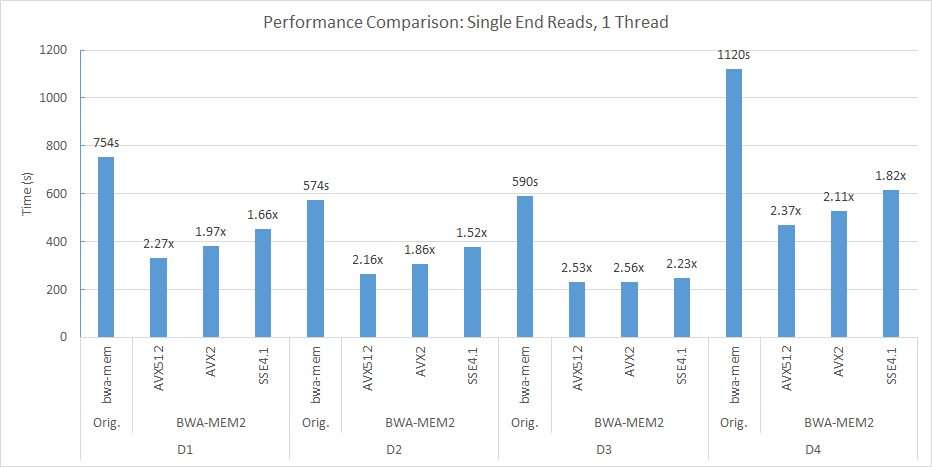
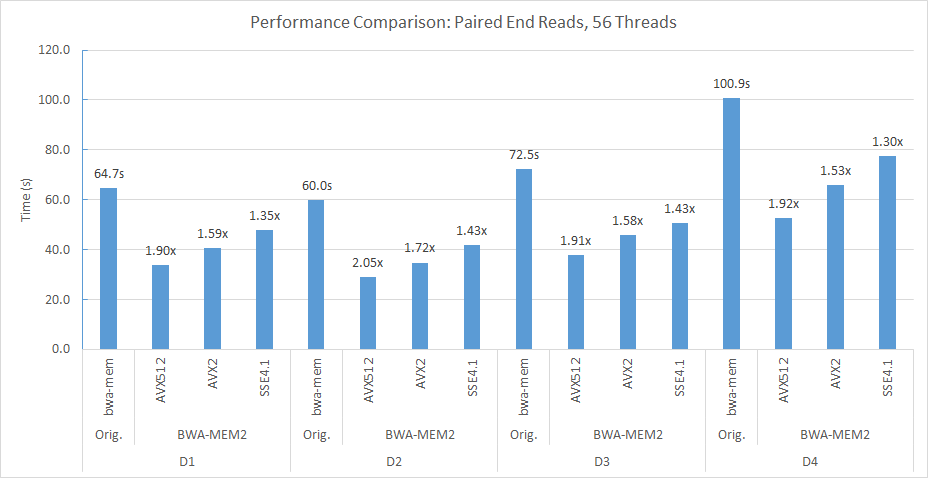
Par exemple, dans notre nœud à double socket (56 threads chacun) et le nœud de calcul NUMA double, nous avons utilisé la ligne de commande suivante pour aligner D2 sur Human_G1K_V37.Fasta Reference Genome.
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

BWA-MEM2-LISA est une version accélérée de BWA-MEM2 où nous appliquons des index savants à la phase d'ensemencement. BWA-MEM2-LISA Branch contient le code source de l'implémentation. Voici les caractéristiques de BWA-MEM2-LISA:
Exactement même sortie que BWA-MEM2.
Toutes les lignes de commande pour créer un index et le mappage de lecture sont exactement les mêmes que BWA-MEM2.
BWA-MEM2-LISA accélère la phase d'ensemencement (l'un des principaux goulots d'étranglement de BWA-MEM2) jusqu'à 4,5x par rapport à BWA-MEM2.
L'empreinte mémoire de l'indice BWA-MEM2-LISA est de ~ 120 Go pour le génome humain.
Le code est présent dans Bwa-Mem2-Lisa Branch: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
La branche ERT du référentiel BWA-MEM2 contient la base de code de l'accélération basée sur l'arbre Radix Euerated de BWA-MEM2. Le code ERT est construit sur le dessus de BWA-MEM2 (grâce au travail acharné de @ arun-sub). Voici les points forts de l'outil BWA-MEM2 basé sur ERT:
Exactement même sortie que BWA-MEM (2)
L'outil a deux drapeaux supplémentaires pour permettre l'utilisation de la solution ERT (pour la création d'index et la cartographie), sinon il fonctionne en mode Vanilla BWA-MEM2
Il utilise 1 indicateur supplémentaire pour créer un index ERT (différent de l'index BWA-MEM2) et 1 indicateur supplémentaire pour l'utilisation de cet index ERT (veuillez consulter le Readme of Ert Branch)
La solution ERT est de 10% - 30% plus rapide (testée sur la configuration ci-dessus de la machine) par rapport à la vanille BWA-MEM2 - les utilisateurs sont conseillés pour utiliser l'option -K 1000000 pour voir les accélérations
L'impression de la mémoire de l'index ERT est d'environ 60 Go
Le code est présent dans Ert Branch: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng Li, Srinivas Aluru. Accélération efficace de l'architecture de BWA-MEM pour les systèmes multicore. IEEE Symposium de traitement parallèle et distribué (IPDPS), 2019. 10.1109 / ipdps.2019.00041