
Isling est un outil pour détecter l'intégration virale ou vectorielle dans les lectures de l'extrémité appariée. Veuillez lire notre article pour plus de détails.
Si vous avez installé conda et snakemake , pour exécuter avec les données de test (incluses) localement:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
Si snakemake et singularity sont installés, vous pouvez utiliser à la place:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
Alternativement, si Docker a installé, sur macOS, vous pouvez exécuter:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
Cela utilisera le fichier de configuration et les données à l'intérieur du conteneur, et les résultats apparaîtront dans un dossier out dans votre répertoire de travail actuel. Sur Linux, vous devrez exécuter cette commande en tant que racine, et sur Windows, vous devrez ajuster la syntaxe Bind-Mount (argument -v ).
Les données d'entrée (lectures et références hôte et virale) sont spécifiées dans un fichier de configuration - pour vos propres données, vous devrez modifier l'exemple de fichier de configuration ( test/config/test.yml ) pour pointer de vos propres données. Voir configfile.md pour plus d'informations sur le format du fichier de configuration.
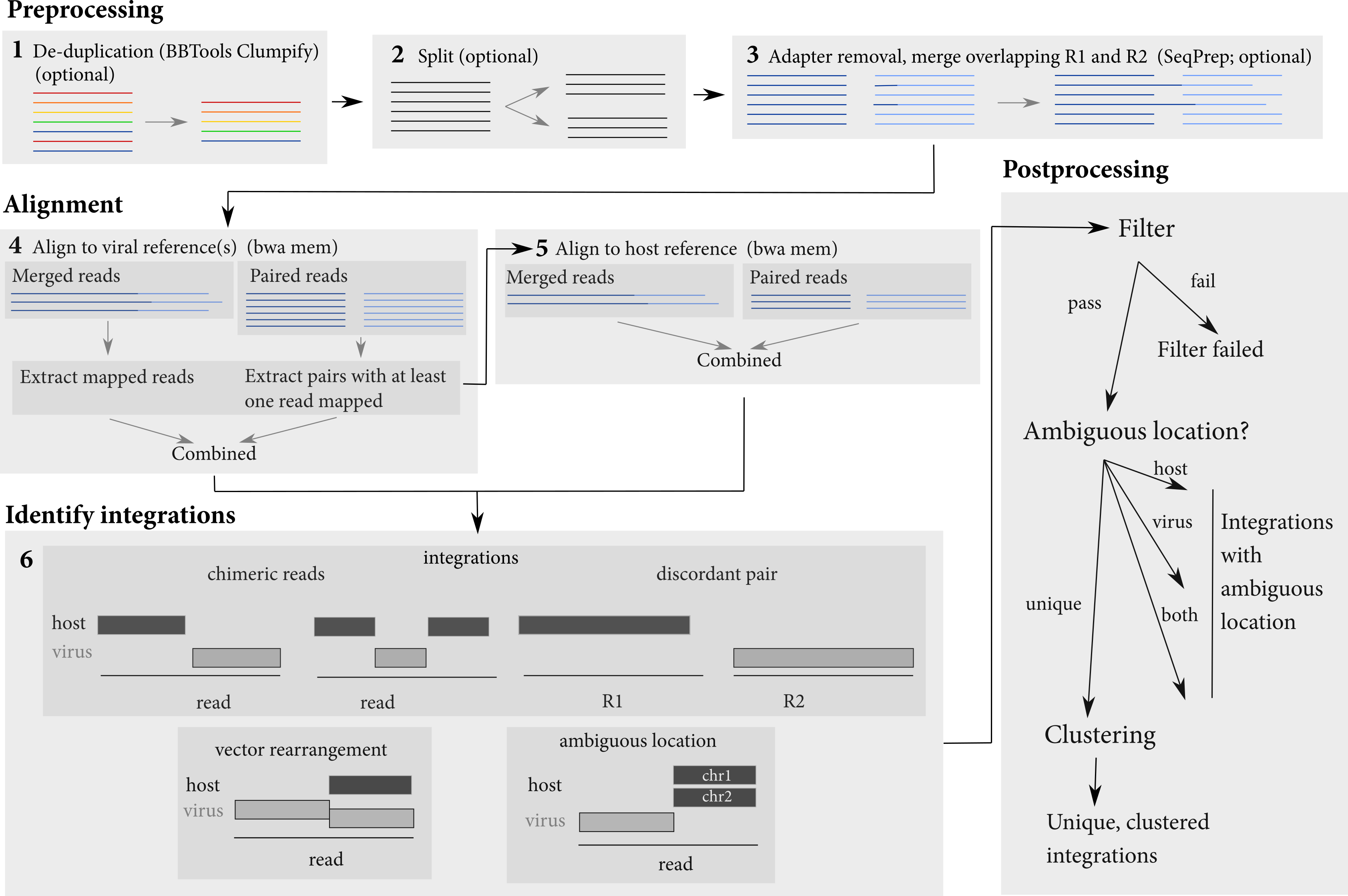
Le pipeline effectue plusieurs étapes afin d'identifier les sites d'intégration. Il prend en tant qu'ensembles de données d'entrée composés de fichiers FastQ ou de fichiers BAM. Il fait un certain prétraitement des lectures (fusion des lectures qui se chevauchent, facultative), puis les aligne sur un hôte et une séquence virale. Les lectures sont d'abord alignées sur la ou les séquences virales, puis les lectures alignées sont extraites et alignées sur l'hôte. Ces alignements sont utilisés pour identifier les intégrations virales.
Isling nécessite snakemake et de singularity (recommandée) ou conda pour fournir des dépendances. De plus, python version 3.5 ou plus et pandas sont nécessaires (ceux-ci doivent être installés automatiquement si l'installation snakemake avec conda .
Alternativley, utilisez la version docker qui contient Isling et toutes les dépendances.
Les entrées requises sont le fichier de configuration, qui spécifie les références hôte et virale / vectorielle, et des lectures sont nécessaires. Spécifiez toutes les entrées dans un fichier de configuration. Isling ne fonctionne actuellement que pour les lectures de l'extrémité appariée.
Voir le fichier configfile.md pour une description du format de ce fichier de configuration.
Isling sort des sites d'intégration dans un format séparé dans le répertoire de sortie spécifié dans le fichier de configuration.
Dans le dossier de sortie, un dossier est créé pour chaque ensemble de données dans le fichier de configuration, et pour chaque ensemble de données, les intégrations peuvent être trouvées dans le répertoire ints . Il y aura un ensemble de fichiers de sortie pour chaque échantillon.
Pour chaque échantillon, il existe un certain nombre de fichiers de sortie, qui peuvent être intéressants pour des cas d'utilisation particulières.
<sample>.<host>.<virus>.integrations.txt<sample>.<host>.<virus>.integrations.post.txt<sample>.<host>.<virus>.integrations.post.unique.txt<sample>.<host>.<virus>.integrations.post.unique.merged.txt : le résultat de la fusion des jonctions ou des jonctions qui se chevauchent avec les mêmes coordonnées dans l'hôte et le virus / vecteur. La fusion est réalisée avec uniquement les jonctions d'intégration qui ont un emplacement sans ambiguïté dans l'hôte et le virus / vecteur.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : informations sur les jonctions détectées qui ont passé tous les filtres et ont un vecteur / virus de localisation sans ambiguïté mais un emplacement ambiguë dans l'hôte<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : informations sur les jonctions détectées qui ont passé tous les filtres et ont un hôte de localisation sans ambiguïté mais une emplacement ambiguë dans le vecteur / virus<sample>.<host>.<virus>.integrations.post.both_ambig.txt : informations sur les jonctions détectées qui ont passé tous les filtres et ont un emplacement ambigu dans l'hôte et le vecteur / virus Les fichiers de sortie donnent l'emplacement des intégrations identifiées et de leurs propriétés. Les coordonnées des jonctions d'intégration sont spécifiées en termes de bases ambiguës. Autrement dit, il y a souvent un écart ou un chevauchement entre l'hôte et les parties virales d'une lecture: 
Étant donné que l'emplacement de l'intégration ne peut pas être déterminé de manière unique dans ce cas, Isling produit les coordonnées de ces bases dans le génome hôte et vecteur / virus comme emplacement de l'intégration.
À l'exception du fichier de sortie de cluster fusionné, tous les fichiers contiennent ces colonnes:
gap ou overlap pour une lecture chimique (parties hôte et virale identifiée sur la même lecture), une jonction est clean si elle n'a pas de bases ambiguës. Alternativement, si la jucntion a été identifiée dans une paire discordante, le OverlapType est discordanthv ) ou virus / hôte ( vh )+ ou -bwa ) de l'alignement de l'hôtebwa ) de l'alignement viralOverlapType est gapchimeric , une paire discordant ou est-ce une short intégration dans laquelle les deux jonctions ont été observées dans la même lecturebwa )bwa )'xxx' Pour reproduire les figures du manuscrit d'Isling, voir le ReadMe dans le répertoire benchmarking .