Proyek ini terdiri dari metode berbeda untuk mengambil sampel populasi dan evaluasi metode berbeda. Kami menyertakan banyak situasi yang dapat menyebabkan bias terhadap perkiraan tingkat infeksi berdasarkan sampel, termasuk non-responden, angka positif/negatif palsu, kemampuan profil penularan pasien selama masa infeksinya. Berdasarkan model EpiABM, paket ini juga dapat menghasilkan metode pengambilan sampel terbaik dengan menjalankan simulasi penularan penyakit untuk melihat kesalahan prediksi setiap metode pengambilan sampel.
EpiOS belum tersedia di PyPI, tetapi modul dapat diinstal secara pip secara lokal. Direktori tersebut harus diunduh terlebih dahulu ke mesin lokal Anda, dan kemudian dapat diinstal menggunakan perintah:
pip install -e .Kami juga menyarankan Anda untuk menginstal model EpiABM untuk menghasilkan data simulasi infeksi. Anda dapat mengunduh pyEpiabm terlebih dahulu ke lokasi mana pun di mesin Anda, dan kemudian dapat menginstalnya menggunakan perintah:
pip install -e path/to/pyEpiabm Dokumentasi dapat diakses melalui lencana docs di atas.
Berikut adalah diagram kelas UML untuk proyek kami: 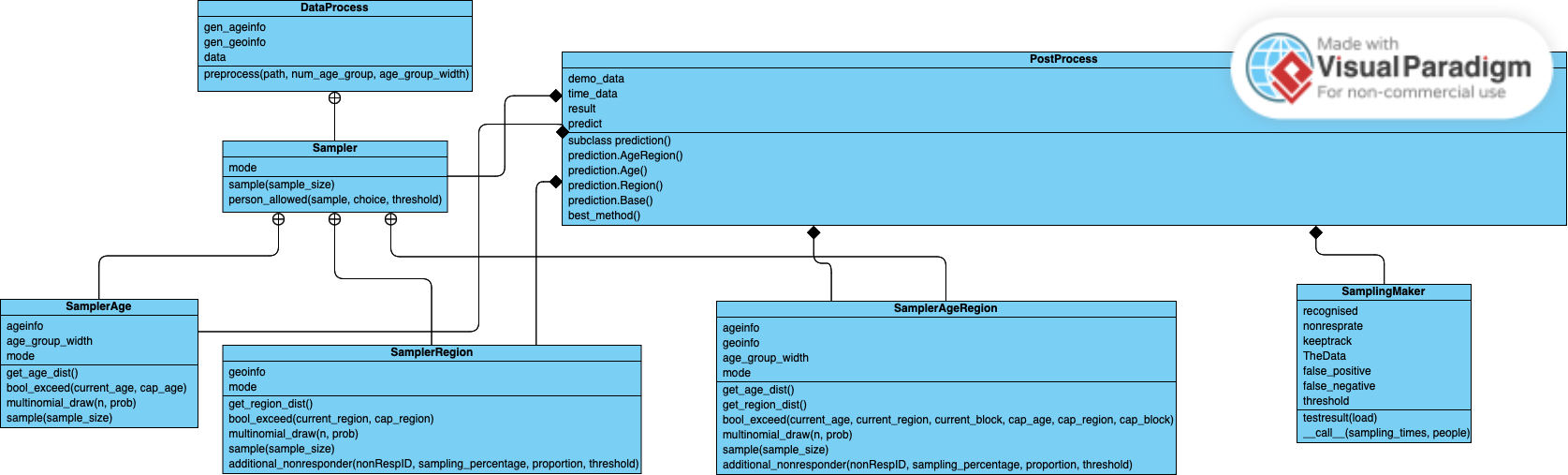
File params.py menyertakan semua parameter yang diperlukan dalam model ini. Selain itu, file dalam folder input adalah contoh file sementara yang dihasilkan selama pra-pemrosesan data. Ini akan digunakan oleh kelas sampler. Parameter data_store_path di setiap kelas sampler adalah jalur untuk menyimpan file-file ini.
PostProcess untuk menghasilkan plot Pertama, Anda perlu mendefinisikan objek PostProcess baru dan memasukkan data demografi demodata dan data timedata infeksi yang dihasilkan dari pyEpiabm. Kedua, Anda dapat menggunakan PostProcess.predict untuk melakukan prediksi berdasarkan metode pengambilan sampel yang berbeda. Anda dapat langsung memanggil metode pengambilan sampel yang ingin Anda gunakan sebagai metode; lalu tentukan titik waktu untuk mengambil sampel dan ukuran sampel. Di sini, kita akan menggunakan AgeRegion sebagai metode pengambilan sampel, [0, 1, 2, 3, 4, 5] sebagai titik waktu untuk dijadikan sampel dan 3 sebagai ukuran sampel. Terakhir, Anda dapat menentukan apakah Anda ingin mempertimbangkan non-responder dan apakah Anda ingin membandingkan hasil Anda dengan data sebenarnya dengan menentukan parameter non_responder dan comparison .
Untuk contoh kodenya, Anda dapat melihat berikut ini:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)Sekarang, sosok Anda akan disimpan ke jalur yang diberikan!
PostProcess untuk memilih metode pengambilan sampel terbaik Pertama, Anda perlu mendefinisikan objek PostProcess baru dan memasukkan data demografi demodata dan data timedata infeksi yang dihasilkan dari pyEpiabm. Kedua, Anda dapat menggunakan PostProcess.best_method untuk membandingkan kinerja berbagai metode pengambilan sampel. Anda dapat memberikan metode yang ingin Anda bandingkan; kemudian tentukan interval pengambilan sampel untuk sampel dan ukuran sampel. Ketiga, Anda dapat menentukan apakah Anda ingin mempertimbangkan non-responder dan apakah Anda ingin membandingkan hasil Anda dengan data sebenarnya dengan menentukan parameter non_responder dan comparison . Selain itu, karena metode pengambilan sampel bersifat stokastik, Anda dapat menentukan jumlah iterasi yang dijalankan untuk mendapatkan performa rata-rata. Selain itu, parallel_computation dapat diaktifkan untuk mempercepat. Terakhir, Anda dapat mengaktifkan hyperparameter_autotune untuk secara otomatis menemukan kombinasi hyperparameter terbaik.
Untuk contoh kodenya, Anda dapat melihat berikut ini:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed