最終更新日: 2024 年 9 月 30 日
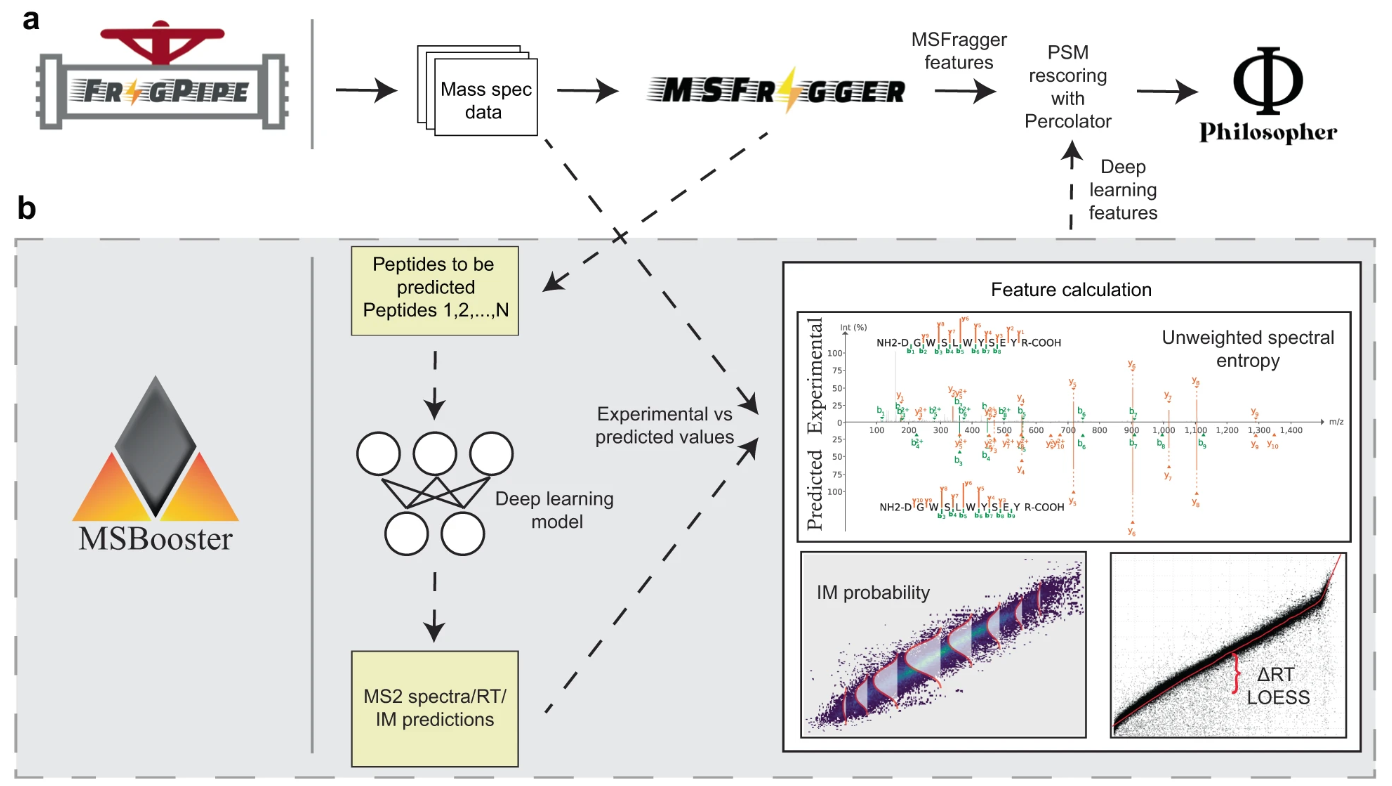
MSBooster は、ボトムアップ タンデム液体クロマトグラフィー質量分析プロテオミクス データのペプチド スペクトル マッチ (PSM) 再スコアリングにスペクトル ライブラリ予測を組み込むためのツールです。大きく分けて4つのステップに分かれます。
検索結果内の PSM からのペプチド抽出、および機械/深層学習 (ML/DL) 予測子の入力ファイルのフォーマット
予測モデルを呼び出して出力を保存する
特徴量計算
検索結果ファイルへの新機能の追加
MSBooster は、HLA イムノペプチドミクス、DDA および DIA、単一細胞プロテオミクスなど、多くの種類のデータベース検索と互換性があります。これは FragPipe に組み込まれており、そのワークフローの多くに含まれています。 MSBooster は、FragPipe-PDV などの他の FragPipe ツールを念頭に置いて開発されました。
MSBooster には、複数の入力ファイル形式とモデルを処理する機能が備わっています。
| 質量分析計の出力 |
|---|
| .mzML |
| .mgf |
| PSMファイル |
|---|
| 。ピン |
| .pepXML (進行中) |
| 予測モデル |
|---|
| ダイヤン |
| こいなモデル |
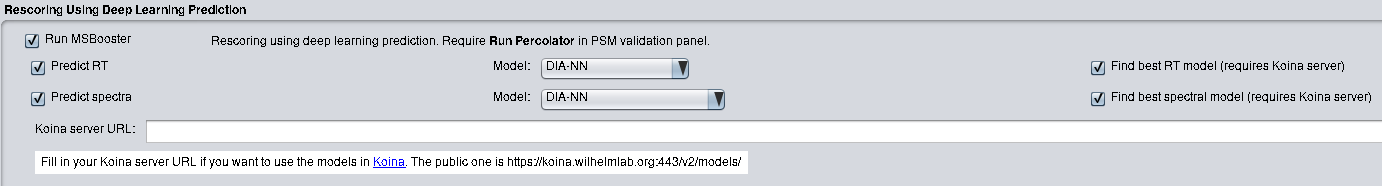
MSBooster は、Windows および Linux システムで実行できます。 FragPipe を使用する場合、FragPipe のインストール以外に他のインストール手順は必要ありません。 MSBooster は [検証] タブにあります。 「Predict RT」で保持時間機能を有効にし、「Predict spectra」で MS/MS スペクトル機能を有効にすることを選択します。分析の実行方法については、FragPipe のドキュメントを参照してください。
スタンドアロン MSBooster を使用してコマンド ラインで実行する場合は、リリースから最新の jar ファイルをダウンロードしてください。 MSBooster では、MS/MS および RT 予測のために DIA-NN も必要です。 DIA-NN をインストールし、DIA-NN 実行可能ファイルへのパスをメモしておいてください (例: Windows の場合は DiaNN.exe、Linux の場合は diann-1.8.1.8)。
次のようなコマンドを使用して MSBooster を実行できます。
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
渡す必要がある最小限のパラメータは次のとおりです。
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
これらのパラメータを個別に渡すこともできますが、paramsList ファイルの各行に 1 つずつ配置する方が簡単です。テンプレートについては、msbooster_params.txt を参照してください。
以下のパラメータは一般的に使用されるものです。 Koina 固有のパラメータは Koina のドキュメントに記載されています
paramsList (String) : この実行のパラメータを含むテキスト ファイルの場所
fragger (String) : MSFragger 実行からの fragger.params ファイルのファイル パス。 MSBooster は複数のパラメーターを読み取り、それらに基づいてフラグメント質量誤差許容値や質量オフセットなどの内部パラメーターを調整します。
outputDirectory (String) : 新しいファイルを出力する場所
editedPin (String) : MSBooster は、提供されたファイルに基づいて新しいファイルに名前を付けます。たとえば、A.pin には A_edited.pin という対応するものがあります。デフォルトの「編集済み」から変更するには、ここに新しい文字列を入力します。
renamePin (int) : 新しいピン ファイルを生成するか、古いピン ファイルを書き換えるか。ここでのデフォルトは 1 で、上書きされません。これを 0 に設定すると、古い pin ファイルが上書きされます。
deletePreds (boolean) : 実行が成功した後にモデル予測を保存するファイルを削除するかどうか。デフォルトでは false に設定されます。これらを削除したい場合は true に設定します
loadingPercent (int) : 進捗状況レポーターを使用してタスクの進捗状況を報告する頻度。デフォルトでは 10 に設定されており、10% ごとに更新が印刷されることを意味します。
numThreads (int) : 使用するスレッドの数。デフォルトでは 0 に設定されます。これは、使用可能なすべてのスレッドから 1 を引いたものを使用します。
splitPredInputFile (int) : メモリ不足エラー (137) により DIA-NN 予測が失敗した場合にのみ使用されます。デフォルトでは 1 に設定されていますが、DIA-NN 入力ファイルをいくつの小さなファイルに分割するかを指定するためにこれを増やすことができます。各ファイルは順番に予測されるため、メモリの負担が軽減されます。
plotExtension (String) : プロットのファイル形式。デフォルトでは png ですが、pdf も使用できます。
features (String) : 計算される特徴のリスト。大文字と小文字が区別され、間にスペースを入れずにカンマで区切られます。デフォルトは「predRTrealUnits、unweightedSpectralEntropy、deltaRTLOESS」です。
spectraPredFile (String) : 古いスペクトル予測 (DIA-NN や Koina など) を再利用している場合は、ここでファイルの場所を指定できます。
RTPredFile (String) : spectraPredFile と同じですが、RT 予測用です。
IMPredFile (String) : spectraPredFile と同じですが、IM 予測用です
spectraModel (String) : どのスペクトル予測モデルを使用するか
rtModel (String) : spectraModel と同じですが、RT 用です。
imModel (String) : spectraModel と同じですが、IM 用です
useSpectra (boolean) : スペクトル予測ベースの機能を使用するかどうか。デフォルトで true に設定されます
useRT (boolean) : RT 予測ベースの機能を使用するかどうか。デフォルトで true に設定されます
useIM (boolean) : IM 予測ベースの機能を使用するかどうか。デフォルトでは false に設定されます
ppmTolerance (float) : フラグメントエラー ppm 許容値 (デフォルトは 20ppm)
matchWithDaltons (boolean) : 予測フラグメントと観測フラグメントをダルトン単位で一致させるかどうか (デフォルトは false)
DaTolerance (float) : 実験ピークを探すために予測ピークの周囲に何ダルトンがあるか (デフォルト 0.05)
useTopFragments (boolean) : スペクトル予測を N 個の最高強度ピークにフィルタリングするかどうか (デフォルトは true)
topFragments (int) : 特徴計算に使用する予測フラグメントの最大数 (デフォルトは 20)。 useTopFragments が true の場合にのみ適用されます
removeRankPeaks (boolean) : デフォルトで true に設定され、一致した実験スペクトルからフラグメントが除外されます。 false の場合、実験フラグメントは同じスキャンからの複数の PSM と一致する可能性があります。
useBasePeak (boolean) : 強度の高いフラグメントのみを使用するために MS2 予測に下限を適用するかどうか (デフォルトは true)
percentBasePeak (float) : ベースピーク強度の数パーセントの強度を持つフラグメントが類似度計算に含まれるパーセント。 useBasePeak が true の場合にのみ適用されます (デフォルトは 1)。
loessEscoreCutoff (float) : RT/IM キャリブレーション用の PSM 収集時の最初のパスに使用される期待値カットオフ。デフォルトは 10^-3.5、つまり約 0.000316 です。
rtLoessRegressionSize (int) : RT LOESS キャリブレーションに使用される PSM の最大数 (デフォルトは 5000)
imLoessRegressionSize (int) : rtLoessRegressionSize と同じですが、IM 用 (デフォルトは 1000)
minLoessRegressionSize (int) : LOESS RT/IM キャリブレーションを試行するために必要な PSM の最小数 (デフォルトは 100)。使用可能な PSM の数がこの数よりも少ない場合は、代わりに線形回帰が使用されます
minLinearRegressionSize (int) : 線形回帰 RT/IM キャリブレーションを試行するために必要な PSM の最小数 (デフォルトは 10)。使用可能な PSM の数がこの数よりも少ない場合、キャリブレーションは試行されません
loessBandwidth (String) : RT/IM LOESS キャリブレーションで試行する帯域幅のリスト (デフォルトは 0.01、0.05、0.1、0.2)。これは、間にスペースを入れずにカンマで区切る必要があります
regressionSplits (int) : RT/IM LOESS キャリブレーションに使用される相互検証の数 (デフォルトは 5)
massesForLoessCalibration (String) : 独自の検量線に適合させる必要がある質量シフトの質量。リストはカンマで区切られ、間にスペースは入りません。質量は PIN ファイルと同じ桁数で書き込まれる必要があります。
loessScatterOpacity (float) : LOESS キャリブレーション数値の散布図の不透明度、0 ~ 1 (デフォルトは 0.35)
新機能を備えた .pin ファイル。デフォルトでは、「_edited.pin」で終わる新しいピン ファイルが生成されます。使用されるデフォルトの機能は、「unweighted_spectral_entropy」、「delta_RT_loess」、および「pred_RT_real_units」です。イオンモビリティ機能が有効になっている場合、「delta_IM_loess」と「ion_Mobility」も含まれます
spectraRT.tsv および spectraRT_full.tsv: DIA-NN 予測モデルの入力ファイル
spectraRT.predicted.bin: MSBooster が特徴計算に使用する DIA-NN からの予測を含むバイナリ ファイル。 FragPipe-PDV を使用する場合、これらのファイルは実験スペクトルと予測スペクトルのミラー プロットを生成するために使用されます。
MSBooster は、データとモデルの予測をどのように比較するかをさらに調べるために使用できる複数のグラフを作成します。
MSBooster_plots フォルダー:
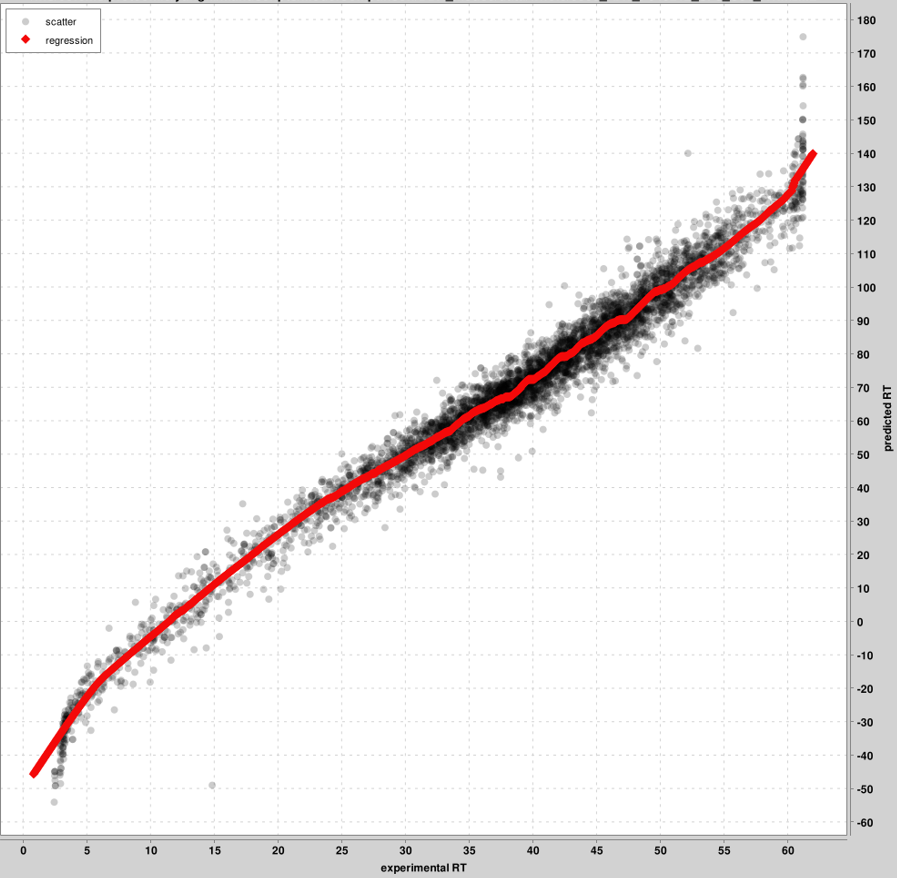
RT_calibration_curves: 実験的 RT スケールと予測 RT スケール間のキャリブレーションには、上位 5000 PSM までが使用されます。すべての PSM ではなく、これらの上位 PSM がグラフに表示されます。ピン ファイルごとに 1 つのグラフが生成されます
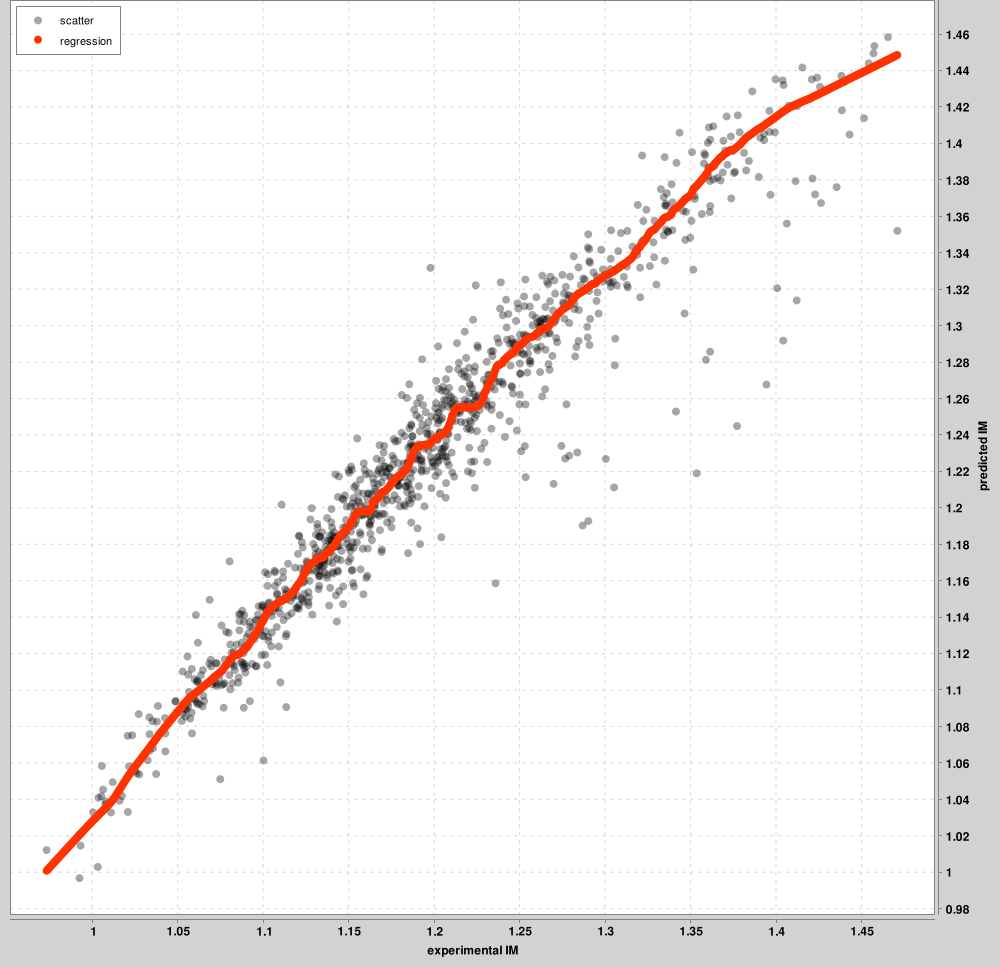
IM_calibration_curves: 実験的 IM スケールと予測された IM スケールの間のキャリブレーションに、上位 1000 PSM までが使用されます。すべての PSM ではなく、これらの上位 PSM がグラフに表示されます。充電状態ごとに個別の曲線が学習されます。以下の図は、電荷 2 プリカーサーの例です。
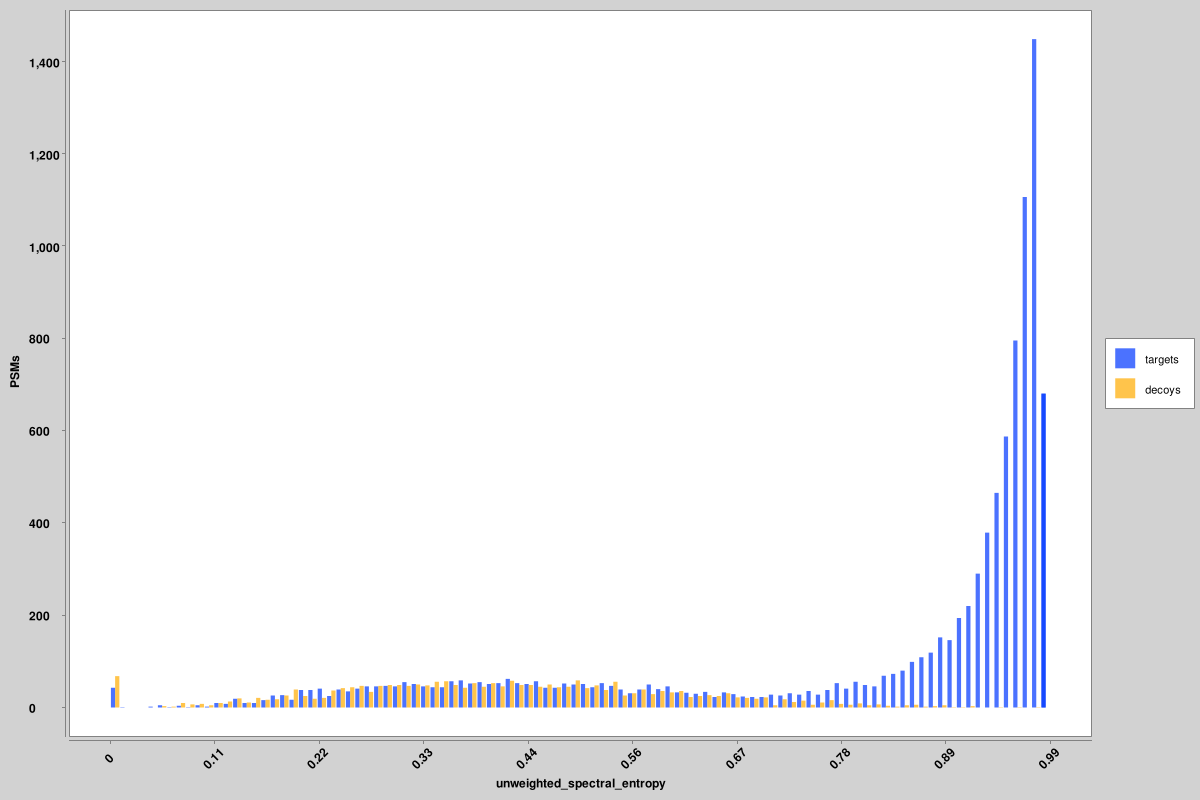
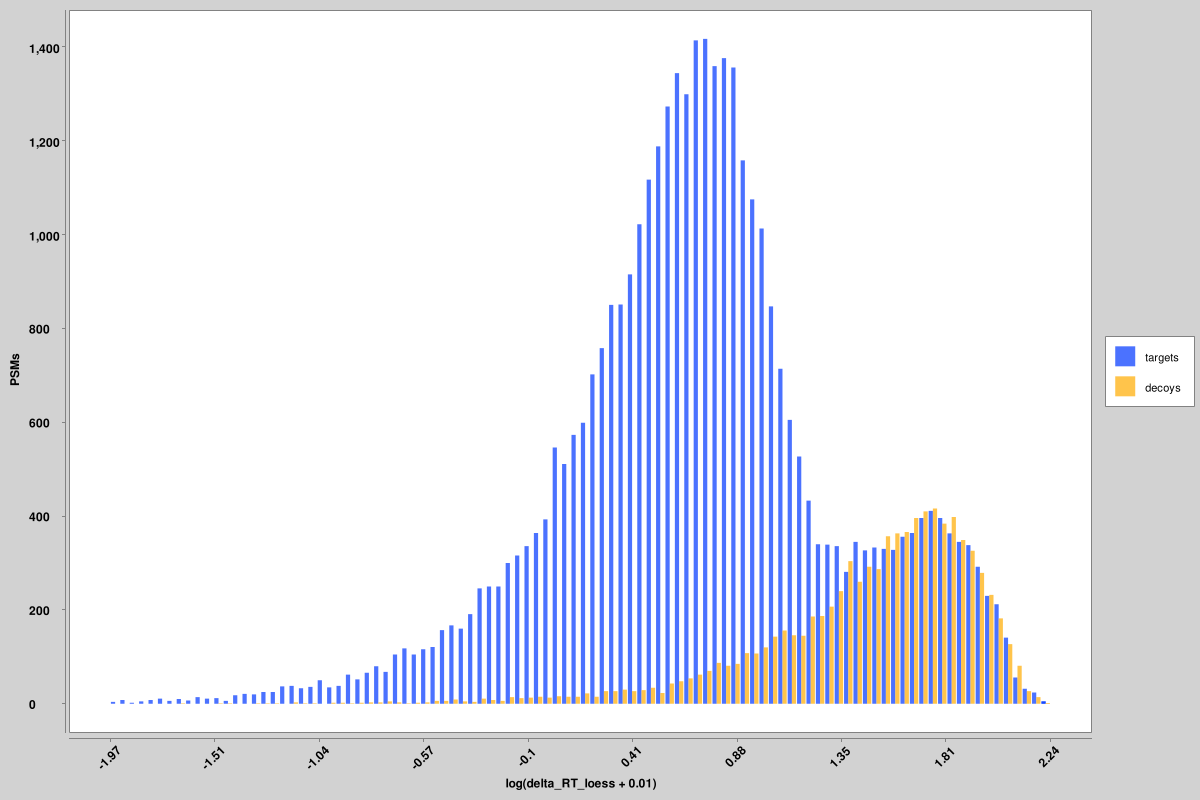
スコア_ヒストグラム: 各ピン ファイルのすべてのターゲットおよびデコイ PSM のオーバーレイ ヒストグラム。ここでは、真陽性と偽陽性の二峰性分布をよりわかりやすく視覚化するために、いくつかの特徴が対数スケールでプロットされていますが、ピン ファイルで使用されるのは元の値であり、対数スケール バージョンではありません。ここに示されているのは、重み付けされていないスペクトル エントロピーとデルタ RT スコアのヒストグラムですが、すべての特徴に対して同様のヒストグラムが生成されます。
MSBooster 特徴生成に Koina のペプチド予測モデルを使用する: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
MGF ファイル経由で任意のモデルから予測を読み取る
許可されているすべての機能と、それらをグラフィカル出力で QC する方法に関するドキュメント
MSBooster を使用する場合は、次を引用してください: https://www.nature.com/articles/s41467-023-40129-9