これは、「One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompts」の公式リポジトリです。
これは、前例のないデータ収集 (72 の公開 3D 医療セグメンテーション データセット) に基づいて構築された知識強化型のユニバーサル セグメンテーション モデルであり、テキスト (解剖学的データ) によって促され、3 つの異なるモダリティ (MR、CT、PET) と 8 つの人体の領域から 497 のクラスをセグメント化できます。用語)。
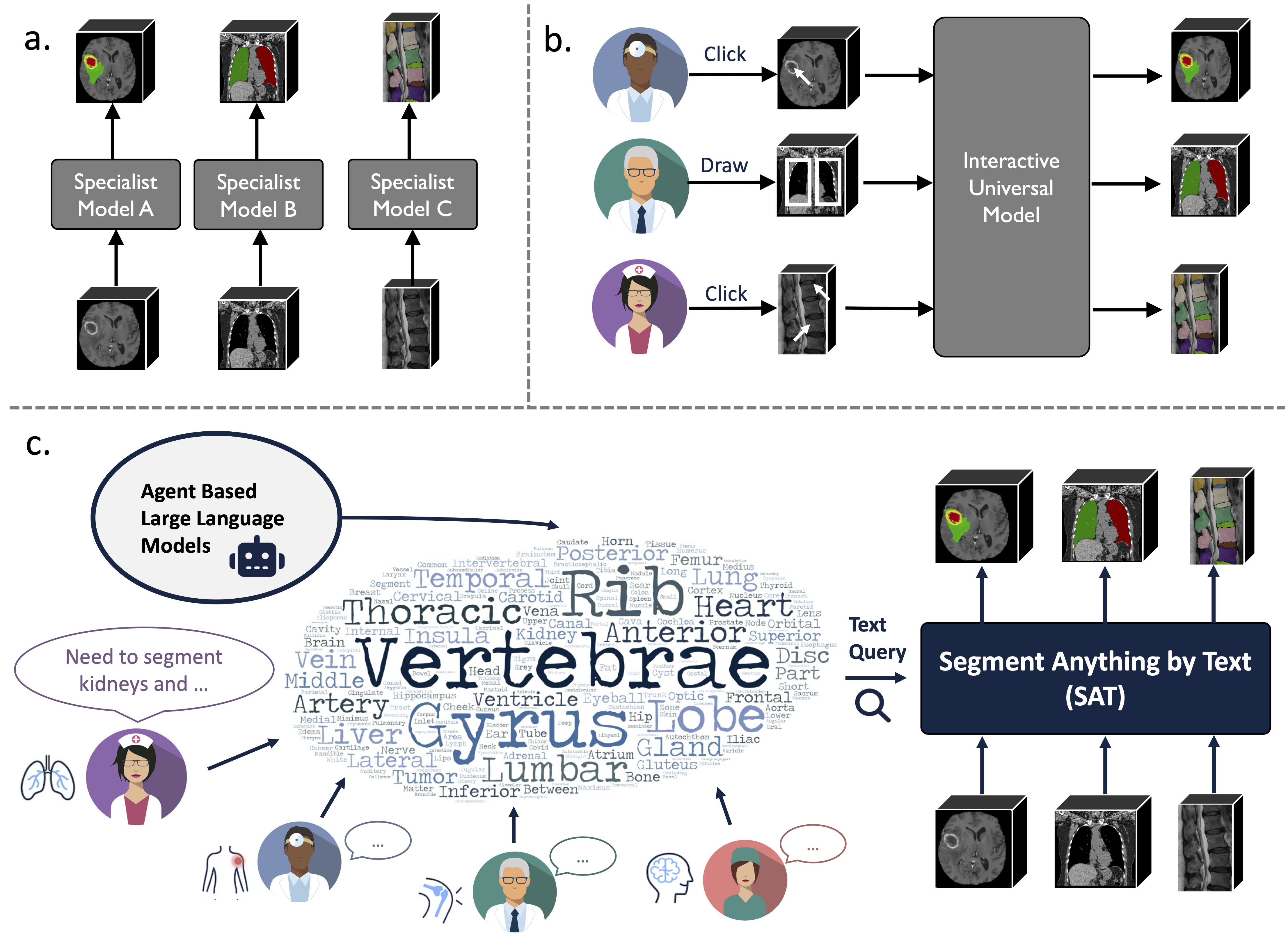
これは、一連の専門モデルをトレーニングしてデプロイするよりも強力で効率的です。詳細については、Web サイトまたは紙面をご覧ください。
2024.08 ? SAT および大規模言語モデルに基づいて、包括的で大規模な領域ガイド付き 3D 胸部 CT 読影データセットを構築します。これには、196 のカテゴリの臓器レベルのセグメンテーションと、各文が対応するセグメンテーションに基づいた多粒度レポートが含まれています。ハグフェイスで確認してください。
2024.06 ?私たちは、72 のパブリック セグメンテーション データセットのコレクションであるSAT-DSを構築するためのコードをリリースしました。これには、22,000 を超える 3D 画像、302,000 のセグメンテーション マスク、および 3 つの異なるモダリティ (MRI、CT、PET) と 8 つの人体の領域からの 497 のクラスが含まれています。私たちはSATを構築します。また、42/72 データセットのショートカット ダウンロード リンクも提供しています。これらは利便性を考慮して当社によって前処理およびパッケージ化されており、ダウンロードして抽出するとすぐに使用できるようになります。詳細については、このリポジトリを確認してください。
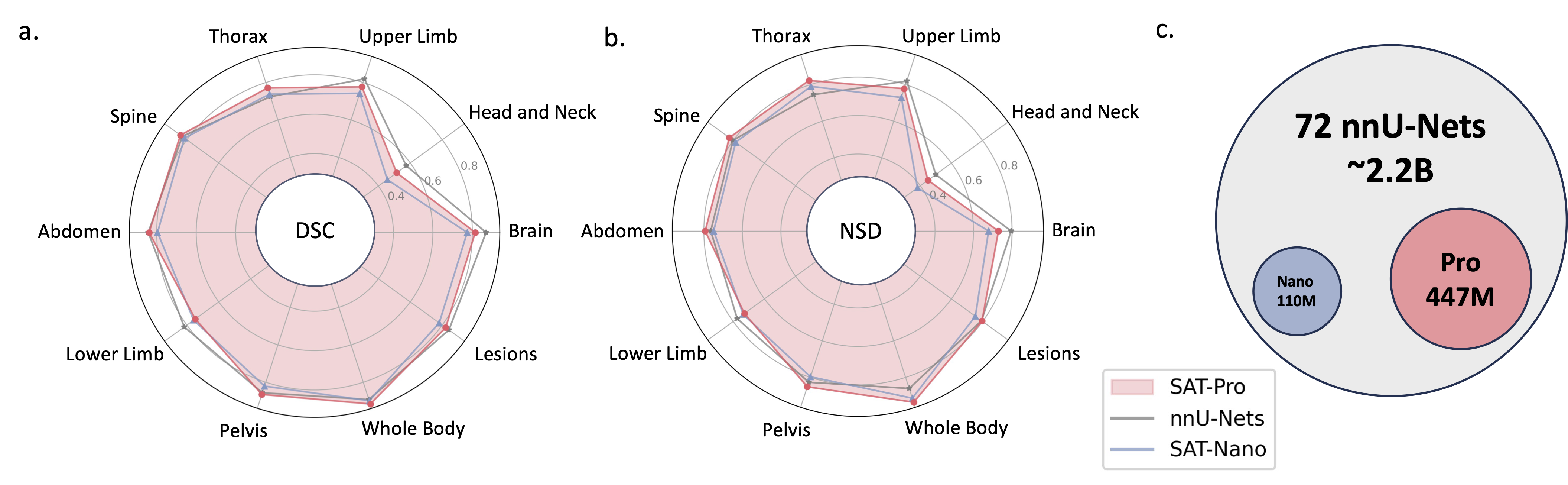
2024.05 ?私たちは、より大きなモデル サイズ ( SAT-Pro ) とより多くのデータセット ( 72 ) を使用して SAT の新しいバージョンをトレーニングし、現在497 のクラスをサポートしています。また、SAT-Nano をリニューアルし、さまざまなビジュアル バックボーン (U-Mamba および SwinUNETR) およびテキスト エンコーダー (MedCPT および BERT-Base) に基づいた SAT-Nano のいくつかのバリアントをリリースします。このアップデートの詳細については、新しい論文を参照してください。
U-Net の実装は、動的ネットワーク アーキテクチャのカスタマイズされたバージョンに依存してインストールされます。
cd model
pip install -e dynamic-network-architectures-main
その他の重要な要件は次のとおりです。
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
SAT-Nano の U-Mamba バリアントが必要な場合は、 mamba_ssmもインストールする必要があります。
S1. requirements.txtに従って環境を構築します。
S2. Huggingface から SAT と Text Encoder のチェックポイントをダウンロードします。
S3.データをjsonlファイルで準備します。 data/inference_demo/demo.jsonlのデモを確認してください。
セグメント化する各サンプルには、 image (画像へのパス)、 labe (セグメンテーション ターゲットの名前)、 dataset (サンプルが属するデータセット)、およびmodality (ct、mri、または pet) が必要です。 SAT がサポートするモダリティとクラスは、この論文の表 12 に記載されています。
orientation_code (方向) はデフォルトでRASであり、アキシャル面のほとんどの画像に適しています。矢状面の画像 (脊椎検査など) の場合は、これをASRに設定します。入力画像はH,W,D形状である必要があります。データ処理コードは、向き、強度、間隔などの観点から入力画像を正規化します。正常に処理された 2 つのイメージはdemoprocessed_dataにあります。SAT のパフォーマンスを保証するために正規化が正しく行われていることを確認してください。
S4. SAT-Pro で推論を開始しますか?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d入力画像パッチのバッチ サイズであり、GPU メモリに基づいて調整する必要があります (下の表を確認してください)。 --max_queries 、GPU メモリが非常に限られている場合を除き、推論データセット内のクラスよりも大きく設定することをお勧めします。
| モデル | バッチサイズ_3d | GPUメモリ |
|---|---|---|
| SAT-Pro | 1 | ~34GB |
| SAT-Pro | 2 | ~62GB |
| SAT-Nano | 1 | ~24GB |
| SAT-Nano | 2 | ~36GB |
S5. --rcd_dirの出力を確認してください。結果はデータセットごとに整理されます。それぞれのケースで、入力画像、集約されたセグメンテーション結果、および各クラスのセグメンテーションを含むフォルダーが見つかります。すべての出力は nifiti ファイルとして保存されます。 ITK-SNAP を使用してそれらを視覚化できます。
72 個のデータセットでトレーニングされた SAT-Nano を使用したい場合は、 --vision_backbone 「UNET」に変更し、それに応じて--checkpointと--text_encoder_checkpointを変更します。
他の SAT-Nano バリアント (49 データセットでトレーニング) の場合:
UNET-Ours: --vision_backbone 'UNET'と--text_encoder 'ours'を設定します。
UNET-CPT: --vision_backbone 'UNET'および--text_encoder 'medcpt'設定します。
UNET-BB: --vision_backbone 'UNET'と--text_encoder 'basebert'設定します。
UMamba-CPT: --vision_backbone 'UMamba'と--text_encoder 'medcpt'を設定します。
SwinUNETR-CPT: --vision_backbone 'SwinUNETR'および--text_encoder 'medcpt'を設定します。
トレーニングを開始する前の準備:
sh/の slurm スクリプトを使用してください。 SAT-Pro を例に挙げます。 sbatch sh/train_sat_pro.sh
これには、このリポジトリに従ってテスト データを構築する必要もあります。評価プロセスを開始するには、slurm スクリプトsh/evaluate_sat_pro.shを参照してください。
sbatch sh/evaluate_sat_pro.sh
このコードを研究やプロジェクトに使用する場合は、次を引用してください。
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}