空間トータル RNA シーケンス (STRS) のための一連の分子および計算ワークフロー
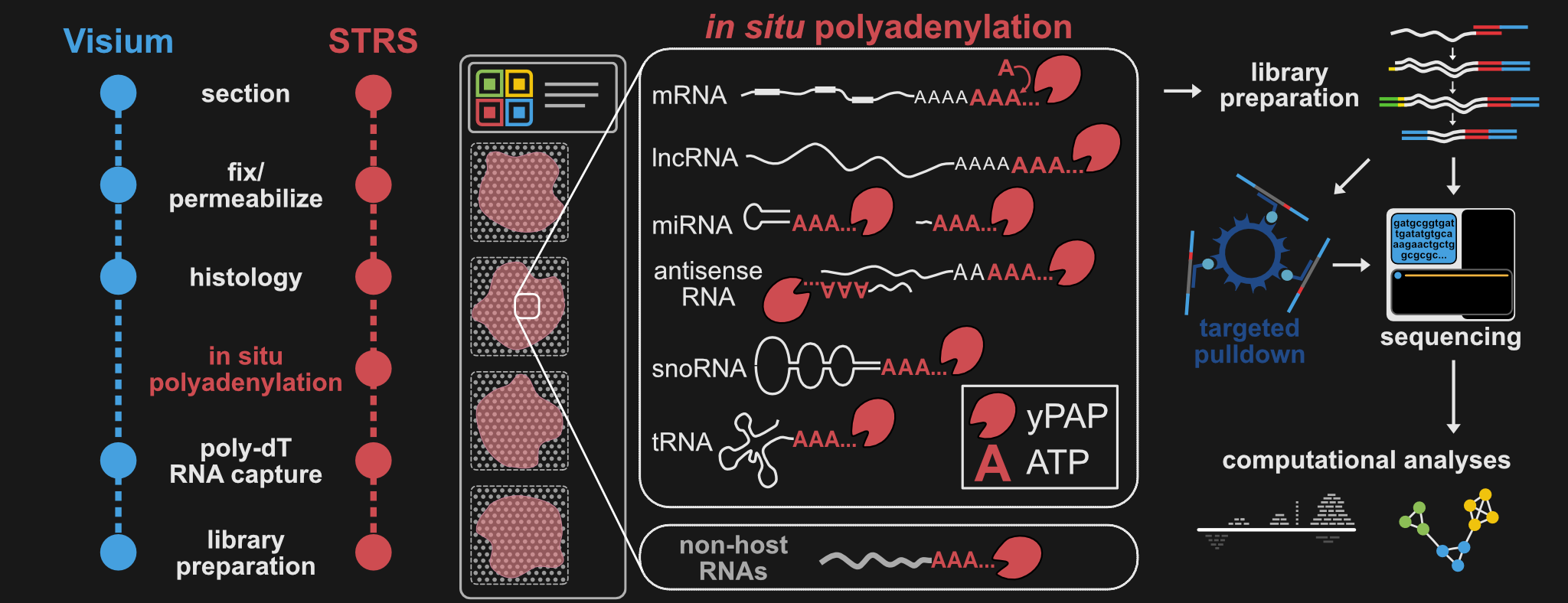
このリポジトリには、ほぼすべてのサンプルで STRS を実行および分析するために必要なすべてのプロトコル、パイプライン、およびスクリプトが含まれています。
txg_snakeチェックしてください。McKellar et al、 Nature Biotechnology 、2022
bioRxiv プレプリント リンク
manuscripts : プレプリントとその後の原稿の .pdf ファイル (できれば!)
protocols : ここで使用されるプロトコルの Microsoft Word および .pdf ドキュメント
pipelines : 研究で使用された Snakemake ワークフロー。これらには、STRS データ (kallisto、STARsolo、および miRge3.0)、小規模 RNAseq データ (STAR および miRge3.0)、SmartSeqTotal (kallisto)、および VASAdrop (kallisto) のアライメント パイプラインが含まれます。
scripts : 使用した他のすべてのコード!主に、空間解析で使用される R スクリプトとユーティリティ関数が含まれています。
references : この研究で使用された参照ゲノムおよび注釈に関する情報とスクリプト
resources : データの分析に使用した各種メタデータ、遺伝子リスト、その他の情報詳細については、各サブディレクトリの README ファイルを参照してください。
STRS データセット: GSE200481
SkM/心臓小型RNAseqデータ:GSE200480
単核トータル RNAseq データ (C2C12 細胞株): GSE209780
Small RNA Atlas データ (Isakova et al、 PNAS 、2020): GSE119661
SmartSeqTotal データ (Isakova et al、 PNAS 、2021): GSE151334
VASAdrop データ (Salmen et al、 Nature Biotechnology 、2022): GSE176588
イウィン・デ・ヴラミンク ([email protected])
ベンジャミン D. コスグローブ ([email protected])
デビッド W. マッケラー ([email protected])