このプロジェクトは、母集団をサンプリングするためのさまざまな方法と、さまざまな方法の評価で構成されています。私たちは、無反応者、偽陽性/陰性率、感染期間中の患者の感染プロファイルの能力など、サンプルに基づく感染レベルの推定にバイアスを引き起こす可能性のある多くの状況を含めています。このパッケージは、EpiABM モデルに基づいて、疾病伝播のシミュレーションを実行して各サンプリング方法の予測誤差を確認することにより、最適なサンプリング方法を出力することもできます。
EpiOS はまだ PyPI では利用できませんが、モジュールはローカルに pip インストールできます。このディレクトリは、まずローカル マシンにダウンロードする必要があり、その後、次のコマンドを使用してインストールできます。
pip install -e .また、感染シミュレーションのデータを生成するために EpiABM モデルをインストールすることをお勧めします。まず pyEpiabm をマシン上の任意の場所にダウンロードし、次のコマンドを使用してインストールできます。
pip install -e path/to/pyEpiabm ドキュメントには、上記のdocsバッジからアクセスできます。
私たちのプロジェクトの UML クラス図は次のとおりです。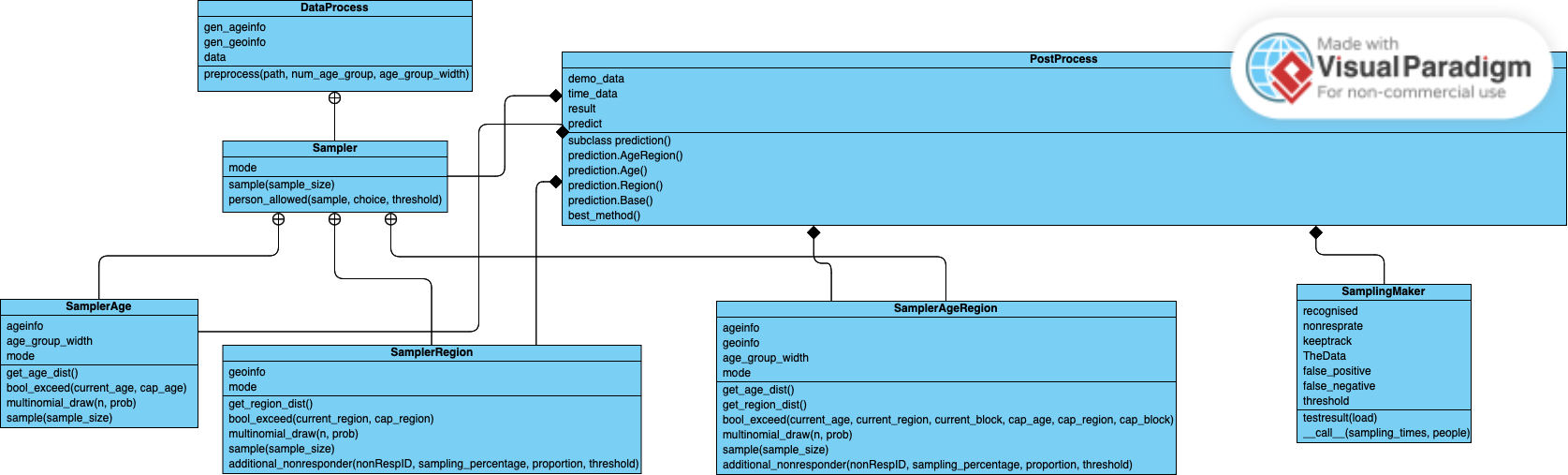
params.pyファイルには、このモデルに必要なすべてのパラメーターが含まれています。また、 inputフォルダー内のファイルは、データの前処理時に生成される一時ファイルの例です。これはサンプラー クラスによって使用されます。各サンプラー クラスのdata_store_pathパラメーターは、これらのファイルを保存するパスです。
PostProcess使用してプロットを生成するまず、新しいPostProcessオブジェクトを定義し、pyEpiabm から生成された人口統計データdemodataと感染データtimedataを入力する必要があります。次に、PostProcess.predict を使用して、さまざまなサンプリング方法に基づいて予測を実行できます。使用するサンプリング メソッドをメソッドとして直接呼び出すことができます。次に、サンプリングする時点とサンプル サイズを指定します。ここでは、サンプリング方法としてAgeRegion使用し、サンプリングされる時点として[0, 1, 2, 3, 4, 5] 、サンプル サイズとして3使用します。最後に、パラメータnon_responderおよびcomparison指定することで、非応答者を考慮するかどうか、および結果を真のデータと比較するかどうかを指定できます。
コード例は次のとおりです。
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)これで、フィギュアが指定されたパスに保存されました。
PostProcess使用して最適なサンプリング方法を選択するまず、新しいPostProcessオブジェクトを定義し、pyEpiabm から生成された人口統計データdemodataと感染データtimedataを入力する必要があります。次に、PostProcess.best_method を使用して、さまざまなサンプリング方法のパフォーマンスを比較できます。比較したいメソッドを指定できます。次に、サンプリングするサンプリング間隔とサンプル サイズを指定します。 3 番目に、パラメータnon_responderおよびcomparison指定することで、非応答者を考慮するかどうか、および結果を真のデータと比較するかどうかを指定できます。さらに、サンプリング方法は確率的であるため、平均的なパフォーマンスを得るために実行される反復回数を指定できます。さらに、 parallel_computationオンにして高速化することもできます。最後に、 hyperparameter_autotuneオンにして、ハイパーパラメータの最適な組み合わせを自動的に見つけることができます。
コード例は次のとおりです。
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed