
このリポジトリは、「CELL-E 2: 双方向テキストから画像への変換器を使用してタンパク質を画像に変換し、元に戻す」の公式実装です。
仮想環境を作成し、次の方法で必要なパッケージをインストールします。
pip install -r requirements.txt
次に、適切な CUDA バージョンでtorch = 2.0.0をインストールします。
モデルはHugging Faceで入手できます。
独自のデータで予測を実行できる 2 つのスペースもご利用いただけます。
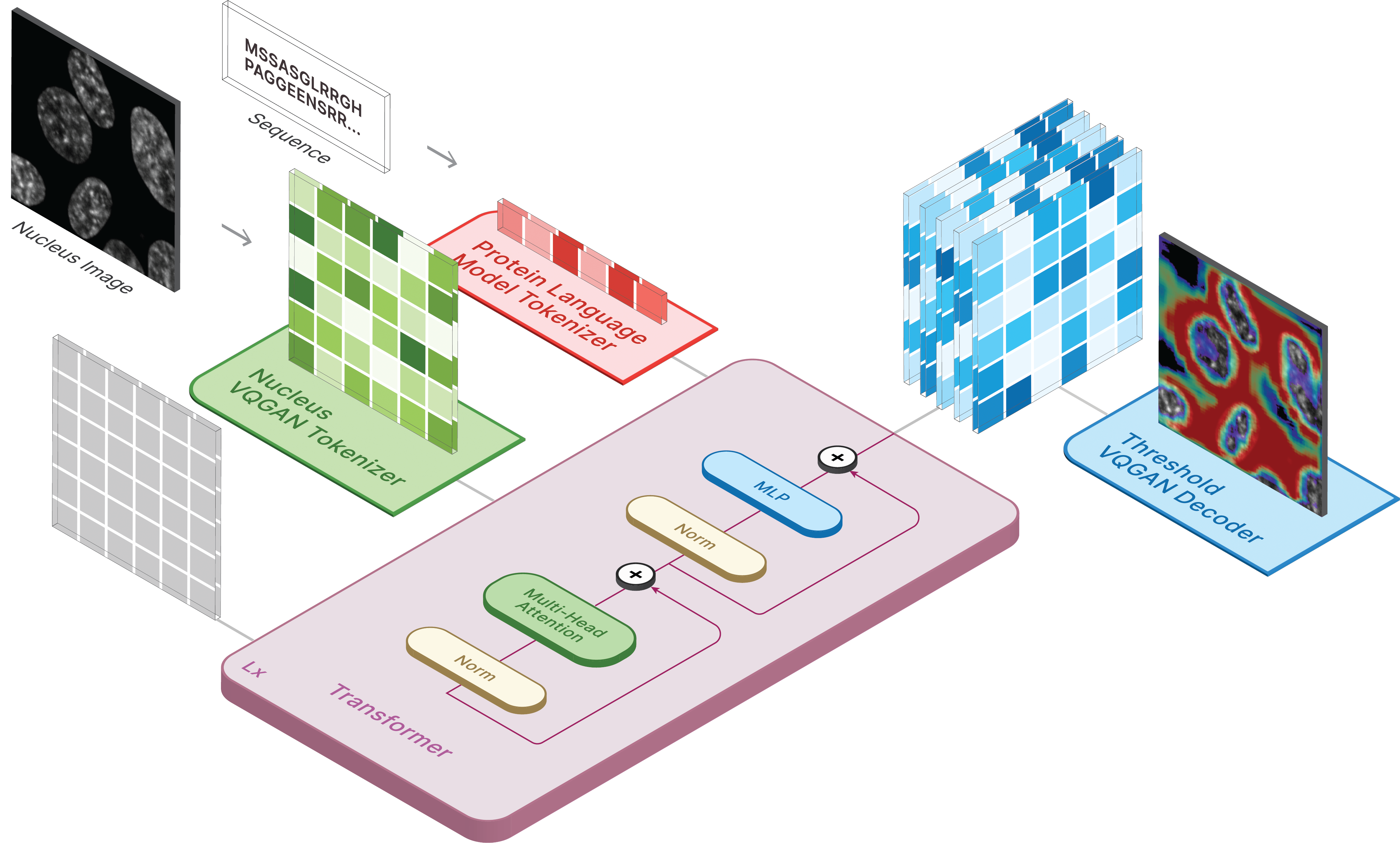
画像を生成するには、保存されたモデルを ckpt_path として設定します。この方法は不安定になる可能性があるため、別の読み込み方法についてはDemo.ipynbを参照してください。
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
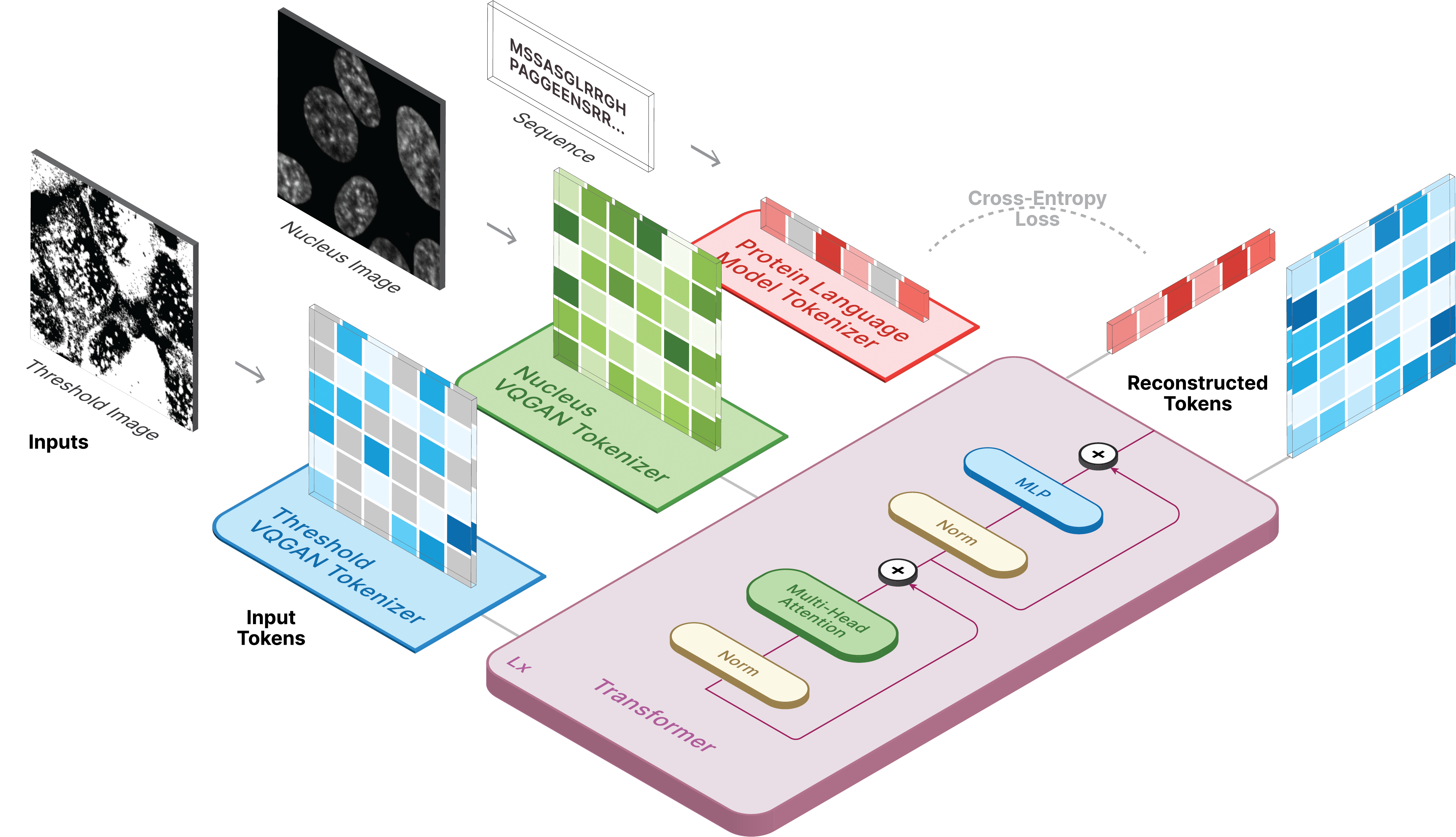
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )CELL-E のトレーニングは 3 つの段階で行われます。
タンパク質しきい値画像を使用する場合は、データセットのthreshold: True設定します。
私たちは、taming-transformers コードのわずかに変更されたバージョンを使用します。
トレーニングするには、次のスクリプトを実行します。
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
--gpusなどの追加のフラグについては、元のリポジトリを参照してください。
Human Protein Atlas および OpenCell イメージをダウンロードするためのスクリプトをスクリプト フォルダーに提供します。データローダーにはdata_csvが必要です。 nucleus_image_path 、 protein_image_path 、 metadata_path 、 split (train または val)、およびsequence (オプション) の列を含む csv ファイルを生成する必要があります。このファイルは、画像ファイルやメタデータ ファイルと同じ一般dataフォルダー内に存在すると想定されます。
メタデータは JSON であり、すべてのタンパク質シーケンスに付随する必要があります。シーケンスがdata_csvに表示されない場合は、 protein_sequenceという名前のキーとともにmetadata.jsonに表示される必要があります。
ここにさらに情報を追加すると、個々のタンパク質をクエリするのに役立ちます。これらは、データセット オブジェクト内にself.metadata変数を作成するretrieve_metadata介して取得できます。
トレーニングするには、次のスクリプトを実行します。
python celle_main.py --base configs/celle.yaml -t True
--gpus VQGAN と同じ形式で指定します。
CELL-E には次のオプションが含まれています。
ckpt_path : 以前の CELL-E 2 トレーニングを再開します。 state_dict を使用して保存されたモデルvqgan_model_path : タンパク質画像エンコーダー用の保存されたタンパク質画像モデル (state_dict 付き)vqgan_config_path : 保存されたタンパク質イメージ モデル yamlcondition_model_path : タンパク質画像エンコーダー用の保存された条件 (核) モデル (state_dict 付き)condition_config_path : 保存された条件 (核) モデル yamlnum_images : タンパク質画像エンコーダのみを使用する場合は 1、条件画像エンコーダを含む場合は 2image_key : nucleus 、 target 、またはthresholddim : 言語モデルの埋め込みの次元num_text_tokens : 言語モデル内のトークンの総数 (ESM-2 の場合は 33)text_seq_len : 考慮されるアミノ酸の総数depth : トランスフォーマー モデルの深さ。通常、VRAM を犠牲にして深いほど優れています。heads : マルチヘッド アテンションで使用されるヘッドの数dim_head : アテンションヘッドのサイズattn_dropout : トレーニングにおける注意ドロップアウト率ff_dropout : トレーニングにおけるフィードフォワードドロップアウト率loss_img_weight : 画像再構成に適用される重み付け。テキストの重み = 1loss_text_weight : 画像再構成の条件に適用される重み付け。stable : 規範重み (爆発的な勾配が発生する場合用)learning_rate : Adam オプティマイザーの学習率monitor : モデルを保存するために使用されるパラメータ研究の一部に私たちのコードを使用することに決めた場合は、引用してください。
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}