深層学習アプリケーション用の GPU を利用した、エピジェネティック トラック データと対応するシーケンスを含む BigWig ファイルの高速バッチ データロード。
Bigwig-loader は主に、rapidsai kvikio ライブラリと cupy に依存しており、どちらも conda/mamba を使用してインストールするのが最適です。 Bigwig-loader は conda/mamba を使用してインストールできるようになりました。 bigwig-loader がインストールされた新しい環境を作成するには:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderまたは、これをenvironment.ymlファイルに追加します。
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loaderそして更新します:
mamba env update -f environment.ymlBigwig-loader は、rapidsai kvikio ライブラリと cupy がすでにインストールされている環境で pip を使用してインストールすることもできます。
pip install bigwig-loaderBigWigDataset を、直接使用できる PyTorch 反復可能データセットにラップしました。
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step ()フレームワークに依存しない Dataset オブジェクトは、 bigwig_loader.datasetからインポートできます。このデータセット オブジェクトは、キューピー テンソルを返します。 Cupy テンソルは cuda 配列インターフェイスに準拠しており、JAX または tensorflow テンソルにゼロコピー変換できます。
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)その他の例については、サンプル ディレクトリを参照してください。
このライブラリは、同じ次元のデータのバッチをロードすることを目的としており、ロード プロセスを高速化できるいくつかの前提を考慮しています。以下のプロットからわかるように、少量のデータを読み込む場合、pyBigWig は非常に高速ですが、機械学習のためのデータ読み込みのバッチ処理の性質を利用していません。
以下のベンチマークでは、GPU ごとに複数の CPU が使用される現実的なシナリオと比較するために、pyBigWig を使用して PyTorch データローダー (set_start_method('spawn') を使用) も作成しました。 CPU データローダーのスループットは CPU の数に比例して増加しないことがわかります。そのため、学習ステップ中に GPU を維持し、ニューラル ネットワークをトレーニングして飽和状態に保つために必要なスループットを得ることが困難になります。
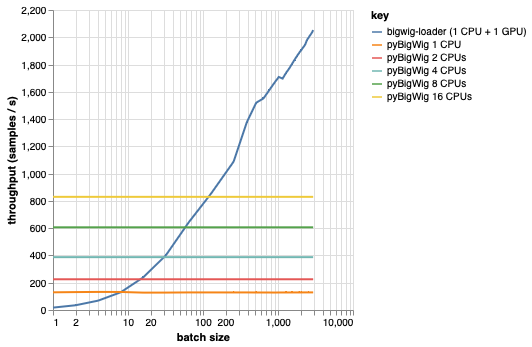
これは、bigwig-loader が解決する問題です。これは、bigwig-loader の使用方法の例です。
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlこの環境では、 pytest -v実行して、テストが成功することを確認できるはずです。注: bigwig-loader を使用するには GPU が必要です。
このセクションでは、新しい機能を追加するために必要な手順について説明します。不明な点がある場合は、問題を開いてください。
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install実行して、pre-commit フックをインストールします。テストはtestsディレクトリにあります。最も重要なテストの 1 つは test_against_pybigwig で、pyBigWIg に間違いがある場合に、それが bigwig-loader にもあることを確認します。
pytest -vv .GPU を備えた Github ランナーが利用可能になったら、これらのテストを CI で実行したいと考えています。ただし今のところ、ローカルで実行できます。
このライブラリを使用する場合は、以下を引用することを検討してください。
Retel、Joren Sebastian、Andreas Poehlmann、Josh Chiou、Andreas Steffen、Djork-Arné Clever です。 「BigWig ファイルからのエピジェネティック トラック用の高速機械学習データローダー」バイオインフォマティクス 40、いいえ。 1 (2024 年 1 月 1 日): btad767。 https://doi.org/10.1093/bioinformatics/btad767。
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}