グアー ( Cyamopsis tetragonoloba (L.) Taub.) のジェノタイピングと GWAS プロジェクトのデータとコードを含むリポジトリ。リポジトリの内容の簡単な説明:
make_consensus_vcf.pyスクリプトは、生の GATK-HC、NGSEP、TASSEL 5 バリアント コールを使用して最終バリアント データセットを生成するために使用され、 make_snp_stats.pyスクリプトは同じ入力とともにバリアント コールの品質統計を調査するために使用されました。
callset_refinement_filtering.ipynbノートブックは、バリアントの品質管理を実行し、一般的なバリアントの最終的なフィルター処理されたデータセットを構築するために使用されました。
genetic_analysis.ipynbノートブックは、PCA、連鎖不平衡分析、一般化線形モデルベースの関連分析など、ほとんどの遺伝子分析を実行するために使用されました。
prepare_farmcpu.pyスクリプトは、FarmCPU ツールの入力データをフォーマットするために使用されました (結果の p 値の QQ プロットは論文と以下に示されています)。
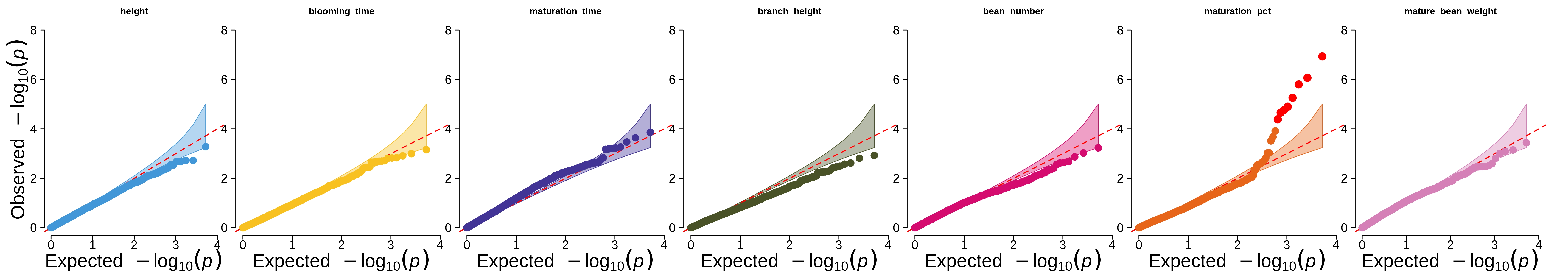
admix_file_create.pyファイルは、ADMIXTURE 分析用の遺伝子型を前処理するために使用されました (この一部は、 genetic_analysis.ipynbの Hail を使用して行われました)。
guar_stat.Rスクリプトは、最終データの統計分析を実行し、FarmCPU ツールを使用してゲノム全体の関連性分析を実行し、主要な数値をプロットするために使用されました。
parse_vcf_fcpu.pyスクリプトは、このリポジトリでgenotypes.xlsxファイルとして利用できる最終的な SNP 遺伝子型テーブルをフォーマットするために使用されました。