
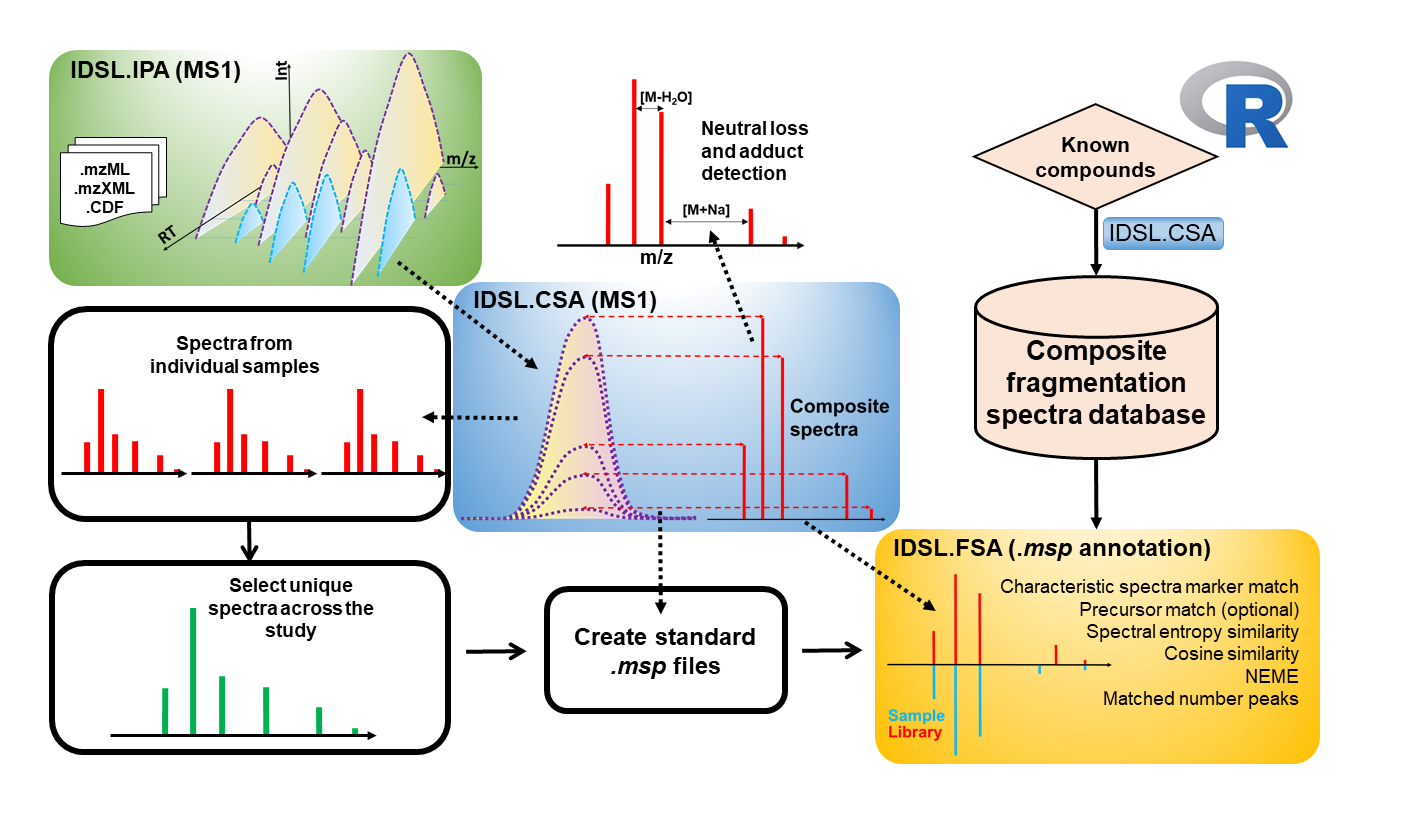
質量分析データの分析のための複合スペクトル分析(IDSL.CSA) Rパッケージは、メタボロミクスおよびエキスポソームのための統合データサイエンス研究所(IDSL.ME)によって開発されました。このパッケージは、MS1のみの複合スペクトルデコンボリューション分析( CSA )、データに依存する取得( DDA )、およびMS Eを含むさまざまなデータに依存しない取得( DIA )メソッドなどのさまざまな分析方法を通じて得られた断片化スペクトルのデコンボリューションに使用できます。 、全イオン断片化(AIF)、およびSwath-MS分析。 IDSL.CSAパッケージの目的は、データ分析プロセスの合理化と、メタボロミクスとエキスポソームの分野での化学構造注釈全体の改善を支援することです。
StartタブでPARAM0006を試してください。 install.packages("IDSL.CSA")
IDSL.CSAワークフローを使用して質量分析データ( MZXML 、 MZML 、 NetCDF )を処理する前に、IDSL.ipaワークフローを使用して質量分析データを処理して、ピークのクロマトグラフィー情報( M/Z-RT )を取得する必要があります。 IDSL.ipaワークフローを使用して、個々および集約されたアライメントされたピークリストのクロマトグラフィー情報が生成された場合、IDSL.CSAパラメータースプレッドシートをダウンロードして、それに応じてパラメーターを選択してから、このスプレッドシートをIDSL.CSAワークフローの入力として使用します:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
これらの手順に従って、Hilic-esi-Pos/negモードで収集されたThermo q Exactive HF Hybrid Orbitrapデータを備えたクイックケーススタディ(n = 33)ST002263を使用してください。
idsl.ipaについて説明した方法を使用して、生の質量分析データとクロマトグラフィー情報を処理します
複合スペクトル分析では、フルスケール分析のために5つの別々のセクションに分散された39のパラメーターが必要です。この調査では、IDSL.CSAパラメータースプレッドシートに表示されるデフォルトのパラメーター値を使用します。次に、情報を提供します
2.1。 StartタブでPARAM0001の[はい]を選択して、 CSAワークフローのみを処理します。
2.2。 HRMSデータの場所アドレスのCSA0005 (MS1レベルHRMSデータ)
2.3。 idsl.ipaワークフローによって生成されたpeaklistsディレクトリのアドレスのCSA0008
2.4。 idsl.ipaワークフローによって生成されたpeak_alignmentディレクトリのアドレスのCSA0009
2.5。出力場所のCSA0011 (.MSPファイルとEIC)
2.6。また、計算能力に応じてCSA0004を使用して処理スレッドの数を増やすこともできます
このコマンドをr/rstudioコンソールまたはターミナルで実行します。
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
CSA0011に提供されたアドレスで結果を解析できます。
4.1。 CSA_MSPには.mspファイルが含まれています
4.2。 CSA_ADDUCT_ANNOTATIONには、潜在的な付加情報を持つピークリストが含まれています
4.3。 peak_alignment_subsetには、各CSAクラスターのメジャーイオン用のアライメントピークテーブルのサブセットが含まれています
4.4。 aligned_spectra_tableには、整列したテーブル上のCSA集約に関する情報が含まれています
[1] Fakouri Baygi、S.、Kumar、Y。Barupal、dk idsl.csa:標的化されていないメタボロミクスデータセットの化学注釈のための複合スペクトル分析。 Analytical Chemistry 、 2023、95 (25) 、9480–9487。
[2] Fakouri Baygi、S.、Kumar、Y。Barupal、DK Idsl。 IPAは、ターゲットを絞っていないLC/HRMSデータセットの有機化学空間を特徴付けます。 Journal of Proteome Research 、 2022、21 (6) 、1485-1494。