
Islingは、ペアリングエンド読み取りでウイルスまたはベクトルの統合を検出するためのツールです。詳細については、私たちの論文を読んでください。
condaとsnakemakeがインストールされている場合は、(含まれている)テストデータをローカルに実行します。
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
snakemakeとsingularityがインストールされている場合は、代わりに使用できます。
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
または、Dockerがインストールされている場合は、MacOSに実行できます。
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
これにより、コンテナ内の構成ファイルとデータが使用され、結果は現在の作業ディレクトリに呼び出さoutたフォルダーに表示されます。 Linuxでは、このコマンドをルートとして実行する必要があり、WindowsでBind -Mount Syntax( -v引数)を調整する必要があります。
入力データ(読み取りとホストおよびウイルス参照)は、configファイルを指定します - 独自のデータについては、独自のデータを指すためにconfigファイルのサンプル( test/config/test.yml )を変更する必要があります。 configfile.md参照して、configファイルの形式の詳細については。
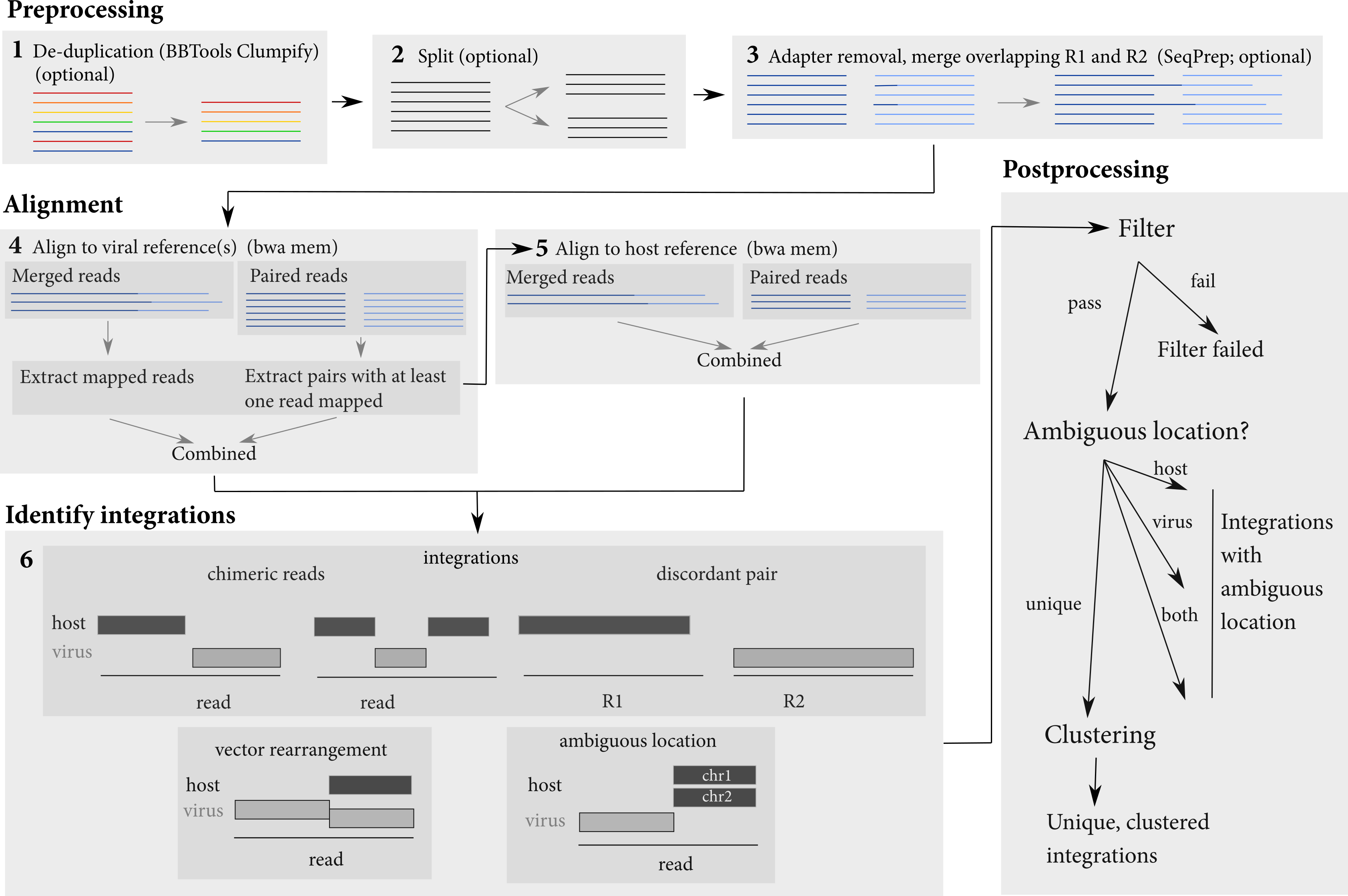
パイプラインは、統合サイトを識別するためにいくつかのステップを実行します。 FASTQファイルまたはBAMファイルのいずれかで構成される入力データセットとしてかかります。読み取りの前処理(オーバーラップの読み取り、オプションのマージ)を行い、ホストとウイルスシーケンスの両方に整列させます。読み取りは最初にウイルスシーケンスに整列され、次に整列された読み取りが抽出され、ホストに整列されます。これらのアラインメントは、ウイルス統合を識別するために使用されます。
Islingには、依存関係を提供するために、 snakemakeとsingularity (推奨)またはcondaいずれかが必要です。追加、 pythonバージョン3.5以上、およびpandasが必要です( condaにsnakemakeインストールする場合は、これらを自動的にインストールする必要があります。
Alternativley、Islingとすべての依存関係を含むDockerバージョンを使用します。
必要な入力は、ホストとウイルス/ベクトルの参照を指定する構成ファイルであり、読み取りが必要です。構成ファイルのすべての入力を指定します。 Islingは現在、ペアエンドリードでのみ機能しています。
この構成ファイルの形式の説明については、ファイルconfigfile.md参照してください。
ISLING OUTPUT統合サイトは、構成ファイルで指定された出力ディレクトリのタブで区切られた形式の統合サイトを使用します。
出力フォルダー内で、構成ファイルの各データセットに1つのフォルダーが作成され、各データセットについては、統合がintsディレクトリにあります。各サンプルに出力ファイルのセットが1つあります。
各サンプルについて、多くの出力ファイルがありますが、これは特定のユースケースにとって興味深い場合があります。
<sample>.<host>.<virus>.integrations.txt :フィルターに合格したかどうかに関係なく、検出されたすべてのジャンクションに関する情報<sample>.<host>.<virus>.integrations.post.txt :すべてのフィルターに合格した検出されたジャンクションに関する情報<sample>.<host>.<virus>.integrations.post.unique.txt :すべてのフィルターを通過し、ホストとベクター/ウイルスの両方に明確な位置を持つ検出された接合部に関する情報<sample>.<host>.<virus>.integrations.post.unique.merged.txt :宿主とウイルス/ベクターの両方で、同じ座標とのオーバーラップジャンクションまたは接合部のマージの結果。マージは、ホストとウイルス/ベクターの両方に明確な位置を持つ統合ジャンクションのみで行われます。<sample>.<host>.<virus>.integrations.post.host_ambig.txt :すべてのフィルターを通過し、明確な位置ベクトル/ウイルスを持っているがホストの曖昧な位置を持つ検出されたジャンクションに関する情報<sample>.<host>.<virus>.integrations.post.virus_ambig.txt :すべてのフィルターを通過し、明確な位置ホストを持っているが、ベクター/ウイルスの曖昧な場所を持つ検出された接合部に関する情報<sample>.<host>.<virus>.integrations.post.both_ambig.txt :すべてのフィルターを通過し、ホストとベクトル/ウイルスの両方に曖昧な位置を持つ検出された接合部に関する情報出力ファイルは、識別された統合の位置とそのプロパティを提供します。積分ジャンクションの座標は、曖昧な基盤の観点から指定されています。つまり、多くの場合、宿主と読み取りのウイルス部分の間にはギャップまたは重複があります。 
この場合、統合の位置を一意に決定することはできないため、Islingは、統合の位置として、ホストとベクター/ウイルスゲノムのこれらの塩基の座標を出力します。
マージされたクラスター出力ファイルを除き、すべてのファイルにはこれらの列が含まれています。
gapまたはoverlapであるため、あいまいなベースが発生した場合、曖昧な塩基がない場合は接合部がcleanです。あるいは、不一致のペアでjucntionが識別された場合、 OverlapTypeはdiscordantですhv )またはウイルス/宿主( vh )のいずれか+または-bwaから)編集bwaから)編集OverlapTypeがgapの場合の曖昧なベースの数chimeric読み取りで観察されるジャンクション、 discordantペアであるか、同じ読み取りで両方の接合部が観察されたshort統合ですかbwaから)bwaから)'xxx'によって分離されますIsling原稿の数字を再現するには、 benchmarkingディレクトリのreadmeを参照してください。