Última atualização: 30/09/2024
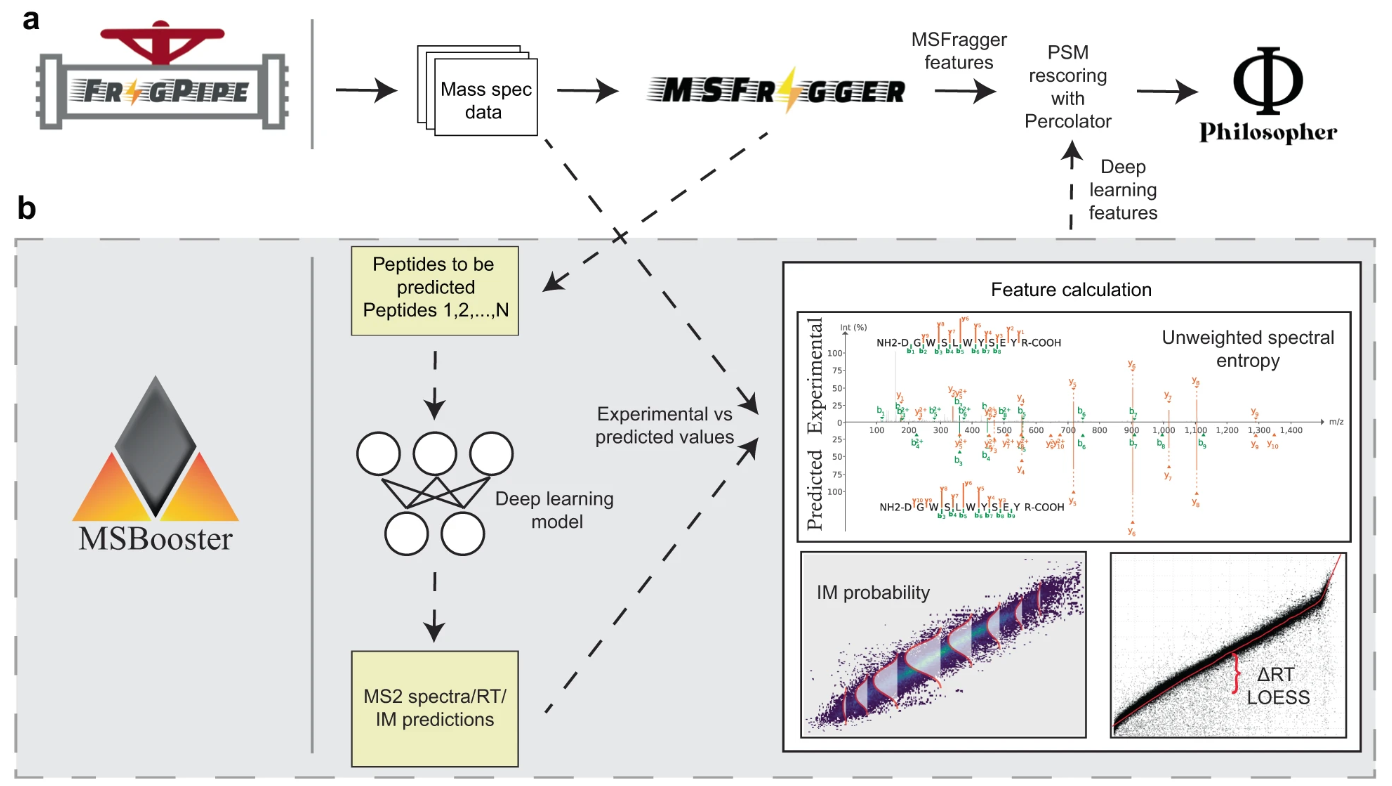
MSBooster é uma ferramenta para incorporar previsões de biblioteca espectral na recuperação de correspondência de espectro de peptídeo (PSM) em dados proteômicos de espectrometria de massa de cromatografia líquida em tandem ascendente. É basicamente dividido em 4 etapas:
Extração de peptídeos de PSMs em resultados de pesquisa e formatação para arquivos de entrada de preditores de aprendizado de máquina/aprendizado profundo (ML/DL)
Chamando o(s) modelo(s) de previsão e salvando a saída
Cálculo de recursos
Adição de novos recursos ao arquivo de resultados de pesquisa
O MSBooster é compatível com muitos tipos de pesquisas em bancos de dados, incluindo imunopeptidomia HLA, DDA e DIA e proteômica de célula única. Ele está incorporado ao FragPipe e está incluído em muitos de seus fluxos de trabalho. O MSBooster foi desenvolvido pensando em outras ferramentas FragPipe, como o FragPipe-PDV.
O MSBooster está equipado para lidar com vários formatos e modelos de arquivos de entrada:
| Saída do espectrômetro de massa |
|---|
| .mzML |
| .mgf |
| arquivo PSM |
|---|
| .alfinete |
| .pepXML (em andamento) |
| Modelo de previsão |
|---|
| DIA-NN |
| Modelos Koina |
MSBooster pode ser executado em sistemas Windows e Linux. Se estiver usando o FragPipe, nenhuma outra etapa de instalação será necessária além de instalar o FragPipe. O MSBooster está localizado na aba "Validação". Escolha ativar recursos de tempo de retenção com "Predict RT" e recursos espectrais MS/MS com "Predict spectra". Consulte a documentação do FragPipe para saber como executar uma análise.
Se estiver usando o MSBooster independente para executar na linha de comando, baixe o arquivo jar mais recente em Releases. MSBooster também requer DIA-NN para previsão de MS/MS e RT. Instale o DIA-NN e anote o caminho para o executável do DIA-NN (ex. DiaNN.exe para Windows, diann-1.8.1.8 para Linux).
Você pode executar o MSBooster usando um comando semelhante ao seguinte:
java -jar MSBooster-1.2.1.jar --paramsList msbooster_params.txt
Os parâmetros mínimos que precisam ser passados são:
- DiaNN (String): path to DIA-NN executable (if using DIA-NN model, which is the MSBooster default) - mzmlDirectory (String): path to mzML/mgf files. Accepts multiple space-separated folder and files - pinPepXMLDirectory (String): path to pin files. Accepts multiple space-separated folder and files. If using in FragPipe, place the pin and pepXML files in the same folder
Embora você possa passar esses parâmetros individualmente, é mais fácil colocar um em cada linha do arquivo paramsList. Consulte msbooster_params.txt para obter um modelo.
Os parâmetros abaixo são para uso geral. Os parâmetros específicos do Koina estão na documentação do Koina
paramsList (String) : localização do arquivo de texto contendo parâmetros para esta execução
fragger (String) : caminho do arquivo fragger.params da execução do MSFragger. O MSBooster lerá vários parâmetros e ajustará os parâmetros internos com base neles, como tolerância a erros de massa de fragmentos e compensações de massa
outputDirectory (String) : onde gerar os novos arquivos
editedPin (String) : O MSBooster nomeará o novo arquivo com base nos fornecidos. Por exemplo, A.pin terá uma contraparte chamada A_edited.pin. Para alterar o padrão "editado", forneça uma nova string aqui
renamePin (int) : se deseja gerar um novo arquivo PIN ou reescrever o antigo. O padrão aqui é 1, que não será sobrescrito. Definir como 0 substituirá o arquivo PIN antigo
deletePreds (boolean) : se os arquivos que armazenam previsões do modelo devem ser excluídos após a conclusão de uma execução bem-sucedida. Por padrão, defina como falso. Defina como verdadeiro se desejar excluir estes
loadingPercent (int) : com que frequência relatar o progresso das tarefas usando um relator de progresso. Por padrão, definido como 10, o que significa que uma atualização será impressa a cada 10%.
numThreads (int) : número de threads a serem usados. Por padrão, definido como 0, que usa todos os threads disponíveis menos 1
splitPredInputFile (int) : usado apenas quando as previsões DIA-NN falham devido a um erro de falta de memória (137). Por padrão, defina como 1, mas você pode aumentar esse valor para especificar em quantos arquivos menores o arquivo de entrada DIA-NN deve ser dividido. Cada arquivo será então previsto sequencialmente, aliviando a carga de memória
plotExtension (String) : em qual formato de arquivo as plotagens devem estar. png por padrão, e pdf também é permitido
features (String) : lista de recursos a serem calculados. Diferencia maiúsculas de minúsculas, separado por comunicação, sem espaços entre elas. O padrão é "predRTrealUnits,unweightedSpectralEntropy,deltaRTLOESS"
spectraPredFile (String) : se você estiver reutilizando previsões espectrais antigas (por exemplo, de DIA-NN ou Koina), poderá especificar o local do arquivo aqui
RTPredFile (String) : igual a spectraPredFile, mas para previsões RT
IMPredFile (String) : igual a spectraPredFile, mas para previsões de IM
spectraModel (String) : qual modelo de previsão espectral usar
rtModel (String) : igual a spectraModel, mas para RT
imModel (String) : igual a spectraModel, mas para IM
useSpectra (boolean) : se deve usar recursos baseados em previsão espectral. Definido como verdadeiro por padrão
useRT (boolean) : se deve usar recursos baseados em previsão de RT. Definido como verdadeiro por padrão
useIM (boolean) : se deve usar recursos baseados em previsão de IM. Definido como falso por padrão
ppmTolerance (float) : erro de fragmento tolerância ppm (padrão 20ppm)
matchWithDaltons (boolean) : se deve corresponder fragmentos previstos e observados em Daltons (padrão falso)
DaTolerance (float) : quantos daltons em torno do pico previsto para procurar o pico experimental (padrão 0,05)
useTopFragments (boolean) : se deve filtrar a previsão espectral para os N picos de intensidade mais alta (padrão verdadeiro)
topFragments (int) : até quantos fragmentos previstos devem ser usados para cálculo de recursos (padrão 20). Aplicado apenas se useTopFragments for verdadeiro
removeRankPeaks (boolean) : definido como verdadeiro por padrão, que filtra fragmentos dos espectros experimentais, uma vez correspondidos. Se for falso, os fragmentos experimentais podem ser correspondidos por vários PSMs da mesma varredura
useBasePeak (boolean) : se um limite inferior deve ser aplicado às previsões MS2 para usar apenas fragmentos com intensidade mais alta (padrão verdadeiro)
percentBasePeak (float) : porcentagem na qual o fragmento com intensidade de alguma porcentagem da intensidade do pico base é incluído no cálculo de similaridade. Aplicado apenas se useBasePeak for verdadeiro (padrão 1)
loessEscoreCutoff (float) : corte do valor esperado usado para a primeira passagem na coleta de PSMs para calibração RT/IM. O padrão é 10 ^ -3,5 ou aproximadamente 0,000316
rtLoessRegressionSize (int) : número máximo de PSMs usados para calibração RT LOESS (padrão 5000)
imLoessRegressionSize (int) : igual a rtLoessRegressionSize, mas para IM (padrão 1000)
minLoessRegressionSize (int) : número mínimo de PSMs necessários para tentar a calibração LOESS RT/IM (padrão 100). Se menos do que esse número de PSMs estiverem disponíveis, a regressão linear será usada
minLinearRegressionSize (int) : número mínimo de PSMs necessários para tentar a calibração de RT/IM de regressão linear (padrão 10). Se menos do que este número de PSMs estiverem disponíveis, nenhuma calibração será tentada
loessBandwidth (String) : lista de larguras de banda a serem testadas para calibração RT/IM LOESS (padrão 0,01,0,05,0,1,0,2). Deve ser separado por vírgulas, sem espaços entre eles
regressionSplits (int) : número de validações cruzadas usadas para calibração RT/IM LOESS (padrão 5)
massesForLoessCalibration (String) : massas para deslocamentos de massa que devem ser ajustadas às suas próprias curvas de calibração. A lista é separada por vírgulas, sem espaços entre elas. As massas devem ser escritas com o mesmo número de dígitos do arquivo PIN
loessScatterOpacity (float) : opacidade dos gráficos de dispersão nos números de calibração LOESS, de 0 a 1 (padrão 0,35)
Arquivo .pin com novos recursos. Por padrão, novos arquivos pin serão produzidos terminando em "_edited.pin". Os recursos padrão usados são "unweighted_spectral_entropy", "delta_RT_loess" e "pred_RT_real_units". Se os recursos de mobilidade iônica estiverem habilitados, "delta_IM_loess" e "ion_mobility" também serão incluídos
spectraRT.tsv e spectraRT_full.tsv: arquivos de entrada para o modelo de previsão DIA-NN
spectraRT.predicted.bin: um arquivo binário com previsões do DIA-NN para ser usado pelo MSBooster para cálculo de recursos. Se estiver usando FragPipe-PDV, esses arquivos são usados para gerar gráficos espelhados de espectros experimentais e previstos
O MSBooster produz vários gráficos que podem ser usados para examinar melhor como seus dados se comparam às previsões do modelo.
Pasta MSBooster_plots:
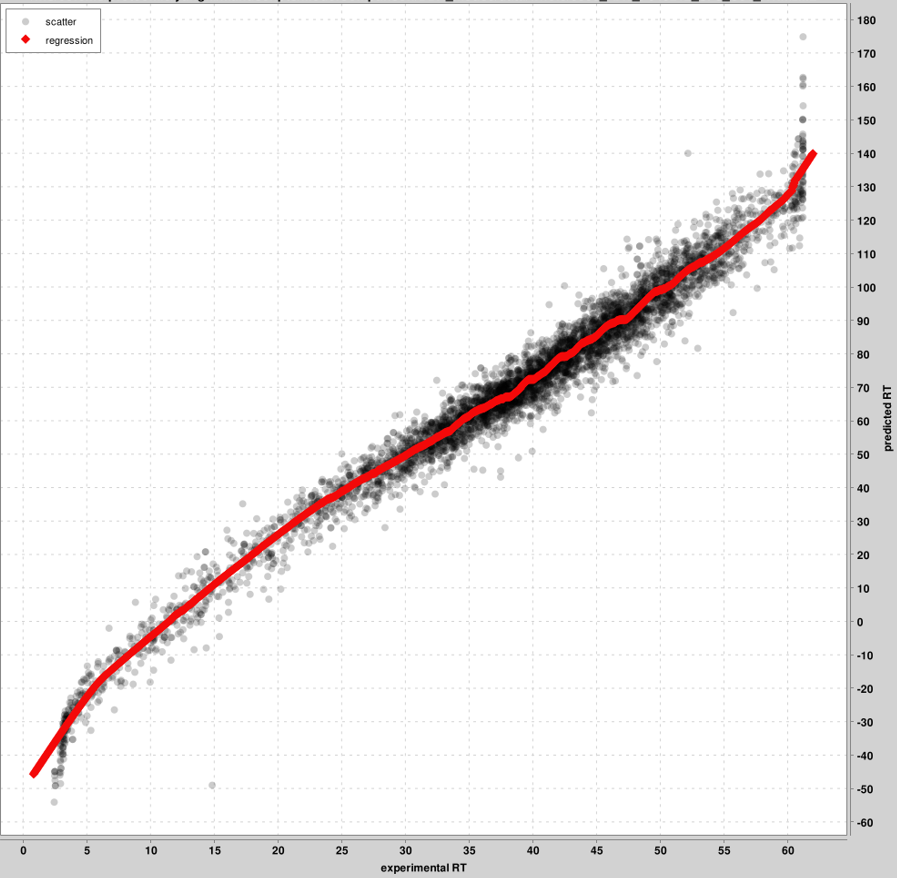
RT_calibration_curves: até os 5.000 principais PSMs serão usados para calibração entre as escalas de RT experimental e prevista. Esses principais PSMs são apresentados no gráfico, nem todos os PSMs. Um gráfico será produzido por arquivo pin
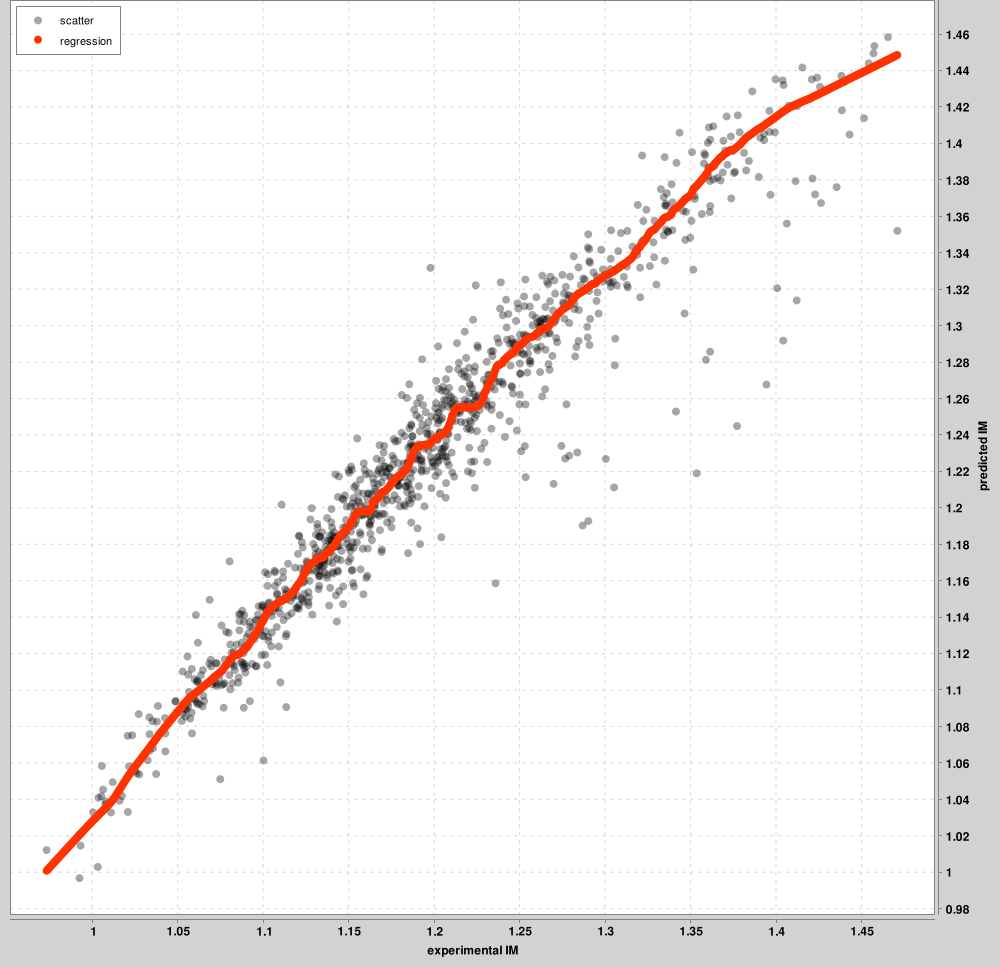
IM_calibration_curves: até os 1000 principais PSMs serão usados para calibração entre as escalas IM experimentais e previstas. Esses principais PSMs são apresentados no gráfico, nem todos os PSMs. Uma curva separada será aprendida para cada estado de carga. A figura abaixo é um exemplo de precursores de carga 2
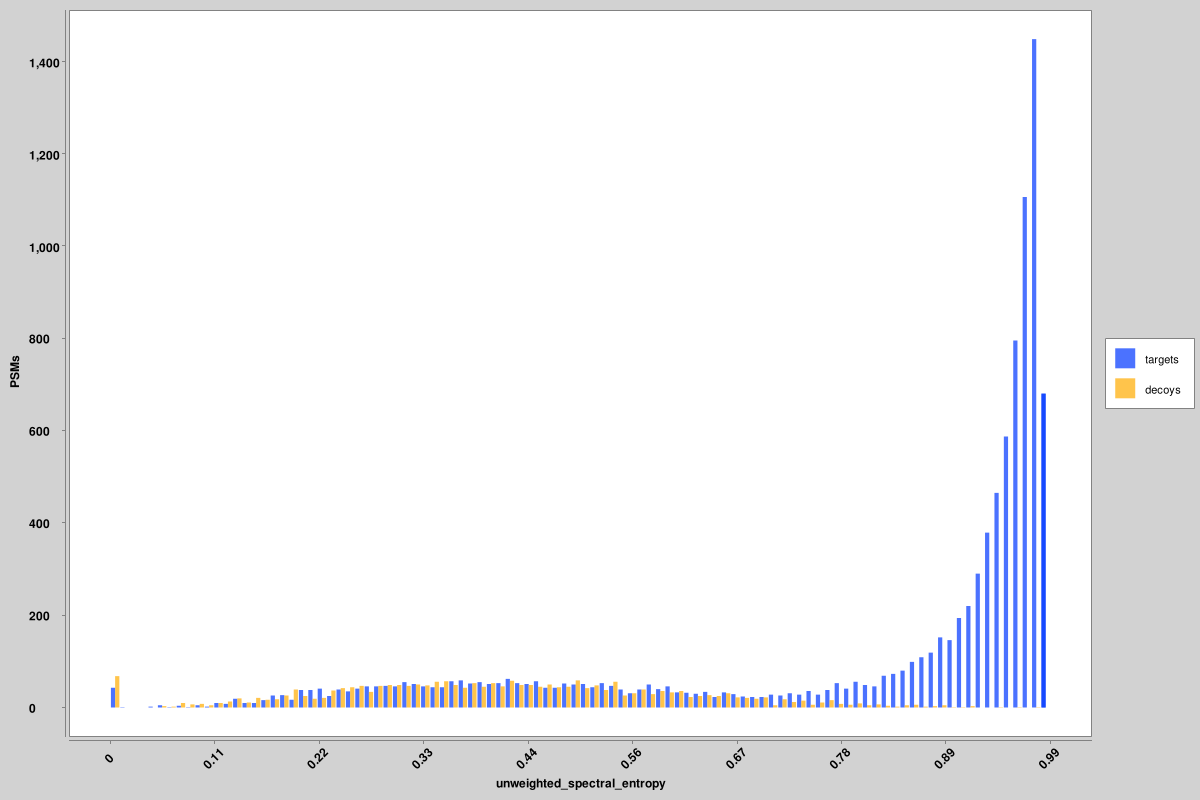
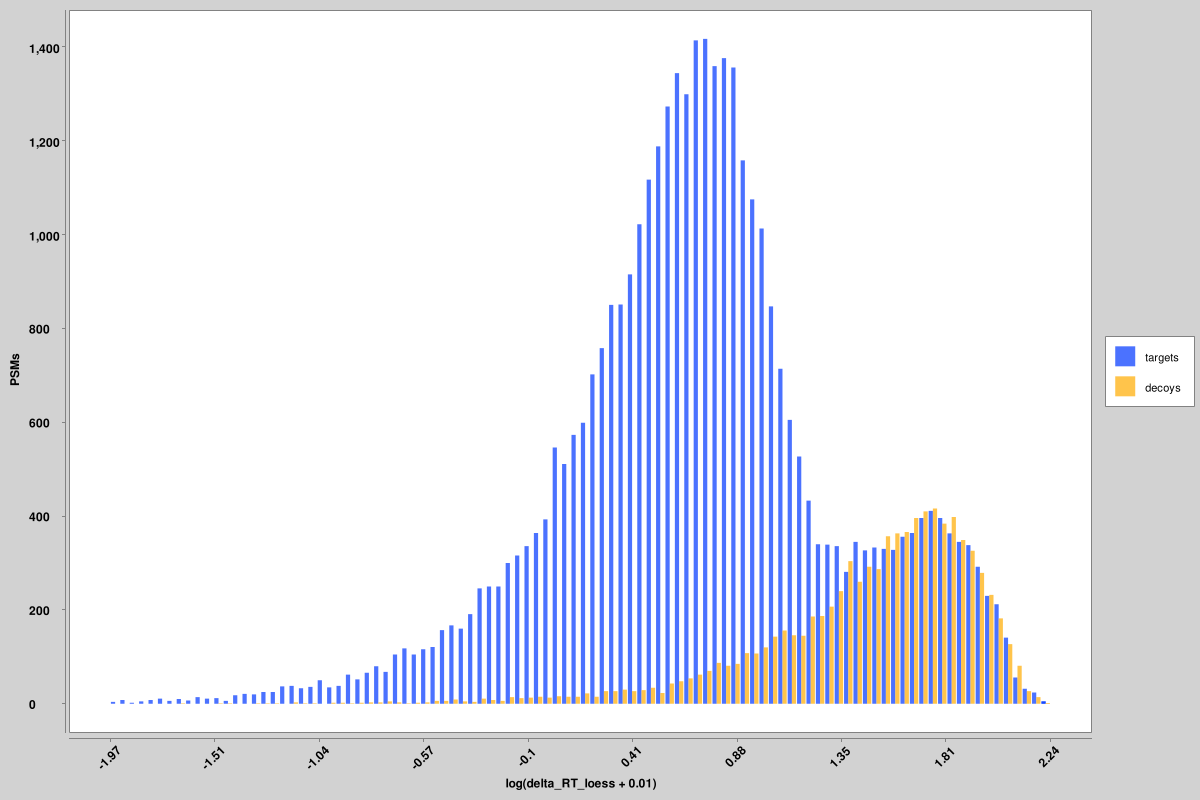
score_histograms: histogramas sobrepostos de todos os PSMs alvo e chamariz para cada arquivo pin. Alguns recursos são plotados aqui em uma escala logarítmica para melhor visualização da distribuição bimodal de verdadeiros e falsos positivos, mas o valor original é o que é usado nos arquivos PIN, não a versão em escala logarítmica. Aqui são mostrados histogramas para a entropia espectral não ponderada e pontuações delta RT, mas outros semelhantes são produzidos para todos os recursos
Use modelos de previsão de peptídeos do Koina para geração de recursos do MSBooster: https://fragpipe.nesvilab.org/docs/tutorial_koina.html
Lendo previsões de qualquer modelo por meio de arquivos MGF
Documentação sobre todos os recursos permitidos e como controlá-los com saída gráfica
Por favor, cite o seguinte ao usar o MSBooster: https://www.nature.com/articles/s41467-023-40129-9