Este é o repositório oficial de "Um modelo para governar todos: Rumo à segmentação universal para imagens médicas com prompts de texto"
É um modelo de segmentação universal baseado em conhecimento, construído sobre uma coleta de dados sem precedentes (72 conjuntos de dados públicos de segmentação médica 3D), que pode segmentar 497 classes de 3 modalidades diferentes (RM, CT, PET) e 8 regiões do corpo humano, orientadas por texto (anatômica). terminologia).
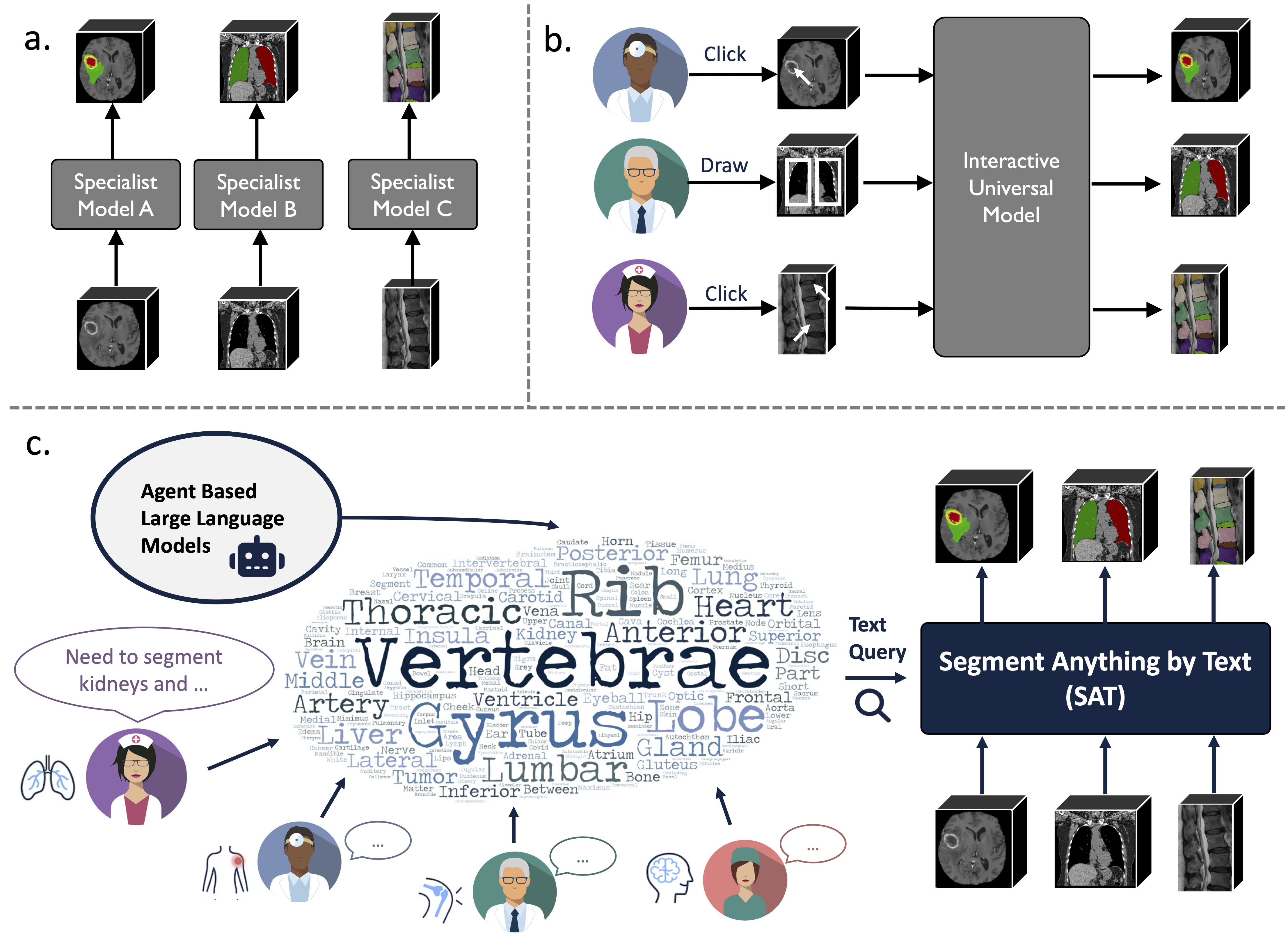
Pode ser poderoso e mais eficiente do que treinar e implementar uma série de modelos especializados. Encontre mais em nosso site ou jornal.
2024.08? Com base no SAT e em modelos de linguagem grande, construímos um conjunto de dados de interpretação de tomografia computadorizada de tórax 3D abrangente, em grande escala e guiado por região. Ele contém segmentação em nível de órgão para 196 categorias e relatórios multigranularidade, onde cada frase é fundamentada na segmentação correspondente. Confira no huggingface.
2024.06? Lançamos o código para construir o SAT-DS , uma coleção de 72 conjuntos de dados de segmentação pública, contendo mais de 22 mil imagens 3D, 302 mil máscaras de segmentação e 497 classes de 3 modalidades diferentes (ressonância magnética, tomografia computadorizada, PET) e 8 regiões do corpo humano, sobre as quais nós construímos o SAT. Também oferecemos links de download de atalhos para conjuntos de dados 42/72, que são pré-processados e empacotados por nós para sua conveniência, prontos para uso imediato após download e extração. Verifique este repositório para obter detalhes.
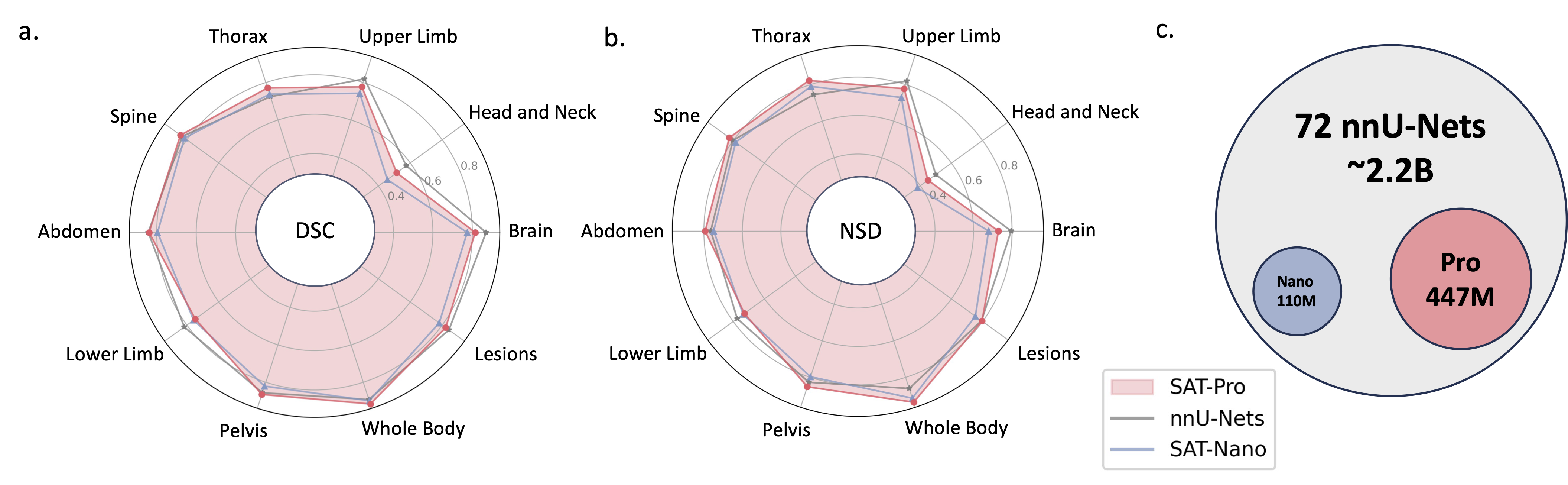
2024.05? Treinamos uma nova versão do SAT com tamanho de modelo maior ( SAT-Pro ) e mais conjuntos de dados ( 72 ), e agora ela suporta 497 classes! Também renovamos o SAT-Nano e lançamos algumas variantes do SAT-Nano, baseadas em diferentes backbones visuais (U-Mamba e SwinUNETR) e codificadores de texto (MedCPT e BERT-Base). Para obter mais detalhes sobre esta atualização, consulte nosso novo artigo.
A implementação do U-Net depende de uma versão customizada de arquiteturas de rede dinâmicas, para instalá-lo:
cd model
pip install -e dynamic-network-architectures-main
Alguns outros requisitos importantes:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
Você também precisa instalar mamba_ssm se quiser a variante U-Mamba do SAT-Nano
S1. Construa o ambiente seguindo requirements.txt .
S2. Baixe o ponto de verificação do SAT e do codificador de texto do huggingface.
S3. Prepare os dados em um arquivo jsonl. Verifique a demonstração em data/inference_demo/demo.jsonl .
image (caminho para a imagem), labe (nome dos alvos de segmentação), dataset (a qual conjunto de dados a amostra pertence) e modality (ct, mri ou pet) são necessários para cada amostra segmentar. As modalidades e classes suportadas pelo SAT podem ser encontradas na Tabela 12 do artigo.
orientation_code (orientação) é RAS por padrão, o que se adapta à maioria das imagens no plano axial. Para imagens no plano sagital (por exemplo, exame da coluna vertebral), defina como ASR . A imagem de entrada deve ter formato H,W,D Nosso código de processo de dados normalizará a imagem de entrada em termos de orientação, intensidade, espaçamento e assim por diante. Duas imagens processadas com sucesso podem ser encontradas em demoprocessed_data , certifique-se de que a normalização seja feita corretamente para garantir o desempenho do SAT.
S4. Inicie a inferência com SAT-Pro?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d é o tamanho do lote dos patches da imagem de entrada e precisa ser ajustado com base na memória GPU (verifique a tabela abaixo); Recomenda-se que --max_queries seja maior que as classes no conjunto de dados de inferência, a menos que a memória da sua GPU seja muito limitada;
| Modelo | tamanho do lote_3d | Memória GPU |
|---|---|---|
| SAT-Pro | 1 | ~ 34 GB |
| SAT-Pro | 2 | ~ 62 GB |
| SAT-Nano | 1 | ~ 24 GB |
| SAT-Nano | 2 | ~ 36 GB |
S5. Verifique --rcd_dir para saídas. Os resultados são organizados por conjuntos de dados. Para cada caso serão encontrados a imagem de entrada, o resultado agregado da segmentação e uma pasta contendo as segmentações de cada classe. Todas as saídas são armazenadas como arquivos nifiti. Você pode visualizá-los usando o ITK-SNAP.
Se você quiser usar o SAT-Nano treinado em 72 conjuntos de dados, basta modificar --vision_backbone para 'UNET' e alterar --checkpoint e --text_encoder_checkpoint de acordo.
Para outras variantes do SAT-Nano (treinadas em 49 conjuntos de dados):
UNET-Nosso: set --vision_backbone 'UNET' e --text_encoder 'ours' ;
UNET-CPT: defina --vision_backbone 'UNET' e --text_encoder 'medcpt' ;
UNET-BB: definir --vision_backbone 'UNET' e --text_encoder 'basebert' ;
UMamba-CPT: definir --vision_backbone 'UMamba' e --text_encoder 'medcpt' ;
SwinUNETR-CPT: defina --vision_backbone 'SwinUNETR' e --text_encoder 'medcpt' ;
Alguma preparação antes de iniciar o treinamento:
sh/ para iniciar o processo de treinamento. Veja o SAT-Pro, por exemplo: sbatch sh/train_sat_pro.sh
Isso também requer a construção de dados de teste seguindo este repositório. Você pode consultar o script slurm sh/evaluate_sat_pro.sh para iniciar o processo de avaliação:
sbatch sh/evaluate_sat_pro.sh
Se você usar este código para sua pesquisa ou projeto, cite:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}