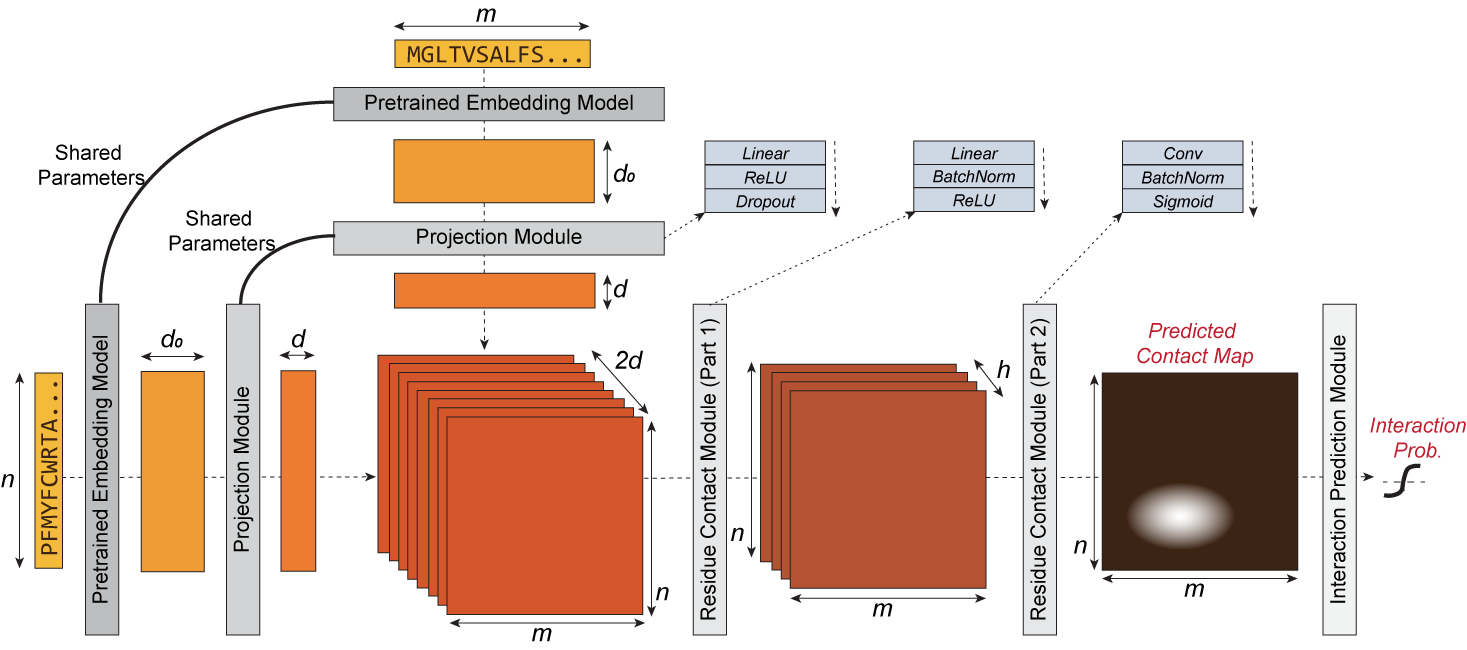
D-SCRIPT é um método de aprendizagem profunda para prever uma interação física entre duas proteínas, dadas apenas suas sequências. Generaliza bem para novas espécies e é robusto às limitações no tamanho dos dados de treinamento. Seu design reflete a intuição de que para duas proteínas interagirem fisicamente, um subconjunto de aminoácidos de cada proteína deve estar em contato um com o outro. Os estágios intermediários do D-SCRIPT implementam diretamente essa intuição, sendo o penúltimo estágio do D-SCRIPT uma estimativa aproximada do mapa de contato interproteico do dímero proteico. Este design motivado estruturalmente aumenta a interpretabilidade dos resultados e, uma vez que a estrutura é mais conservada evolutivamente do que a sequência, melhora a generalização entre as espécies.
D-SCRIPT é descrito no artigo “Predição baseada em sequência de interações proteína-proteína: um modelo de aprendizagem profunda interpretável com reconhecimento de estrutura” de Sam Sledzieski, Rohit Singh, Lenore Cowen e Bonnie Berger.
Página inicial
Documentação