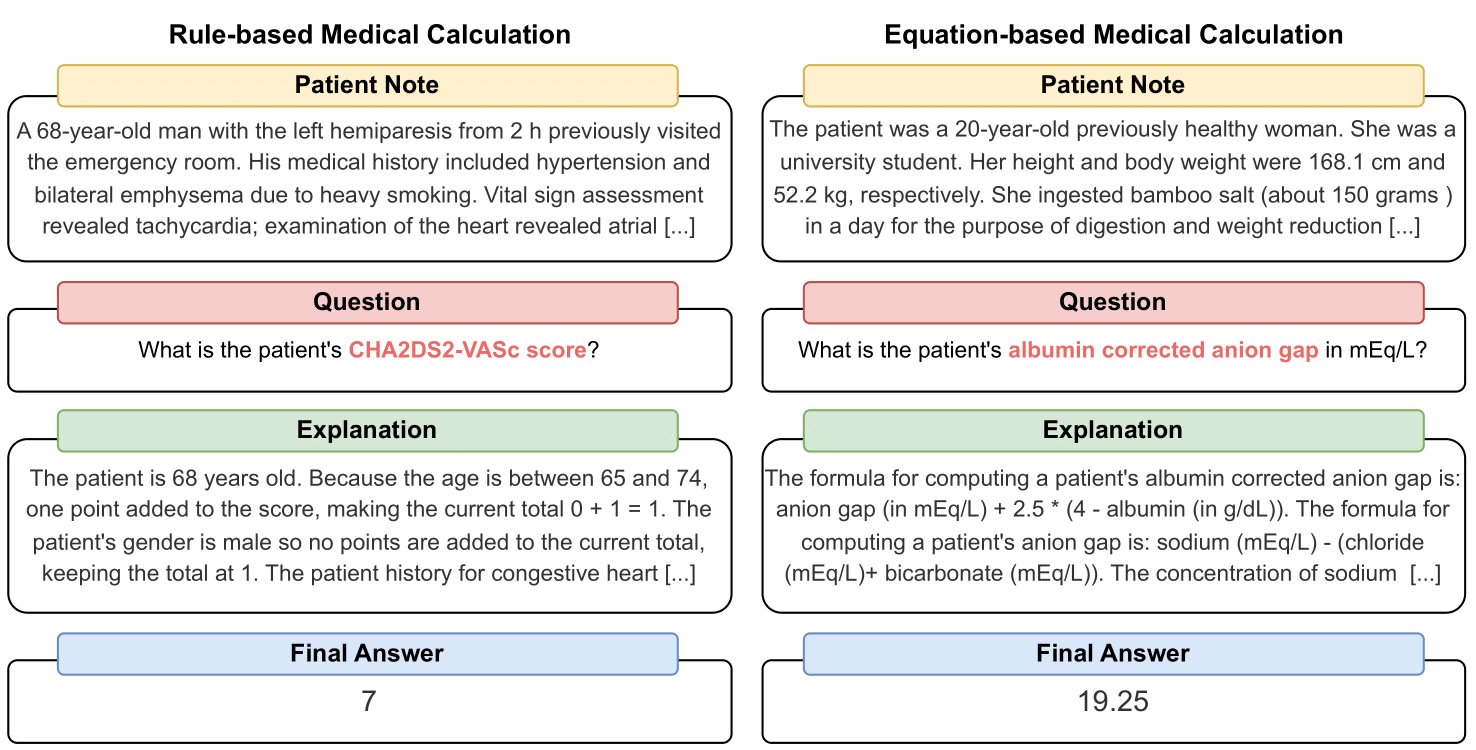
MedCalc-Bench é o primeiro conjunto de dados de cálculo médico usado para avaliar a capacidade dos LLMs de servir como calculadoras clínicas. Cada instância no conjunto de dados consiste em uma nota do paciente, uma pergunta solicitando o cálculo de um valor clínico específico, um valor de resposta final e uma solução passo a passo explicando como a resposta final foi obtida. Nosso conjunto de dados cobre 55 tarefas de cálculo diferentes que são cálculos baseados em regras ou cálculos baseados em equações. Este conjunto de dados contém um conjunto de dados de treinamento de 10.053 instâncias e um conjunto de dados de teste de 1.047 instâncias.
Ao todo, esperamos que nosso conjunto de dados e benchmark sirvam como um apelo para melhorar as habilidades de raciocínio computacional dos LLMs em ambientes médicos.
Nossa pré-impressão está disponível em: https://arxiv.org/abs/2406.12036.
Para baixar o CSV para o conjunto de dados de avaliação MedCalc-Bench, baixe o arquivo test_data.csv dentro da pasta dataset deste repositório. Você também pode baixar a divisão do conjunto de testes do HuggingFace em https://huggingface.co/datasets/ncbi/MedCalc-Bench.
Além das 1.047 instâncias de avaliação, também fornecemos um conjunto de dados de treinamento de 10.053 instâncias que podem ser usadas para ajustar LLMs de código aberto (consulte a Seção C do Apêndice). Os dados de treinamento podem ser encontrados no arquivo dataset/train_data.csv.zip e podem ser descompactados para obter train_data.csv . Este conjunto de dados de treinamento também pode ser encontrado na divisão do trem do link HuggingFace.
Cada instância no conjunto de dados contém as seguintes informações:
Para instalar todos os pacotes necessários para este projeto, execute o seguinte comando: conda env create -f environment.yml . Este comando criará o ambiente conda medcalc-bench . Para executar modelos OpenAI, você precisará fornecer sua chave OpenAI neste ambiente conda. Você pode fazer isso executando o seguinte comando no ambiente medcalc-bench : export OPENAI_API_KEY = YOUR_API_KEY , onde YOUR_API_KEY é sua chave de API OpenAI. Você também precisará fornecer seu token HuggingFace neste ambiente executando o seguinte comando: export HUGGINGFACE_TOKEN=your_hugging_face_token , onde your_hugging_face_token é seu token huggingface.
Para reproduzir a Tabela 2 do artigo, primeiro cd na pasta evaluation . Em seguida, execute o seguinte comando: python run.py --model <model_name> and --prompt <prompt_style> .
As opções para --model estão abaixo:
As opções para --prompt estão abaixo:
A partir disso, você obterá um arquivo jsonl exibindo o status de cada pergunta: Ao executar run.py , os resultados serão salvos em um arquivo chamado <model>_<prompt>.jsonl . Este arquivo pode ser encontrado na pasta de outputs .
Cada instância no jsonl terá os seguintes metadados associados a ela:
{
"Row Number": Row index of the item,
"Calculator Name": Name of calculation task,
"Calculator ID": ID of the calculator,
"Category": type of calculation (risk, severity, diagnosis for rule-based calculators and lab, risk, physical, date, dosage for equation-based calculators),
"Note ID": ID of the note taken directly from MedCalc-Bench,
"Patient Note": Paragraph which is the patient note taken directly from MedCalc-Bench,
"Question": Question asking for a specific medical value to be computed,
"LLM Answer": Final Answer Value from LLM,
"LLM Explanation": Step-by-Step explanation by LLM,
"Ground Truth Answer": Ground truth answer value,
"Ground Truth Explanation": Step-by-step ground truth explanation,
"Result": "Correct" or "Incorrect"
}
Além disso, fornecemos a precisão média e a porcentagem de desvio padrão para cada subcategoria em um json intitulado results_<model>_<prompt_style>.json . A precisão cumulativa e o desvio padrão entre todas as 1.047 instâncias podem ser encontrados na chave “geral” do JSON. Este arquivo pode ser encontrado na pasta de results .
Além dos resultados da Tabela 2 no artigo principal, também solicitamos aos LLMs que escrevessem código para realizar aritmética, em vez de deixar que o LLM fizesse isso sozinho. Os resultados para isso podem ser encontrados no Apêndice D. Devido à computação limitada, executamos apenas os resultados para GPT-3.5 e GPT-4. Para examinar os prompts e executar com esta configuração, examine o arquivo generate_code_prompt.py na pasta evaluation .
Para executar este código, basta cd na pasta evaluations e executar o seguinte: python generate_code_prompt.py --gpt <gpt_model> . As opções para <gpt_model> são 4 para executar GPT-4 ou 35 para executar GPT-3.5-turbo-16k. Os resultados serão salvos em um arquivo jsonl chamado: code_exec_{model_name}.jsonl na pasta de outputs . Observe que, neste caso, model_name será gpt_4 se você optar por executar usando GPT-4. Caso contrário, model_name será gpt_35_16k se você optou por executar com GPT-3.5-turbo.
Os metadados de cada instância no arquivo jsonl para os resultados do interpretador de código são as mesmas informações de instância fornecidas na seção acima. A única diferença é que armazenamos o histórico de bate-papo LLM entre o usuário e o assistente e temos uma chave "LLM Chat History" em vez da chave "LLM Explanation". Além disso, a subcategoria e a precisão geral são armazenadas em um arquivo JSON denominado results_<model_name>_code_augmented.json . Este JSON está localizado na pasta de results .
Esta pesquisa foi apoiada pelo Programa de Pesquisa Intramural do NIH, Biblioteca Nacional de Medicina. Além disso, as contribuições feitas por Soren Dunn foram feitas usando o recurso avançado de computação e dados Delta, que é apoiado pela National Science Foundation (prêmio OAC tel:2005572) e pelo Estado de Illinois. Delta é um esforço conjunto da Universidade de Illinois Urbana-Champaign (UIUC) e do seu Centro Nacional para Aplicações de Supercomputação (NCSA).
Para a curadoria das anotações dos pacientes no MedCalc-Bench, usamos apenas anotações dos pacientes disponíveis publicamente a partir de artigos de relatos de casos publicados no PubMed Central e vinhetas anônimas de pacientes geradas por médicos. Como tal, nenhuma informação pessoal identificável de saúde é revelada neste estudo. Embora o MedCalc-Bench seja projetado para avaliar as capacidades de cálculo médico dos LLMs, deve-se observar que o conjunto de dados não se destina ao uso diagnóstico direto ou à tomada de decisões médicas sem revisão e supervisão por um profissional clínico. Os indivíduos não devem mudar o seu comportamento de saúde apenas com base no nosso estudo.
Conforme descrito na Seção 1, as calculadoras médicas são comumente usadas no ambiente clínico. Com o interesse crescente no uso de LLMs para aplicações específicas de domínio, os profissionais de saúde podem solicitar diretamente chatbots como o ChatGPT para realizar tarefas de cálculo médico. No entanto, as capacidades dos LLMs nestas tarefas são atualmente desconhecidas. Uma vez que os cuidados de saúde são um domínio de alto risco e cálculos médicos errados podem levar a consequências graves, incluindo diagnósticos errados, planos de tratamento inadequados e potenciais danos aos pacientes, é crucial avaliar minuciosamente o desempenho dos LLMs em cálculos médicos. Surpreendentemente, os resultados da avaliação em nosso conjunto de dados MedCalc-Bench mostram que todos os LLMs estudados têm dificuldades nas tarefas de cálculo médico. O modelo GPT-4 mais capaz atinge apenas 50% de precisão com aprendizado único e solicitação de cadeia de pensamento. Como tal, nosso estudo indica que os atuais LLMs ainda não estão prontos para serem usados em cálculos médicos. Deve-se notar que, embora pontuações altas no MedCalc-Bench não garantam excelência em tarefas de cálculo médico, a falha neste conjunto de dados indica que os modelos não devem ser considerados para tais fins. Em outras palavras, acreditamos que a aprovação no MedCalc-Bench deve ser uma condição necessária (mas não suficiente) para que um modelo seja utilizado para cálculo médico.
Para quaisquer alterações neste conjunto de dados (ou seja, adicionar novas notas ou calculadoras), atualizaremos as instruções README, test_set.csv e train_set.csv. Ainda manteremos versões mais antigas desses conjuntos de dados em um archive/ pasta. Também atualizaremos conjuntos de treinamento e teste para HuggingFace.
Esta ferramenta mostra os resultados de pesquisas realizadas no Ramo de Biologia Computacional, NCBI/NLM. As informações produzidas neste site não se destinam ao uso diagnóstico direto ou à tomada de decisões médicas sem revisão e supervisão de um profissional clínico. Os indivíduos não devem alterar o seu comportamento de saúde apenas com base nas informações produzidas neste site. O NIH não verifica de forma independente a validade ou utilidade das informações produzidas por esta ferramenta. Se você tiver dúvidas sobre as informações produzidas neste site, consulte um profissional de saúde. Mais informações sobre a política de isenção de responsabilidade do NCBI estão disponíveis.
Dependendo da calculadora, nosso conjunto de dados consiste em notas que foram projetadas a partir de funções baseadas em modelos implementadas em Python, escritas à mão por médicos ou retiradas de nosso conjunto de dados, Open-Patients.
Open-Patients é um conjunto de dados agregado de 180 mil anotações de pacientes provenientes de três fontes diferentes. Temos autorização para usar o conjunto de dados de todas as três fontes. A primeira fonte são as perguntas USMLE do MedQA, que são lançadas sob a licença do MIT. A segunda fonte do nosso conjunto de dados é o Trec Clinical Decision Support e o Trec Clinical Trial, que estão disponíveis para redistribuição porque ambos são conjuntos de dados de propriedade do governo divulgados ao público. Por último, PMC-Patients é lançado sob a licença CC-BY-SA 4.0 e, portanto, temos permissão para incorporar PMC-Patients dentro de Open-Patients e MedCalc-Bench, mas o conjunto de dados deve ser lançado sob a mesma licença. Portanto, nossa fonte de notas, Open-Patients, e o conjunto de dados curado a partir dela, MedCalc-Bench, são ambos lançados sob a licença CC-BY-SA 4.0.
Com base na justificativa das regras de licença, tanto o Open-Patients quanto o MedCalc-Bench cumprem a licença CC-BY-SA 4.0, mas os autores deste artigo assumirão toda a responsabilidade em caso de violação de direitos.
@misc { khandekar2024medcalcbench ,
title = { MedCalc-Bench: Evaluating Large Language Models for Medical Calculations } ,
author = { Nikhil Khandekar and Qiao Jin and Guangzhi Xiong and Soren Dunn and Serina S Applebaum and Zain Anwar and Maame Sarfo-Gyamfi and Conrad W Safranek and Abid A Anwar and Andrew Zhang and Aidan Gilson and Maxwell B Singer and Amisha Dave and Andrew Taylor and Aidong Zhang and Qingyu Chen and Zhiyong Lu } ,
year = { 2024 } ,
eprint = { 2406.12036 } ,
archivePrefix = { arXiv } ,
primaryClass = { id='cs.CL' full_name='Computation and Language' is_active=True alt_name='cmp-lg' in_archive='cs' is_general=False description='Covers natural language processing. Roughly includes material in ACM Subject Class I.2.7. Note that work on artificial languages (programming languages, logics, formal systems) that does not explicitly address natural-language issues broadly construed (natural-language processing, computational linguistics, speech, text retrieval, etc.) is not appropriate for this area.' }
}