Conjunto de fluxos de trabalho moleculares e computacionais para sequenciamento espacial total de RNA (STRS)
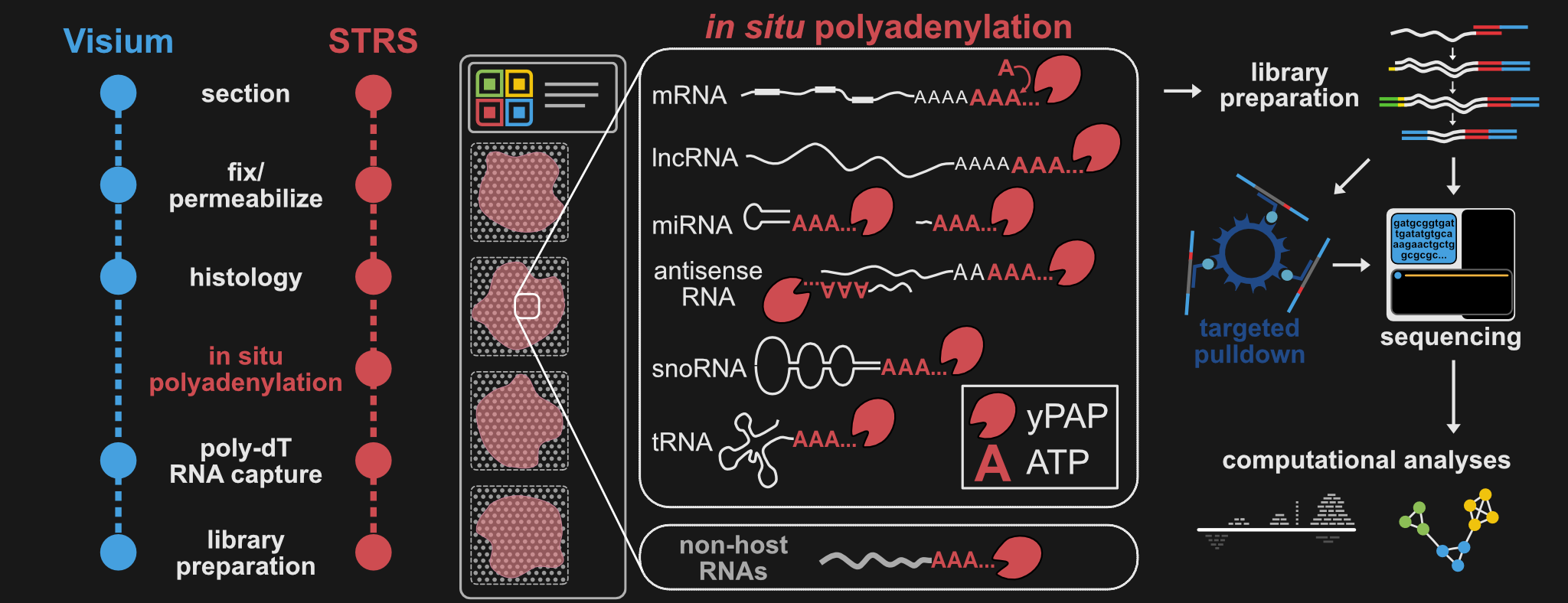
Este repositório contém todos os protocolos, pipelines e scripts que você precisa para executar e analisar STRS em quase qualquer amostra!
txg_snake , um pipeline de trabalho em andamento para bibliotecas 10x, especialmente conjuntos de dados STRS!McKellar et al, Nature Biotechnology , 2022
Link de pré-impressão bioRxiv
manuscripts : arquivos .pdf da pré-impressão e do manuscrito subsequente (espero!)
protocols : documentos Microsoft Word e .pdf dos protocolos usados aqui
pipelines : fluxos de trabalho Snakemake usados em nosso estudo. Isso inclui pipelines de alinhamento para dados STRS (kallisto, STARsolo e miRge3.0), pequenos dados RNAseq (STAR e miRge3.0), SmartSeqTotal (kallisto) e VASAdrop (kallisto)
scripts : Todos os outros códigos que usamos! Contém principalmente scripts R e funções utilitárias usadas em nossas análises espaciais.
references : informações e scripts sobre genomas de referência e anotações usadas neste estudo
resources : metadados variados, listas de genes e outras informações que usamos para analisar nossos dados, consulte os arquivos README em cada subdiretório para obter mais detalhes
Conjuntos de dados STRS: GSE200481
Dados SkM/coração pequenos RNAseq: GSE200480
dados de RNAseq total de núcleo único (linha celular C2C12): GSE209780
Dados do Small RNA Atlas (Isakova et al, PNAS , 2020): GSE119661
Dados SmartSeqTotal (Isakova et al, PNAS , 2021): GSE151334
Dados VASAdrop (Salmen et al, Nature Biotechnology , 2022): GSE176588
Iwijn De Vlaminck ([email protected])
Benjamin D. Cosgrove ([email protected])
David W. McKellar ([email protected])