Este projeto consiste em diferentes métodos de amostragem da população e avaliação de diferentes métodos. Incluímos muitas situações que podem causar viés na estimativa do nível de infecção com base na amostra, incluindo não respondedores, taxa de falsos positivos/negativos, capacidade de perfil de transmissão para pacientes durante o período de infecção. Com base no modelo EpiABM, este pacote também pode gerar o melhor método de amostragem executando simulações de transmissão de doenças para ver o erro de previsão de cada método de amostragem.
O EpiOS ainda não está disponível no PyPI, mas o módulo pode ser instalado localmente pelo pip. O diretório deve primeiro ser baixado em sua máquina local e depois instalado usando o comando:
pip install -e .Recomendamos também que você instale o modelo EpiABM para gerar os dados de simulação de infecção. Você pode primeiro baixar o pyEpiabm para qualquer local da sua máquina e depois instalá-lo usando o comando:
pip install -e path/to/pyEpiabm As documentações podem ser acessadas através do crachá docs acima.
Aqui está um diagrama de classes UML para nosso projeto: 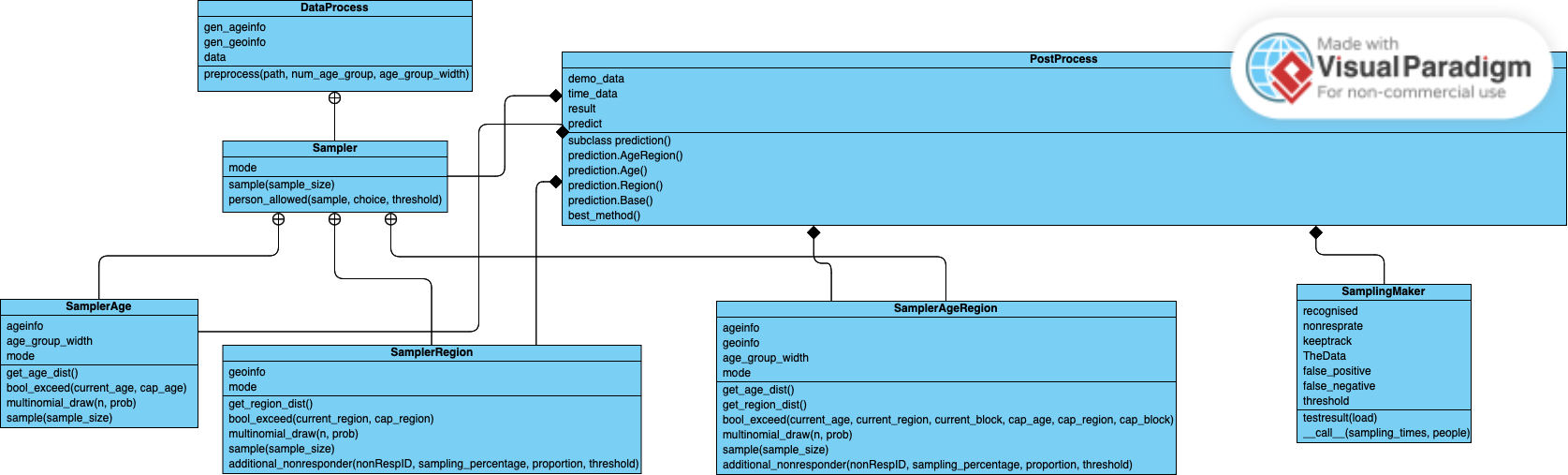
O arquivo params.py inclui todos os parâmetros necessários neste modelo. Além disso, os arquivos da pasta input são exemplos de arquivos temporários gerados durante o pré-processamento dos dados. Ele será usado pelas classes de amostragem. O parâmetro data_store_path em cada classe de amostra é o caminho para armazenar esses arquivos.
PostProcess para gerar gráficos Primeiramente, você precisa definir um novo objeto PostProcess e inserir os dados demodata demográficos e os dados de timedata de infecção gerados a partir do pyEpiabm. Em segundo lugar, você pode usar PostProcess.predict para realizar previsões com base em diferentes métodos de amostragem. Você pode chamar diretamente o método de amostragem que deseja usar como método; em seguida, especifique os pontos de tempo para amostrar e o tamanho da amostra. Aqui, usaremos AgeRegion como método de amostragem, [0, 1, 2, 3, 4, 5] como pontos no tempo a serem amostrados e 3 como o tamanho da amostra. Por último, você pode especificar se deseja considerar os não respondedores e se deseja comparar seus resultados com os dados verdadeiros, especificando os parâmetros non_responder e comparison .
Por exemplo de código, você pode ver o seguinte:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)Agora você terá sua figura salva no caminho indicado!
PostProcess para selecionar o melhor método de amostragem Primeiramente, você precisa definir um novo objeto PostProcess e inserir os dados demodata demográficos e os dados de timedata de infecção gerados a partir do pyEpiabm. Em segundo lugar, você pode usar PostProcess.best_method para comparar o desempenho de diferentes métodos de amostragem. Você pode fornecer métodos que deseja comparar; em seguida, especifique os intervalos de amostragem para amostrar e o tamanho da amostra. Em terceiro lugar, você pode especificar se deseja considerar os não respondedores e se deseja comparar seus resultados com os dados verdadeiros, especificando os parâmetros non_responder e comparison . Além disso, como os métodos de amostragem são estocásticos, é possível especificar o número de iterações executadas para obter o desempenho médio. Além disso, parallel_computation pode ser ativada para acelerar. Por último, você pode ativar hyperparameter_autotune para encontrar automaticamente a melhor combinação de hiperparâmetros.
Por exemplo de código, você pode ver o seguinte:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed