AMRICA (AMR Inspector for Cross-Language Alignments) é uma ferramenta simples para alinhar e representar visualmente AMRs (Banarescu, 2013), tanto para contextos bilíngues quanto para acordo monolíngue entre anotadores. Baseia-se e amplia o sistema Smatch (Cai, 2012) para identificar concordância entre anotadores AMR.
Também é possível usar AMRICA para visualizar alinhamentos manuais que você mesmo editou ou compilou (veja Sinalizadores Comuns).
Baixe a fonte python do github.
Presumimos que você tenha pip . Para instalar as dependências (supondo que você já tenha as dependências do Graphviz mencionadas abaixo), basta executar:
pip install argparse_config networkx==1.8 pygraphviz pynlpl
pygraphviz requer que o graphviz funcione. No Linux, pode ser necessário instalar graphviz libgraphviz-dev pkg-config . Além disso, para preparar dados de alinhamento bilíngues, você precisará do GIZA++ e possivelmente do JAMR.
./disagree.py -i sample.amr -o sample_out_dir/
Este comando irá ler os AMRs em sample.amr (separados por linhas vazias) e colocar suas visualizações graphviz em arquivos .png localizados em sample_out_dir/ .
Para gerar visualizações de alinhamentos Smatch, precisamos de um arquivo de entrada AMR com cada campo ::tok ou ::snt contendo sentenças tokenizadas, campos ::id com um ID de sentença e campos ::annotator ou ::anno com um ID de anotador. As anotações para uma frase específica são listadas sequencialmente e a primeira anotação é considerada o padrão ouro para fins de visualização.
Se você quiser visualizar apenas a anotação única por frase sem concordância entre anotadores, poderá usar um arquivo AMR com apenas um anotador. Nesse caso, os campos anotador e ID da frase são opcionais. O gráfico resultante será todo preto.
Para alinhamentos bilíngues, começamos com dois arquivos AMR, um contendo as anotações de destino e outro com as anotações de origem na mesma ordem, com campos ::tok e ::id para cada anotação. Se quisermos alinhamentos JAMR para qualquer um dos lados, incluímos esses em um campo ::alignments .
Os alinhamentos das frases devem estar na forma de dois arquivos .NBEST de alinhamento GIZA++, um origem-destino e um alvo-fonte. Para gerá-los, use o sinalizador --nbestalignments em seu arquivo de configuração GIZA++ definido para sua contagem nbest preferida.
Os sinalizadores podem ser definidos na linha de comando ou em um arquivo de configuração. A localização de um arquivo de configuração pode ser definida com -c CONF_FILE na linha de comando.
Além de --conf_file , existem vários outros sinalizadores que se aplicam a textos monolíngues e bilíngues. --outdir DIR é o único obrigatório e especifica o diretório no qual gravaremos os arquivos de imagem.
Os sinalizadores compartilhados opcionais são:
--verbose para imprimir frases à medida que as alinhamos.--no-verbose para substituir uma configuração padrão detalhada.--json FILE.json para gravar os gráficos de alinhamento em um arquivo .json.--num_restarts N para especificar o número de reinicializações aleatórias que o Smatch deve executar.--align_out FILE.csv para gravar os alinhamentos no arquivo.--align_in FILE.csv para ler os alinhamentos do disco em vez de executar o Smatch.--layout para modificar o parâmetro de layout para graphviz.Os arquivos .csv de alinhamento estão em um formato em que cada conjunto de correspondência de gráficos é separado por uma linha vazia e cada linha dentro de um conjunto contém um comentário ou uma linha indicando um alinhamento. Por exemplo:
3 它 - 1 it
2 多长 - -1
-1 - 2 take
Os campos separados por tabulação são o índice do nó de teste (conforme processado pelo Smatch), o rótulo do nó de teste, o índice do nó gold e o rótulo do nó gold.
O alinhamento monolíngue requer um sinalizador adicional, --infile FILE.amr , com FILE.amr definido como o local do arquivo AMR.
A seguir está um exemplo de arquivo de configuração:
[default]
infile: data/events_amr.txt
outdir: data/events_png/
json: data/events.json
verbose
No alinhamento bilíngue, há mais bandeiras obrigatórias.
--src_amr FILE para o arquivo AMR de anotação de origem.--tgt_amr FILE para o arquivo AMR de anotação de destino.--align_tgt2src FILE.A3.NBEST para o arquivo GIZA++ .NBEST alinhando destino-fonte (com destino como vcb1), gerado com --nbestalignments N--align_src2tgt FILE.A3.NBEST para o arquivo GIZA++ .NBEST alinhando origem ao destino (com origem como vcb1), gerado com --nbestalignments N Agora, se --nbestalignments N foi definido como >1, devemos especificá-lo com --num_aligned_in_file . Se quisermos contar apenas o topo --num_align_read também.
--nbestalignments é um sinalizador complicado de usar, porque só será gerado em uma execução de alinhamento final. Eu só consegui fazê-lo funcionar com as configurações padrão do GIZA++.
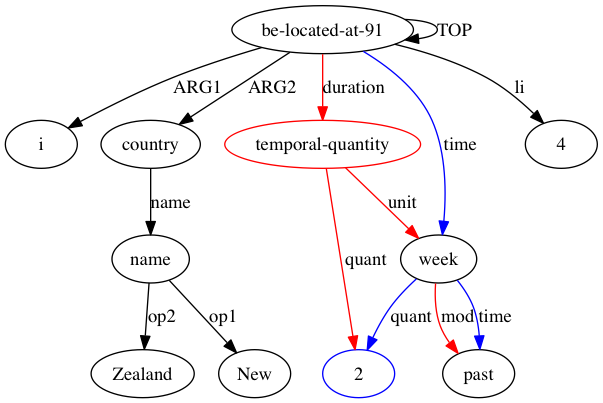
Como AMRICA é uma variação do Smatch, deve-se começar entendendo o Smatch. Smatch tenta identificar uma correspondência entre os nós variáveis de duas representações AMR da mesma sentença, a fim de medir a concordância entre anotadores. A correspondência deve ser selecionada para maximizar a pontuação do Smatch, que atribui um ponto para cada aresta que aparece em ambos os gráficos, enquadrando-se em três categorias. Cada categoria é ilustrada na seguinte anotação de “Não demorou muito”.
(t / take-10
:ARG0 (i / it)
:ARG1 (l2 / long
:polarity -))
(instance, t, take-10)(ARG0, t, i)(polarity, l2, -)Como o problema de encontrar a correspondência que maximiza a pontuação do Smatch é NP-completo, o Smatch usa um algoritmo de escalada para aproximar a melhor solução. Ele é propagado combinando cada nó com um nó que compartilha seu rótulo, se possível, e combinando os nós restantes no gráfico menor (doravante denominado alvo) aleatoriamente. Smatch então executa uma etapa encontrando a ação que aumentará mais a pontuação, trocando as correspondências de dois nós de destino ou movendo uma correspondência de seu nó de origem para um nó de origem sem correspondência. Ele repete esta etapa até que nenhuma etapa possa aumentar imediatamente a pontuação do Smatch.
Para evitar ótimos locais, o Smatch geralmente reinicia 5 vezes.
Para obter detalhes técnicos sobre o funcionamento interno do AMRICA, pode ser mais útil ler nosso documento de demonstração da NAACL.
AMRICA começa substituindo todos os nós constantes por nós variáveis que são instâncias do rótulo da constante. Isso é necessário para que possamos alinhar os nós constantes e também as variáveis. Portanto, os únicos pontos adicionados à pontuação AMRICA virão da correspondência entre arestas variáveis e rótulos de instância.
Enquanto o Smatch tenta combinar cada nó no gráfico menor com algum nó no gráfico maior, AMRICA remove correspondências que não aumentam a pontuação Smatch modificada, ou pontuação AMRICA.
AMRICA então gera arquivos de imagem a partir de gráficos graphviz dos alinhamentos. Se um nó ou aresta aparecer apenas nos dados dourados, ele será vermelho. Se esse nó ou aresta aparecer apenas nos dados de teste, ele será azul. Se o nó ou aresta corresponder em nosso alinhamento final, ele será preto.
No AMRICA, em vez de adicionar um ponto para cada rótulo de instância perfeitamente correspondente, adicionamos um ponto com base em uma pontuação de probabilidade no alinhamento desses rótulos. A pontuação de probabilidade ℓ(aLt,Ls[i]|Lt,Wt,Ls,Ws) com conjunto de rótulos de destino Lt, conjunto de rótulos de origem Ls, sentença de destino Wt, sentença de origem Ws e alinhamento aLt,Ls[i] mapeamento Lt[ i] em algum rótulo Ls[aLt,Ls[i]], é calculado a partir de uma probabilidade definida pelas seguintes regras:
Em geral, o AMRICA bilíngue parece exigir mais reinicializações aleatórias do que o AMRICA monolíngue para ter um bom desempenho. Esta contagem de reinicializações pode ser modificada com o sinalizador --num_restarts .
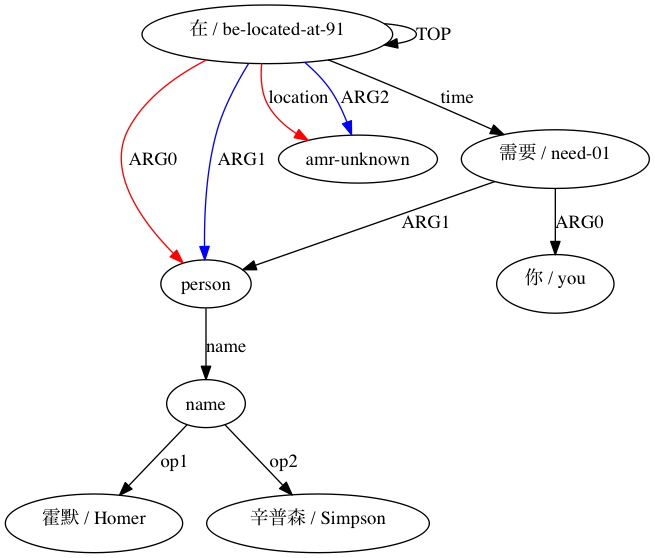
Podemos observar até que ponto o uso de aproximações do tipo Smatch (aqui, com 20 inicializações aleatórias) melhora a precisão em relação à seleção de correspondências prováveis a partir de dados de alinhamento brutos (inicialização inteligente). Para um emparelhamento declarado estruturalmente compatível por (Xue 2014).
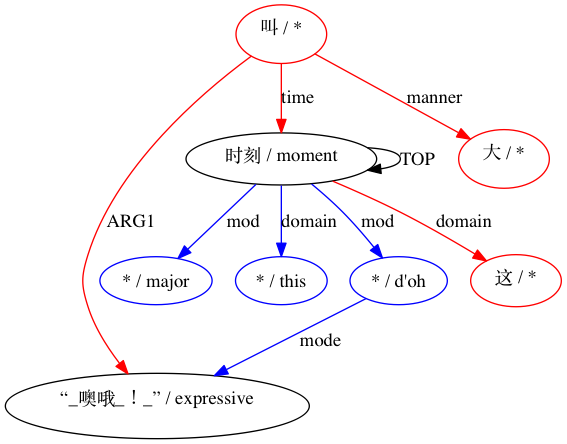
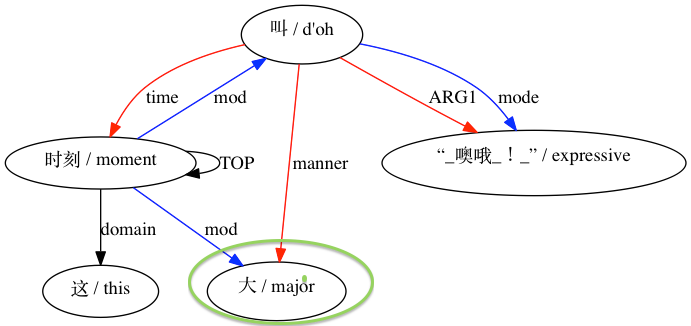
Para um emparelhamento considerado incompatível:
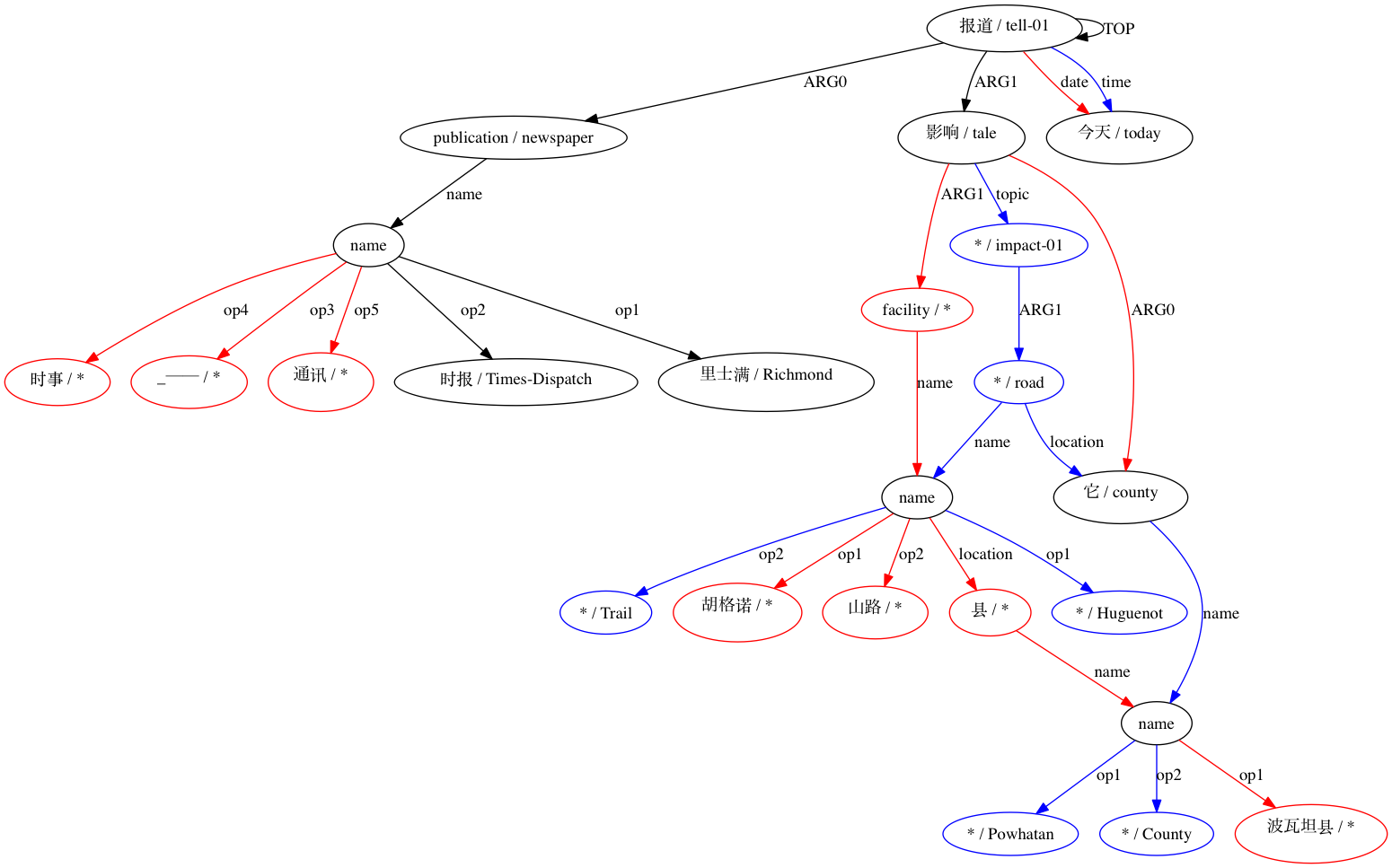
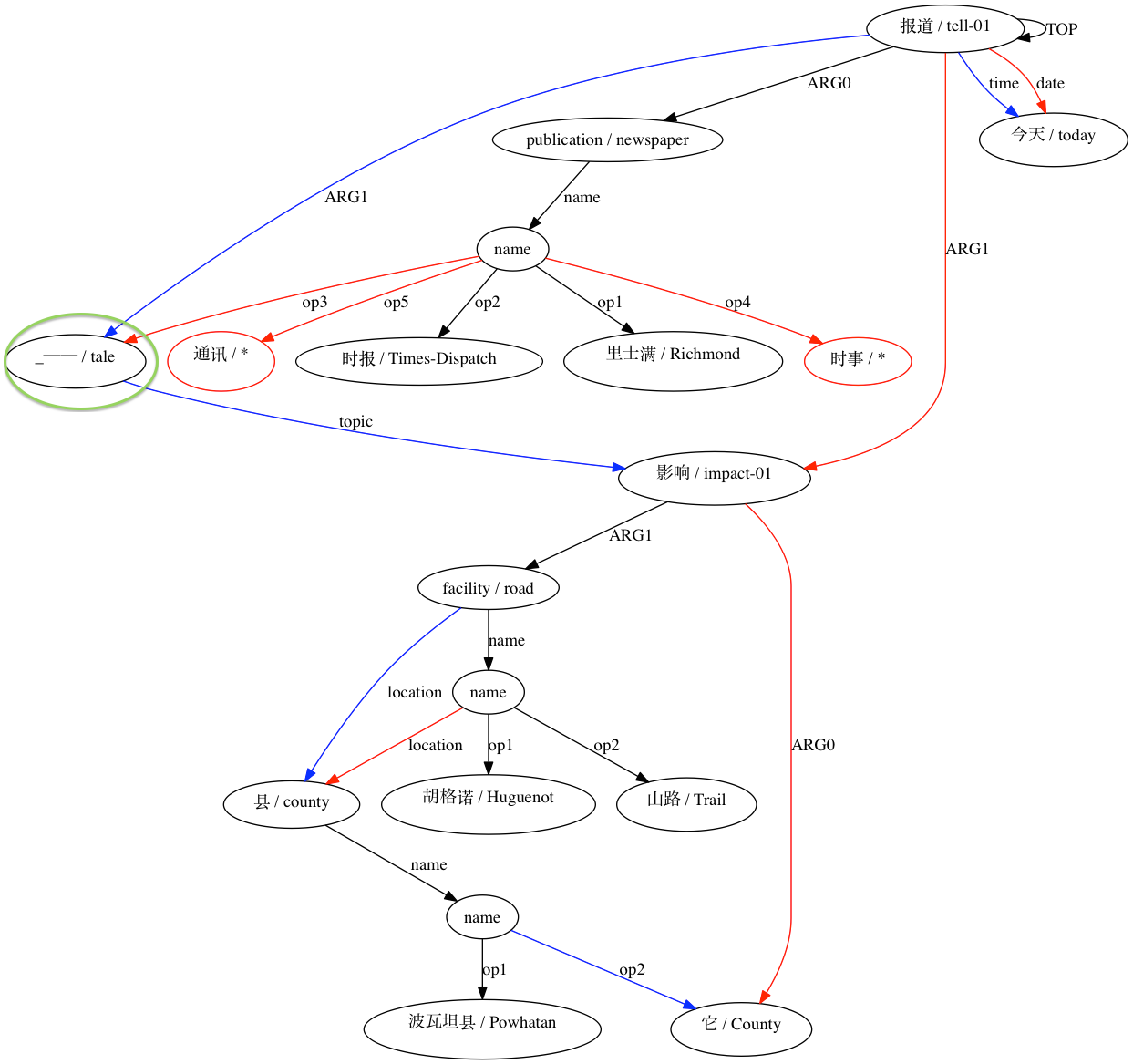
Este software foi desenvolvido parcialmente com o apoio da National Science Foundation (EUA) sob os prêmios 1349902 e 0530118. A Universidade de Edimburgo é uma instituição de caridade, registrada na Escócia, com número de registro SC005336.