Esteja ciente de que este repositório é apenas para fins de arquivamento. As funções ANCOM autônomas neste repositório não são mais mantidas. É altamente recomendável que os usuários usem as funções ancom ou ancombc em nosso pacote ANCOMBC R. Para relatórios de bugs, acesse o repositório ANCOMBC.
O código atual implementa ANCOM em conjuntos de dados transversais e longitudinais, permitindo ao mesmo tempo o uso de covariáveis. As seguintes bibliotecas precisam ser incluídas para que o código R seja executado:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )Adotamos a metodologia do ANCOM-II como etapa de pré-processamento para lidar com diferentes tipos de zeros antes de realizar a análise de abundância diferencial.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : quadro de dados ou matriz que representa a tabela OTU/SV observada com táxons em linhas ( rownames ) e amostras em colunas ( colnames ). Observe que esta é a tabela de abundância absoluta , não a transforme em tabela de abundância relativa (onde os totais das colunas são iguais a 1).meta_data : quadro de dados ou matriz de todas as variáveis e covariáveis de interesse.sample_var : Caractere. O nome da coluna que armazena IDs de amostra.group_var : Personagem. O nome do indicador do grupo. group_var é necessário para detectar zeros estruturais e valores discrepantes. Para as definições dos diferentes zeros (zero estrutural, zero outlier e zero amostral), consulte ANCOM-II.out_cut Fração numérica entre 0 e 1. Para cada táxon, observações com proporção de distribuição de mistura menor que out_cut serão detectadas como zeros outliers; enquanto observações com proporção de distribuição de mistura maior que 1 - out_cut serão detectadas como valores discrepantes.zero_cut : Fração numérica entre 0 e 1. Taxa com proporção de zeros maior que zero_cut não são incluídos na análise.lib_cut : Numérico. Amostras com tamanho de biblioteca menor que lib_cut não são incluídas na análise.neg_lb : Lógico. TRUE indica que um táxon seria classificado como zero estrutural no grupo experimental correspondente usando seu limite inferior assintótico. Mais especificamente, neg_lb = TRUE indica que você está usando ambos os critérios indicados na seção 3.2 do ANCOM-II para detectar zeros estruturais; Caso contrário, neg_lb = FALSE utilizará apenas a equação 1 da secção 3.2 da ANCOM-II para declaração de zeros estruturais. feature_table : um quadro de dados da tabela OTU pré-processada.meta_data : um quadro de dados de metadados pré-processados.structure_zeros : uma matriz consiste em 0 e 1s com 1 indicando que o táxon é identificado como um zero estrutural no grupo correspondente.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : quadro de dados que representa a tabela OTU/SV com táxons em linhas ( rownames ) e amostras em colunas ( colnames ). Pode ser o valor de saída de feature_table_pre_process . Observe que esta é a tabela de abundância absoluta , não a transforme em tabela de abundância relativa (onde os totais das colunas são iguais a 1).meta_data : quadro de dados de variáveis. Pode ser o valor de saída de feature_table_pre_process .struc_zero : Uma matriz consiste em 0 e 1s com 1 indicando que o táxon é identificado como um zero estrutural no grupo correspondente. Pode ser o valor de saída de feature_table_pre_process .main_var : Personagem. O nome da principal variável de interesse. ANCOM v2.1 atualmente suporta main_var categórico.p_adjust_method : Caractere. Especificar o método para ajustar valores p para comparações múltiplas. O padrão é “BH” (procedimento Benjamin-Hochberg).alpha : Nível de significância. O padrão é 0,05.adj_formula : Cadeia de caracteres representando a fórmula de ajuste (ver exemplo).rand_formula : sequência de caracteres que representa a fórmula para efeitos aleatórios em lme . Para obter detalhes, consulte ?lme .lme_control : uma lista especificando valores de controle para lme fit. Para obter detalhes, consulte ?lmeControl . 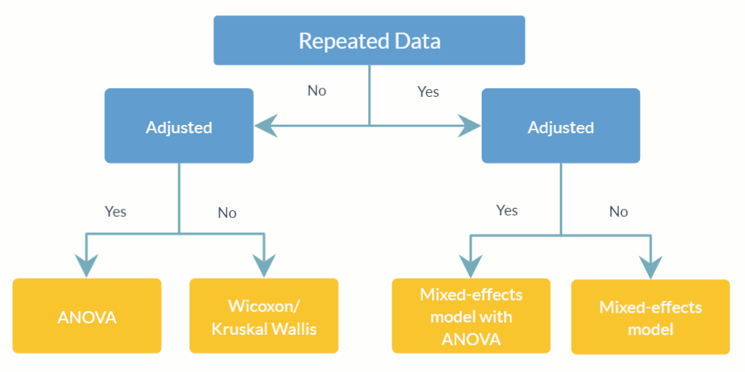
out : Um quadro de dados com a estatística W para cada táxon e colunas subsequentes que são indicadores lógicos de se uma OTU ou táxon é diferencialmente abundante sob uma série de pontos de corte (0,9, 0,8, 0,7 e 0,6). detected_0.7 é comumente usado. No entanto, você pode escolher detected_0.8 ou detected_0.9 se quiser ser mais conservador em seus resultados (FDR menor) ou usar detected_0.6 se quiser explorar mais descobertas (maior poder)
fig : Um objeto ggplot da trama do vulcão.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Aqui gostaríamos de saber se existem alguns zeros estruturais em diferentes níveis de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . Aqui gostaríamos de saber se existem alguns zeros estruturais em diferentes níveis de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . Aqui gostaríamos de saber se existem alguns zeros estruturais em diferentes níveis de delivery library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )