
Este repositório é a implementação oficial do CELL-E 2: Traduzindo proteínas em imagens e vice-versa com um transformador bidirecional de texto para imagem.
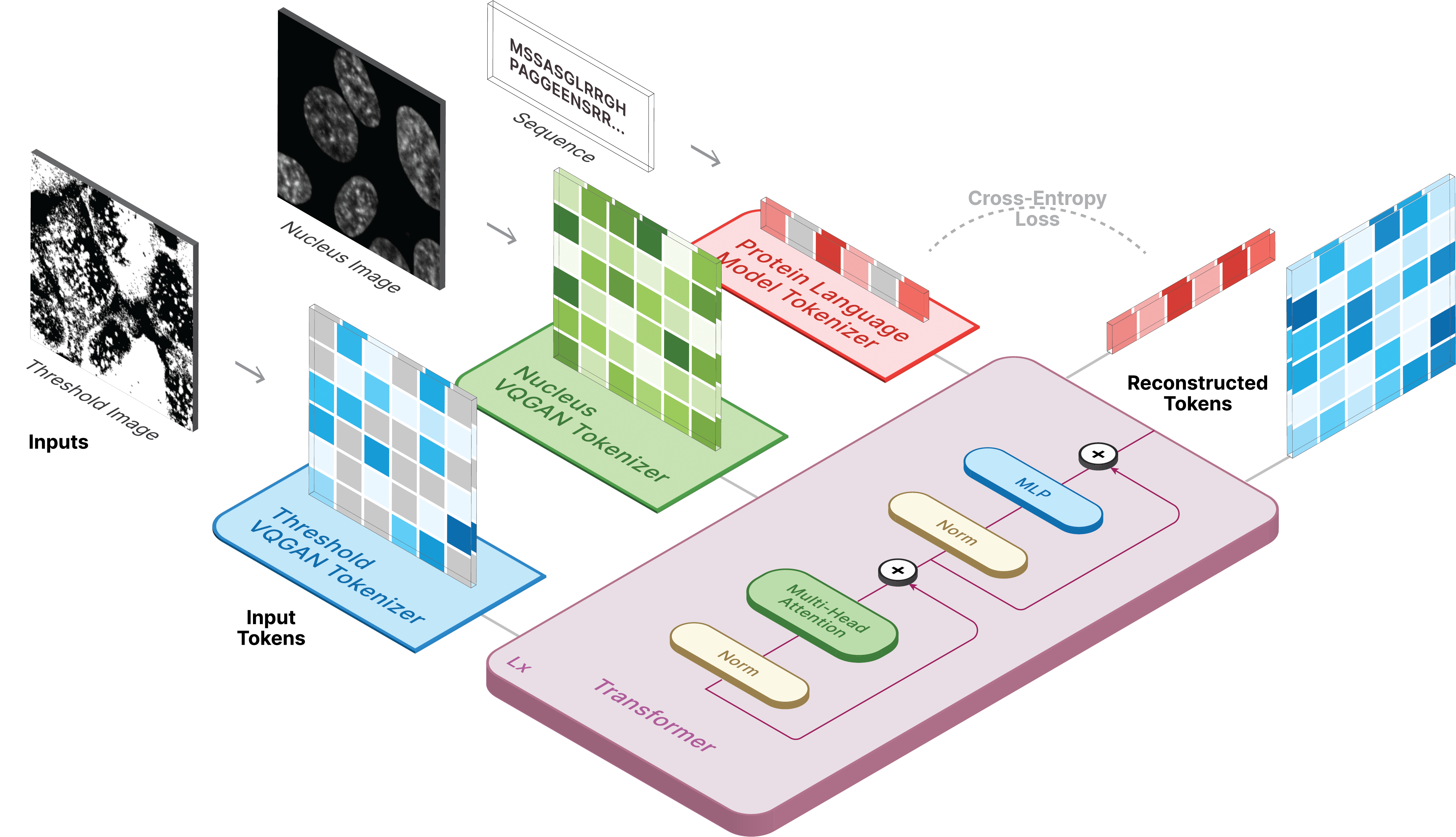
Crie um ambiente virtual e instale os pacotes necessários por meio de:
pip install -r requirements.txt
Em seguida, instale torch = 2.0.0 com a versão CUDA apropriada
Os modelos estão disponíveis no Hugging Face.
Também temos dois espaços disponíveis onde você pode fazer previsões com seus próprios dados!
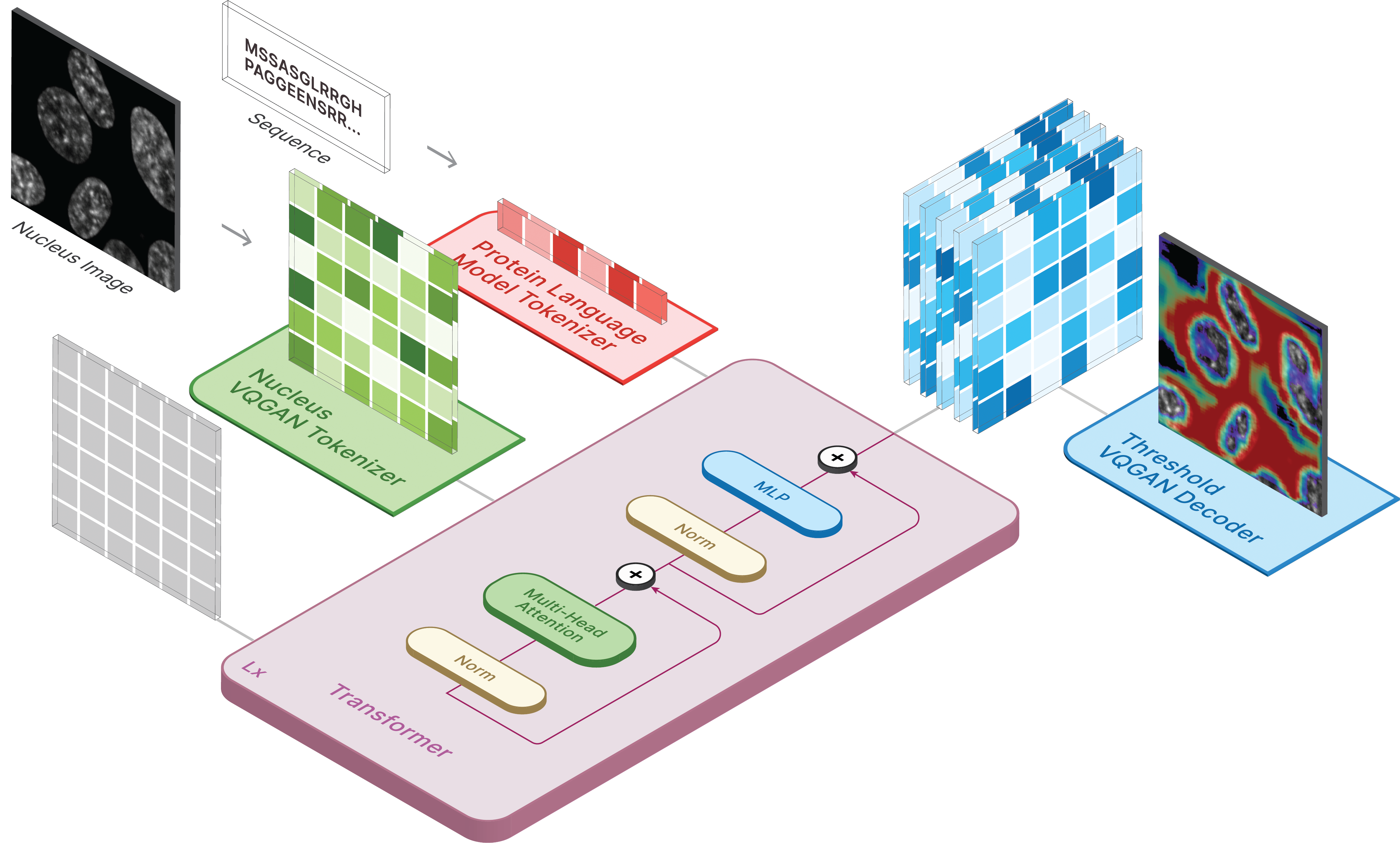
Para gerar imagens, defina o modelo salvo como ckpt_path. Este método pode ser instável, então consulte Demo.ipynb para ver outra forma de carregamento.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )O treinamento para CELL-E ocorre em 3 etapas:
Se estiver usando a imagem de limite de proteína, defina threshold: True para o conjunto de dados.
Usamos uma versão ligeiramente modificada do código dos transformadores domesticadores.
Para treinar, execute o seguinte script:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Consulte o repositório original para sinalizadores adicionais, como --gpus .
Fornecemos scripts para download de imagens Human Protein Atlas e OpenCell na pasta de scripts. Um data_csv é necessário para o dataloader. Você deve gerar um arquivo csv que contenha as colunas nucleus_image_path , protein_image_path , metadata_path , split (train ou val) e sequence (opcional). Supõe-se que este arquivo exista na mesma pasta data gerais que os arquivos de imagens e metadados.
Os metadados são um JSON que deve acompanhar cada sequência de proteína. Se uma sequência não aparecer em data_csv , ela deverá aparecer em metadata.json com uma chave chamada protein_sequence .
Adicionar mais informações aqui pode ser útil para consultar proteínas individuais. Eles podem ser recuperados por meio de retrieve_metadata , que cria uma variável self.metadata dentro do objeto de conjunto de dados.
Para treinar, execute o seguinte script:
python celle_main.py --base configs/celle.yaml -t True
Especifique --gpus no mesmo formato do VQGAN.
CELL-E contém as seguintes opções:
ckpt_path : Retoma o treinamento anterior do CELL-E 2. Modelo salvo com state_dictvqgan_model_path : modelo de imagem de proteína salvo (com state_dict) para codificador de imagem de proteínavqgan_config_path : yaml do modelo de imagem de proteína salvacondition_model_path : modelo de condição salva (núcleo) (com state_dict) para codificador de imagem de proteínacondition_config_path : modelo de condição salva (núcleo) yamlnum_images : 1 se estiver usando apenas o codificador de imagem de proteína, 2 se incluir o codificador de imagem de condiçãoimage_key : nucleus , target ou thresholddim : Dimensão da incorporação do modelo de linguagemnum_text_tokens : número total de tokens no modelo de linguagem (33 para ESM-2)text_seq_len : Número total de aminoácidos consideradosdepth : Profundidade do modelo do transformador, mais profundo geralmente é melhor ao custo de VRAMheads : número de cabeças usadas na atenção multicabeçasdim_head : tamanho das cabeças de atençãoattn_dropout : Taxa de abandono de atenção no treinamentoff_dropout : taxa de abandono feed-forward no treinamentoloss_img_weight : Ponderação aplicada à reconstrução da imagem. peso do texto = 1loss_text_weight : ponderação aplicada à reconstrução da imagem condicionada.stable : pesos das normas (para quando ocorrem gradientes explosivos)learning_rate : taxa de aprendizagem para o otimizador Adammonitor : Parâmetro usado para salvar modelos Por favor, cite-nos se você decidir usar nosso código para qualquer parte de sua pesquisa.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}