Carregamento rápido de dados em lote de arquivos BigWig contendo dados de trilhas epigenéticas e sequências correspondentes alimentadas por GPU para aplicações de aprendizado profundo.
Bigwig-loader depende principalmente da biblioteca rapidsai kvikio e cupy, ambas melhor instaladas usando conda/mamba. Bigwig-loader agora também pode ser instalado usando conda/mamba. Para criar um novo ambiente com o bigwig-loader instalado:
mamba create -n my-env -c rapidsai -c conda-forge -c bioconda -c dataloading bigwig-loaderOu adicione isto ao seu arquivo Environment.yml:
name : my-env
channels :
- rapidsai
- conda-forge
- bioconda
- dataloading
dependencies :
- bigwig-loadere atualizar:
mamba env update -f environment.ymlO Bigwig-loader também pode ser instalado usando pip em um ambiente que já possui a biblioteca rapidsai kvikio e o cupy instalados:
pip install bigwig-loaderEnvolvemos o BigWigDataset em um conjunto de dados iterável PyTorch que você pode usar diretamente:
# examples/pytorch_example.py
import pandas as pd
import torch
from torch . utils . data import DataLoader
from bigwig_loader import config
from bigwig_loader . pytorch import PytorchBigWigDataset
from bigwig_loader . download_example_data import download_example_data
# Download example data to play with
download_example_data ()
example_bigwigs_directory = config . bigwig_dir
reference_genome_file = config . reference_genome
train_regions = pd . DataFrame ({ "chrom" : [ "chr1" , "chr2" ], "start" : [ 0 , 0 ], "end" : [ 1000000 , 1000000 ]})
dataset = PytorchBigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
n_threads = 4 ,
return_batch_objects = True ,
)
# Don't use num_workers > 0 in DataLoader. The heavy
# lifting/parallelism is done on cuda streams on the GPU.
dataloader = DataLoader ( dataset , num_workers = 0 , batch_size = None )
class MyTerribleModel ( torch . nn . Module ):
def __init__ ( self ):
super (). __init__ ()
self . linear = torch . nn . Linear ( 4 , 2 )
def forward ( self , batch ):
return self . linear ( batch ). transpose ( 1 , 2 )
model = MyTerribleModel ()
optimizer = torch . optim . SGD ( model . parameters (), lr = 0.01 )
def poisson_loss ( pred , target ):
return ( pred - target * torch . log ( pred . clamp ( min = 1e-8 ))). mean ()
for batch in dataloader :
# batch.sequences.shape = n_batch (32), sequence_length (1000), onehot encoding (4)
pred = model ( batch . sequences )
# batch.values.shape = n_batch (32), n_tracks (2) center_bin_to_predict (500)
loss = poisson_loss ( pred [:, :, 250 : 750 ], batch . values )
print ( loss )
optimizer . zero_grad ()
loss . backward ()
optimizer . step () Um objeto Dataset independente de estrutura pode ser importado de bigwig_loader.dataset . Este objeto de conjunto de dados retorna tensores cupy. Os tensores Cupy aderem à interface do array cuda e podem ser transformados em cópia zero em tensores JAX ou tensorflow.
from bigwig_loader . dataset import BigWigDataset
dataset = BigWigDataset (
regions_of_interest = train_regions ,
collection = example_bigwigs_directory ,
reference_genome_path = reference_genome_file ,
sequence_length = 1000 ,
center_bin_to_predict = 500 ,
window_size = 1 ,
batch_size = 32 ,
super_batch_size = 1024 ,
batches_per_epoch = 20 ,
maximum_unknown_bases_fraction = 0.1 ,
sequence_encoder = "onehot" ,
)Veja o diretório de exemplos para mais exemplos.
Esta biblioteca destina-se ao carregamento de lotes de dados com a mesma dimensionalidade, o que permite algumas suposições que podem acelerar o processo de carregamento. Como pode ser visto no gráfico abaixo, ao carregar uma pequena quantidade de dados, o pyBigWig é muito rápido, mas não explora a natureza em lote do carregamento de dados para aprendizado de máquina.
No benchmark abaixo também criamos dataloaders PyTorch (com set_start_method('spawn')) usando pyBigWig para comparar com o cenário realista onde múltiplas CPUs seriam usadas por GPU. Vemos que o throughput do dataloader da CPU não aumenta linearmente com o número de CPUs e, portanto, fica difícil obter o throughput necessário para manter a GPU, treinando a rede neural, saturada durante as etapas de aprendizado.
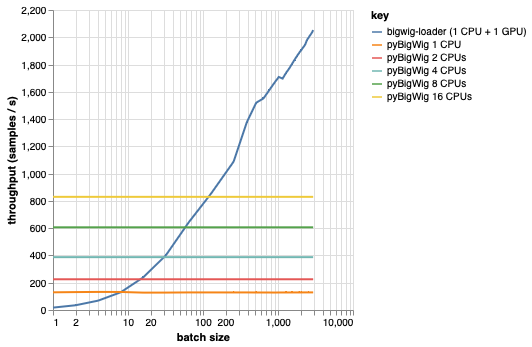
Este é o problema que o bigwig-loader resolve. Este é um exemplo de como usar o bigwig-loader:
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.yml Neste ambiente, você poderá executar pytest -v e ver os testes bem-sucedidos. NOTA: você precisa de uma GPU para usar o bigwig-loader!
Esta seção orienta você nas etapas necessárias para adicionar novas funcionalidades. Se algo não estiver claro, abra um problema.
git clone [email protected]:pfizer-opensource/bigwig-loadercd bigwig-loaderconda env create -f environment.ymlpip install -e '.[dev]'pre-commit install para instalar os ganchos de pré-commitOs testes estão no diretório de testes. Um dos testes mais importantes é test_against_pybigwig que garante que se houver um erro no pyBigWIg, também será no bigwig-loader.
pytest -vv .Quando os executores do GitHub com GPUs estiverem disponíveis, também gostaríamos de executar esses testes no CI. Mas, por enquanto, você pode executá-los localmente.
Se você usa esta biblioteca, considere citar:
Retel, Joren Sebastian, Andreas Poehlmann, Josh Chiou, Andreas Steffen e Djork-Arné Clevert. “Um carregador de dados de aprendizado de máquina rápido para trilhas epigenéticas de arquivos BigWig.” Bioinformática 40, não. 1 (1º de janeiro de 2024): btad767. https://doi.org/10.1093/bioinformatics/btad767.
@article {
retel_fast_2024,
title = { A fast machine learning dataloader for epigenetic tracks from {BigWig} files } ,
volume = { 40 } ,
issn = { 1367-4811 } ,
url = { https://doi.org/10.1093/bioinformatics/btad767 } ,
doi = { 10.1093/bioinformatics/btad767 } ,
abstract = { We created bigwig-loader, a data-loader for epigenetic profiles from BigWig files that decompresses and processes information for multiple intervals from multiple BigWig files in parallel. This is an access pattern needed to create training batches for typical machine learning models on epigenetics data. Using a new codec, the decompression can be done on a graphical processing unit (GPU) making it fast enough to create the training batches during training, mitigating the need for saving preprocessed training examples to disk.The bigwig-loader installation instructions and source code can be accessed at https://github.com/pfizer-opensource/bigwig-loader } ,
number = { 1 } ,
urldate = { 2024-02-02 } ,
journal = { Bioinformatics } ,
author = { Retel, Joren Sebastian and Poehlmann, Andreas and Chiou, Josh and Steffen, Andreas and Clevert, Djork-Arné } ,
month = jan,
year = { 2024 } ,
pages = { btad767 } ,
}