
(c) 2017 Timothy Becker e Wan-Ping Lee
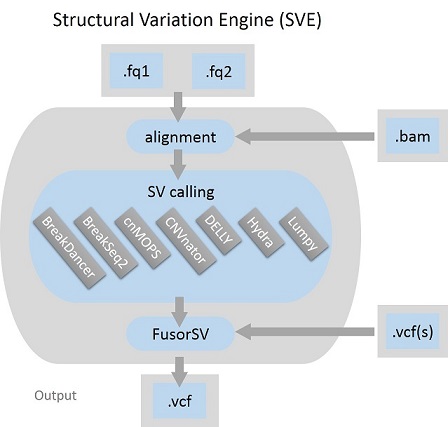
SVE é um mecanismo de execução baseado em script python para detecção de variação estrutural (SV) e pode ser usado para qualquer nível de entrada de dados, FASTQs brutos, BAMs alinhados ou formato de chamada variante (VCFs) e gera um VCF unificado como saída. Por design, o SVE consiste em alinhamento, realinhamento e o conjunto de algoritmos de chamada SV de última geração por padrão. São eles BreakDancer, BreakSeq, cnMOPS, CNVnator, DELLY, Hydra e LUMPY. O FusorSV também está incorporado, sendo uma abordagem de mineração de dados para avaliar o desempenho e mesclar conjuntos de chamadas de um conjunto de algoritmos de chamada SV.
Por favor, defina o ambiente ROOT.
export ROOTSYS=/ROOT_Build_Path
export LD_LIBRARY_PATH=$LD_LIBRARY_PATH:$ROOTSYS/lib
git clone --recursive https://github.com/TheJacksonLaboratory/SVE.git
cd SVE
makeVerifique os arquivos de cabeçalho python2.7 e modifique "CFLAGS_FUSOR_SV" no Makefile. Os arquivos de cabeçalho podem estar em "/usr/include/python2.7" e usar "CFLAGS_FUSOR_SV=-I /usr/include/python2.7".
make FusorSV
Ou você pode instalar o FusorSV por setup.py
cd SVE/scripts/FusorSV/
python setup.py build_ext --inplace
tar -zxvf data.tar.gz
Alternativamente, Dockerfile e imagem Docker são fornecidos. Observe que o sudo pode ser necessário para uso do docker, dependendo da configuração da sua máquina.
cd SVE
docker build .
Extraia a imagem do docker do repositório.
docker pull wanpinglee/sve
SVE é construído em /tools/SVE. Verifique a ajuda por
/tools/SVE/bin/sve
Leituras curtas no FASTQ serão mapeadas em relação ao FASTA fornecido e um BAM classificado será gerado.
bin/sve align [options] -r <FASTA> <FASTQ1 [FASTQ2]>
Se as leituras forem fornecidas no formato BAM, o realinhamento remapeará as leituras em relação ao FASTA e gerará um BAM classificado. Usamos SpeedSeq para realizar o realinhamento.
bin/sve realign -r <FASTA> <BAM>
Existem sete algoritmos de chamada SV que podem ser usados para chamadas SV. O VCF será gerado.
bin/sve call -r <FASTA> -g <hg19|hg38|others> -a <breakdancer|breakseq|cnvnator|hydra|delly|lumpy|cnmops> <BAM [BAM ...]>
Após a chamada, cada amostra pode ter vários VCFs dependendo de quantos chamadores foram usados. Colete VCFs de uma amostra em uma pasta.
Os vcfs devem usar SVE IDs para indicar os chamadores.
| ID SVE | Chamador |
|---|---|
| 4 | BreakDancer (v1.4.5) |
| 9 | cn.MOPS (v1.20) |
| 10 | CNVnator (v0.3.3) |
| 11 | DELLY (v2) |
| 14* | GenomaSTRiP |
| 17 | Hidra |
| 18 | GRUMOSO |
| 35 | BreakSeq (v2.2) |
| 0 | Verdade (opcional) |
Nota*: Devido a problemas de licença, GenomeSTRiP não está incorporado no SVE. No entanto, o modelo padrão FusorSV é capaz de lidar com GenomeSTRiP VCF.
Arquivos vcf de entrada de exemplo podem ser organizados da seguinte maneira. Observe que vcfFiles é o argumento para -i para FusorSV.
python scripts/FusorSV/FusorSV.py -f scripts/FusorSV/data/models/default.pickle -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
De acordo com S0.vcf, um novo modelo será gerado e os VCFs serão mesclados pelo novo modelo.
python scripts/FusorSV/FusorSV.py -L DEFAULT -r <FASTA> -i <vcfFiles>/ -p <THREADS> -o <OUT_DIR>
O projeto está licenciado sob a licença GPL-3.0. Consulte LICENÇA para obter detalhes.