Repositório contendo dados e código para genotipagem do guar ( Cyamopsis tetragonoloba (L.) Taub.) e projeto GWAS. Uma breve descrição do conteúdo do repositório:
O script make_consensus_vcf.py foi usado para gerar o conjunto de dados de variante final usando chamadas de variante brutas GATK-HC, NGSEP e TASSEL 5, o script make_snp_stats.py foi usado com as mesmas entradas para explorar as estatísticas de qualidade das chamadas de variante.
O notebook callset_refinement_filtering.ipynb foi usado para executar o controle de qualidade de variantes e construir o conjunto de dados filtrado final de variantes comuns.
O caderno genetic_analysis.ipynb foi usado para realizar a maioria das análises genéticas, incluindo PCA, análise de desequilíbrio de ligação e análise de associação baseada em modelo linear generalizado.
O script prepare_farmcpu.py foi usado para formatar os dados de entrada para a ferramenta FarmCPU (o gráfico QQ dos valores p resultantes é mostrado no artigo e abaixo).
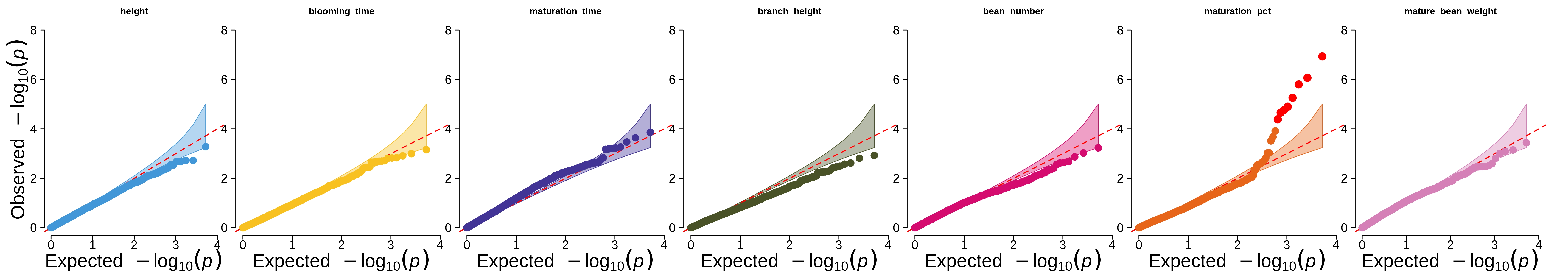
O arquivo admix_file_create.py foi usado para pré-processar genótipos para análise ADMIXTURE (parte disso foi feita usando Hail no genetic_analysis.ipynb ).
O script guar_stat.R foi usado para executar a análise estatística dos dados finais, conduzir a análise de associação genômica ampla usando a ferramenta FarmCPU e traçar os números principais
O script parse_vcf_fcpu.py foi usado para formatar a tabela final de genótipos SNP disponível como o arquivo genotypes.xlsx neste repositório.