MedSegDiff, uma estrutura baseada em modelo probabilístico de difusão (DPM) para segmentação de imagens médicas. O algoritmo é elaborado em nosso artigo MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model e MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer.
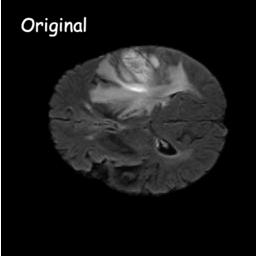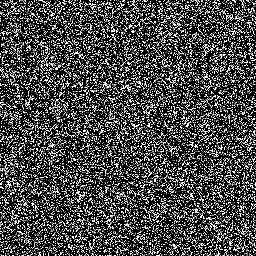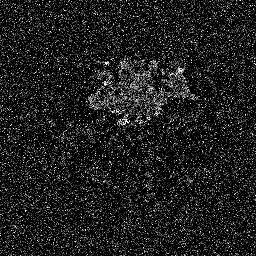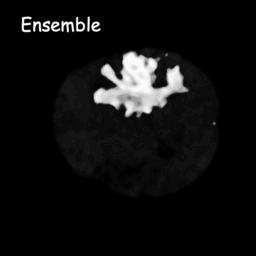 Os modelos de difusão funcionam destruindo dados de treinamento por meio da adição sucessiva de ruído gaussiano e, em seguida, aprendendo a recuperar os dados revertendo esse processo de ruído. Após o treinamento, podemos usar o modelo de difusão para gerar dados simplesmente passando o ruído amostrado aleatoriamente através do processo de remoção de ruído aprendido. Neste projeto, estendemos essa ideia à segmentação de imagens médicas. Utilizamos a imagem original como condição e geramos múltiplos mapas de segmentação a partir de ruídos aleatórios e, em seguida, realizamos a montagem deles para obter o resultado final. Esta abordagem captura a incerteza nas imagens médicas e supera os métodos anteriores em vários benchmarks.
Os modelos de difusão funcionam destruindo dados de treinamento por meio da adição sucessiva de ruído gaussiano e, em seguida, aprendendo a recuperar os dados revertendo esse processo de ruído. Após o treinamento, podemos usar o modelo de difusão para gerar dados simplesmente passando o ruído amostrado aleatoriamente através do processo de remoção de ruído aprendido. Neste projeto, estendemos essa ideia à segmentação de imagens médicas. Utilizamos a imagem original como condição e geramos múltiplos mapas de segmentação a partir de ruídos aleatórios e, em seguida, realizamos a montagem deles para obter o resultado final. Esta abordagem captura a incerteza nas imagens médicas e supera os métodos anteriores em vários benchmarks.
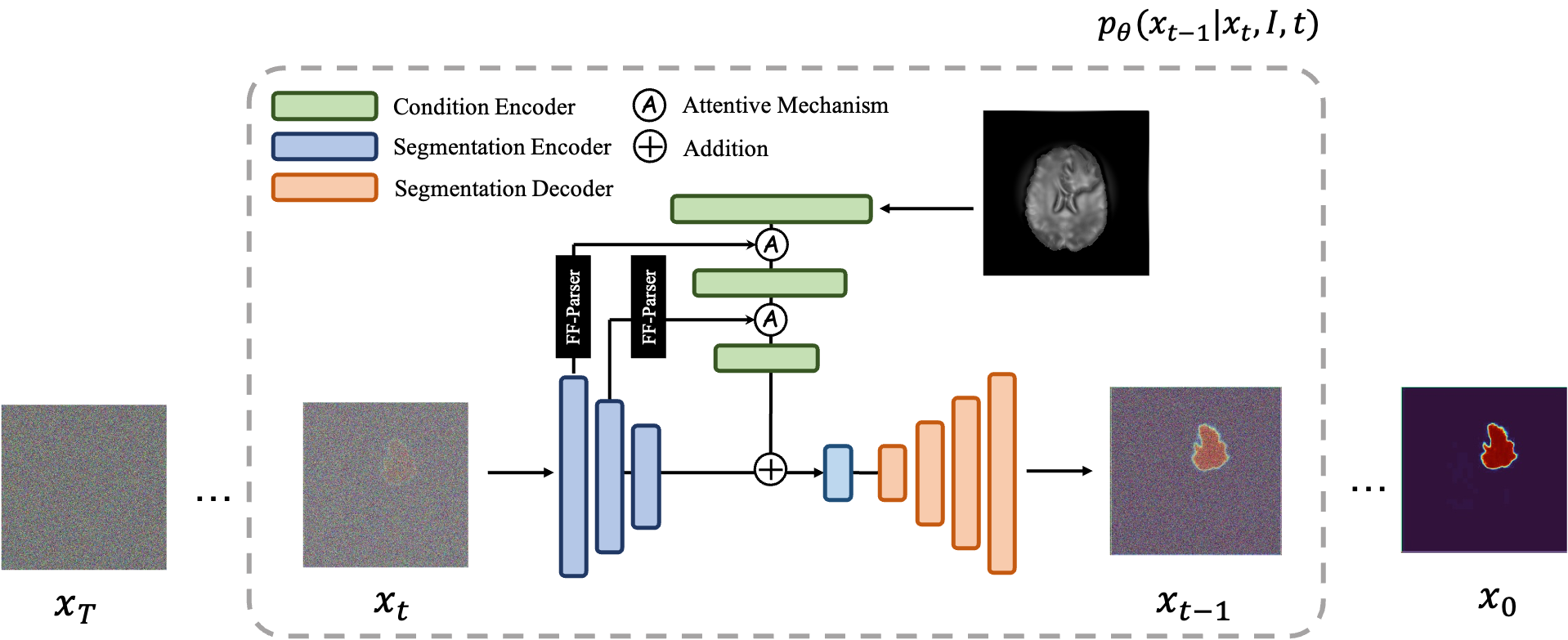 | 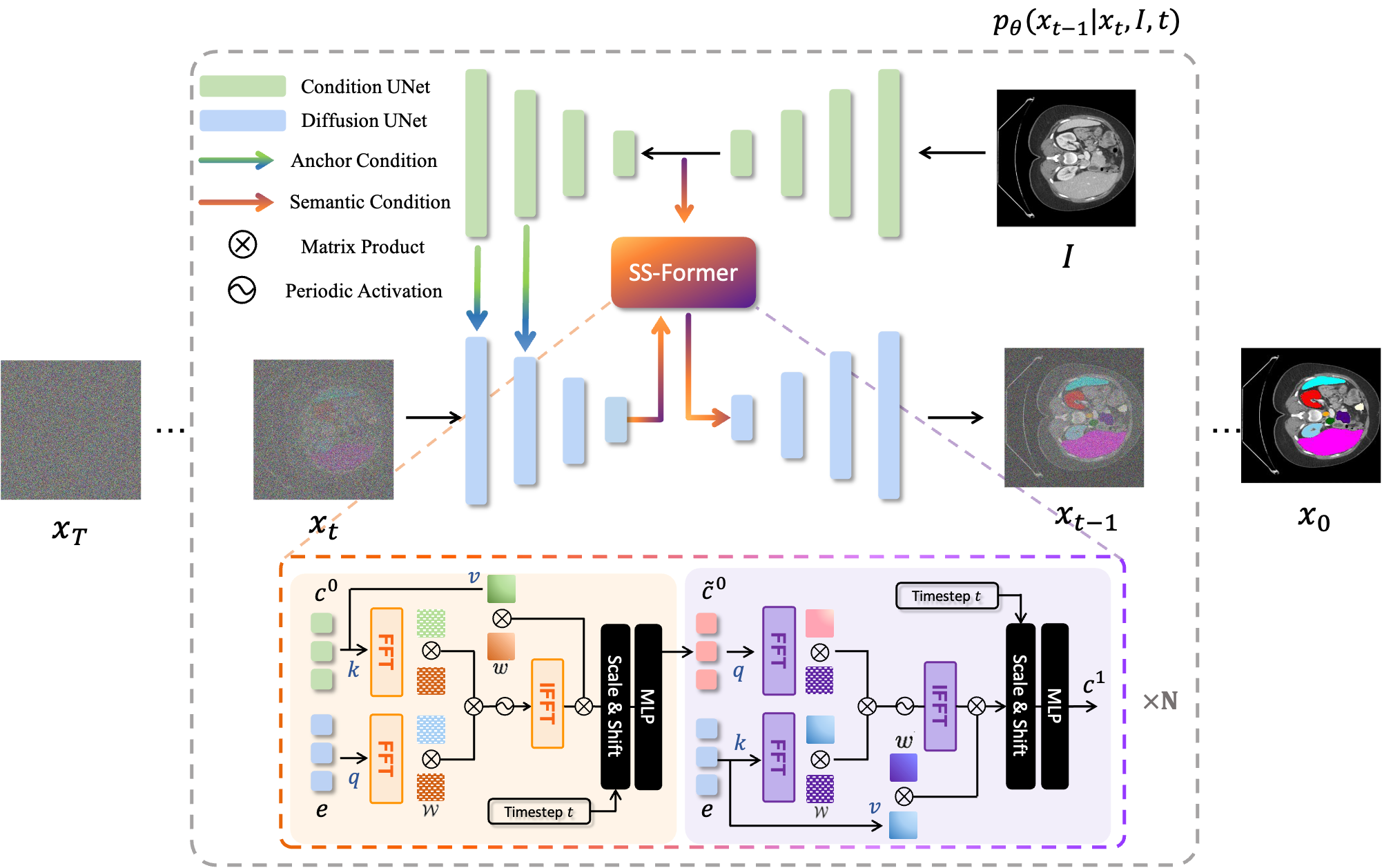 |
|---|---|
| MedSegDiff-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Para treinamento, execute: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Para amostragem, execute: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Para avaliação, execute python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
Por padrão, as amostras serão salvas em ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Para treinamento, execute: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Para amostragem, execute: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
É simples executar o MedSegDiff em outros conjuntos de dados. Basta escrever outro arquivo do carregador de dados seguindo ./guided_diffusion/isicloader.py ou ./guided_diffusion/bratsloader.py . Bem-vindo a problemas abertos se você encontrar algum problema. Agradeceríamos se você pudesse contribuir com suas extensões de conjunto de dados. Ao contrário das imagens naturais, as imagens médicas variam muito dependendo das diferentes tarefas. Expandir a generalização de um método requer o esforço de todos.
Para treinar um modelo fino, ou seja, MedSegDiff-B no artigo, defina os hiperparâmetros do modelo como:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
hiperparâmetros de difusão como:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Para acelerar a amostragem:
--diffusion_steps 50 --dpm_solver True
executado em várias GPUs:
--multi-gpu 0,1,2 (for example)
treinando hiperparâmetros como:
--lr 5e-5 --batch_size 8
e defina --num_ensemble 5 na amostragem.
A execução de cerca de 100.000 etapas de treinamento convergirá na maioria dos conjuntos de dados. Observe que embora a perda não diminua na maioria das etapas posteriores, a qualidade dos resultados ainda está melhorando. Tal processo também é observado nas demais aplicações DPM, como geração de imagens. Espero que alguém inteligente possa me dizer por quê?
Em breve publicarei seu desempenho em lotes menores (adequados para rodar em GPU de 24 GB) para necessidade de comparação?
Uma configuração para liberar todo o seu potencial é (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Em seguida, treine-o com o tamanho do lote --batch_size 64 e faça uma amostra com o número do conjunto --num_ensemble 25 .
Bem-vindo a contribuir com o MedSegDiff. Qualquer técnica pode melhorar o desempenho ou acelerar o algoritmo. Estou escrevendo o MedSegDiff V2, visando publicações do tipo periódicos Nature/CVPR. Fico feliz em listar os colaboradores como meus coautores.
Código copiado muito de openai/improved-diffusion, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion, guide-diffusion, bigmb/Unet-Segmentation-Pytorch-Nest -of-Unets, nnUnet, lucidrains/vit-pytorch
Por favor cite
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu