Importante
Confira o novo modelo DL tabular: TabM
arXiv? Pacote Python Outros projetos DL tabulares
Esta é a implementação oficial do artigo "On Embeddings for Numerical Features in Tabular Deep Learning".
Em uma frase: transformar os recursos escalares contínuos originais em vetores antes de misturá-los no backbone principal (por exemplo, em MLP, Transformer, etc.) melhora o desempenho downstream de redes neurais tabulares.
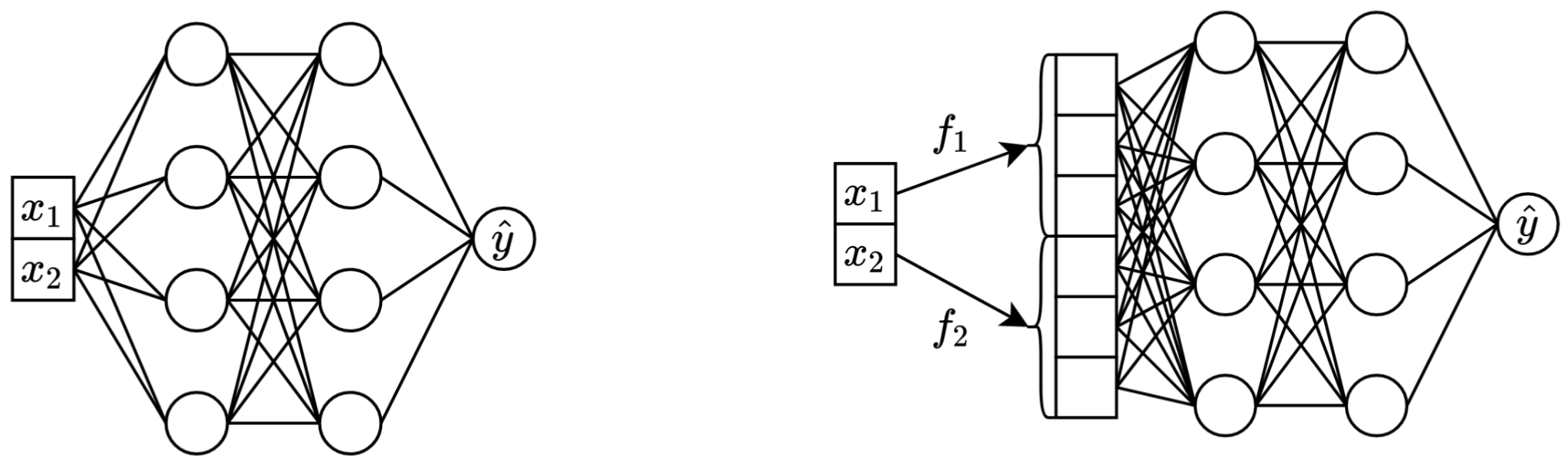
Esquerda: vanilla MLP tomando dois recursos contínuos como entrada.
À direita: o mesmo MLP, mas agora com embeddings para recursos contínuos.
Mais detalhadamente:
A rigor, não há uma explicação única. Evidentemente, os embeddings ajudam a lidar com vários desafios associados a recursos contínuos e a melhorar as propriedades gerais de otimização dos modelos.
Em particular, recursos contínuos distribuídos irregularmente (e suas distribuições conjuntas irregulares com rótulos) são comuns em dados tabulares do mundo real e representam um grande desafio de otimização fundamental para modelos DL tabulares tradicionais. Uma ótima referência para entender esse desafio (e um ótimo exemplo de como enfrentar esses desafios transformando o espaço de entrada) é o artigo "Fourier Features Let Networks Learn High Frequency Functions in Low Dimensional Domains".
No entanto, não está claro se as distribuições irregulares são a única razão pela qual os embeddings são úteis.
O pacote Python no diretório package/ é a forma recomendada de usar o artigo na prática e em trabalhos futuros.
O resto do documento :
O diretório exp/ contém vários resultados e hiperparâmetros (ajustados) para vários modelos e conjuntos de dados usados no artigo.
Por exemplo, vamos explorar as métricas do modelo MLP. Primeiro, vamos carregar os relatórios (os arquivos report.json ):
import json
from pathlib import Path
import pandas as pd
df = pd . json_normalize ([
json . loads ( x . read_text ())
for x in Path ( 'exp' ). glob ( 'mlp/*/0_evaluation/*/report.json' )
])Agora, para cada conjunto de dados, vamos calcular a média da pontuação do teste de todas as sementes aleatórias:
print ( df . groupby ( 'config.data.path' )[ 'metrics.test.score' ]. mean (). round ( 3 ))A saída corresponde exatamente à Tabela 3 do artigo:
config.data.path
data/adult 0.854
data/california -0.495
data/churn 0.856
data/covtype 0.964
data/fb-comments -5.686
data/gesture 0.632
data/higgs-small 0.720
data/house -32039.399
data/microsoft -0.747
data/otto 0.818
data/santander 0.912
Name: metrics.test.score, dtype: float64
A abordagem acima também pode ser usada para explorar hiperparâmetros para obter intuição sobre valores típicos de hiperparâmetros para diferentes algoritmos. Por exemplo, é assim que se pode calcular a taxa mediana de aprendizagem ajustada para o modelo MLP:
Observação
Para alguns algoritmos (por exemplo, MLP, MLP-LR, MLP-PLR), projetos mais recentes oferecem mais resultados que podem ser explorados de forma semelhante. Por exemplo, consulte este artigo no TabR.
Aviso
Use esta abordagem com cautela. Ao estudar valores de hiperparâmetros:
print ( df [ df [ 'config.seed' ] == 0 ][ 'config.training.lr' ]. quantile ( 0.5 ))
# Output: 0.0002716544410603358Importante
Esta seção é longa. Use o recurso "Esboço" no GitHub em seu editor de texto para obter uma visão geral desta seção.
Preliminares:
/usr/local/cuda-11.1/bin esteja sempre em sua variável de ambiente PATH export PROJECT_DIR= < ABSOLUTE path to the repository root >
# example: export PROJECT_DIR=/home/myusername/repositories/num-embeddings
git clone https://github.com/yandex-research/tabular-dl-num-embeddings $PROJECT_DIR
cd $PROJECT_DIR
conda create -n num-embeddings python=3.9.7
conda activate num-embeddings
pip install torch==1.10.1+cu111 -f https://download.pytorch.org/whl/torch_stable.html
pip install -r requirements.txt
# if the following commands do not succeed, update conda
conda env config vars set PYTHONPATH= ${PYTHONPATH} : ${PROJECT_DIR}
conda env config vars set PROJECT_DIR= ${PROJECT_DIR}
# the following command appends ":/usr/local/cuda-11.1/lib64" to LD_LIBRARY_PATH;
# if your LD_LIBRARY_PATH already contains a path to some other CUDA, then the content
# after "=" should be "<your LD_LIBRARY_PATH without your cuda path>:/usr/local/cuda-11.1/lib64"
conda env config vars set LD_LIBRARY_PATH= ${LD_LIBRARY_PATH} :/usr/local/cuda-11.1/lib64
conda env config vars set CUDA_HOME=/usr/local/cuda-11.1
conda env config vars set CUDA_ROOT=/usr/local/cuda-11.1
# (optional) get a shortcut for toggling the dark mode with cmd+y
conda install nodejs
jupyter labextension install jupyterlab-theme-toggle
conda deactivate
conda activate num-embeddingsLICENÇA: ao baixar nosso conjunto de dados você aceita as licenças de todos os seus componentes. Não impomos quaisquer novas restrições além dessas licenças. Você pode encontrar a lista de fontes no jornal.
cd $PROJECT_DIR
wget " https://www.dropbox.com/s/r0ef3ij3wl049gl/data.tar?dl=1 " -O num_embeddings_data.tar
tar -xvf num_embeddings_data.tarO código abaixo reproduz os resultados do MLP no conjunto de dados California Housing. O pipeline para outros algoritmos e conjuntos de dados é absolutamente o mesmo.
# You must explicitly set CUDA_VISIBLE_DEVICES if you want to use GPU
export CUDA_VISIBLE_DEVICES="0"
# Create a copy of the 'official' config
cp exp/mlp/california/0_tuning.toml exp/mlp/california/1_tuning.toml
# Run tuning (on GPU, it takes ~30-60min)
python bin/tune.py exp/mlp/california/1_tuning.toml
# Evaluate single models with 15 different random seeds
python bin/evaluate.py exp/mlp/california/1_tuning 15
# Evaluate ensembles (by default, three ensembles of size five each)
python bin/ensemble.py exp/mlp/california/1_evaluation
A seção “Métricas” mostra como resumir os resultados obtidos.
O código está organizado da seguinte forma:
bintrain4.py para redes neurais (ele implementa todos os embeddings e backbones do papel)xgboost_.py para XGBoostcatboost_.py para CatBoosttune.py para ajusteevaluate.py para avaliaçãoensemble.py para montagemdatasets.py foi usado para construir as divisões do conjunto de dadossynthetic.py para gerar conjuntos de dados sintéticos compatíveis com GBDTtrain1_synthetic.py para os experimentos com dados sintéticoslib contém ferramentas comuns usadas por programas no binexp contém configurações e resultados de experimentos (métricas, configurações ajustadas, etc.). Os nomes das pastas aninhadas seguem os nomes do artigo (exemplo: exp/mlp-plr corresponde ao modelo MLP-PLR do artigo).package contém o pacote Python para este artigoCUDA_VISIBLE_DEVICES ao executar scriptslib.dump_config e lib.load_config em vez de bibliotecas TOML simplesO padrão comum para execução de scripts é:
python bin/my_script.py a/b/c.toml onde a/b/c.toml é o arquivo de configuração de entrada (config). A saída estará localizada em a/b/c . A estrutura de configuração geralmente segue a classe Config de bin/my_script.py .
Existem também scripts que usam argumentos de linha de comando em vez de configurações (por exemplo, bin/{evaluate.py,ensemble.py} ).
Você precisa de todos eles para reproduzir resultados, mas precisa apenas de train4.py para trabalhos futuros, porque:
bin/train1.py implementa um superconjunto de recursos de bin/train0.pybin/train3.py implementa um superconjunto de recursos de bin/train1.pybin/train4.py implementa um superconjunto de recursos de bin/train3.py Para ver qual dos quatro scripts foi usado para executar um determinado experimento, verifique o campo "programa" da configuração de ajuste correspondente. Por exemplo, aqui está a configuração de ajuste para MLP no conjunto de dados California Housing: exp/mlp/california/0_tuning.toml . A configuração indica que bin/train0.py foi usado. Isso significa que as configurações em exp/mlp/california/0_evaluation são compatíveis especificamente com bin/train0.py . Para verificar isso, você pode copiar um deles para um local separado e passar para bin/train0.py :
mkdir exp/tmp
cp exp/mlp/california/0_evaluation/0.toml exp/tmp/0.toml
python bin/train0.py exp/tmp/0.toml
ls exp/tmp/0
@inproceedings{gorishniy2022embeddings,
title={On Embeddings for Numerical Features in Tabular Deep Learning},
author={Yury Gorishniy and Ivan Rubachev and Artem Babenko},
booktitle={{NeurIPS}},
year={2022},
}