
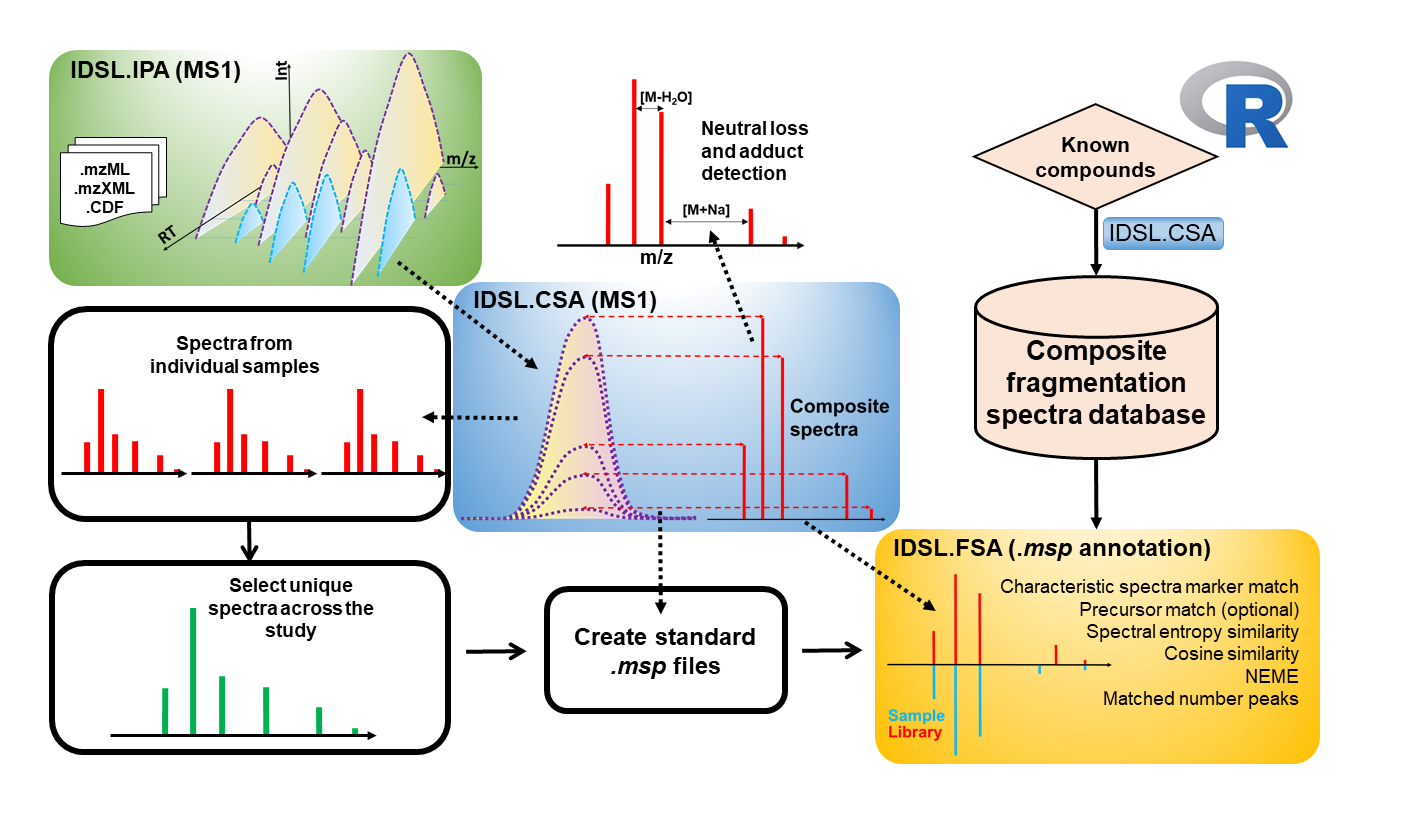
O pacote de Análise de Espectros Compostos (IDSL.CSA) para a análise de dados de espectrometria de massa foi desenvolvida pelo Laboratório de Ciência de Dados Integrado para Metabolômica e Exposomia (IDSL.ME) . Este pacote pode ser usado para a desconvolução dos espectros de fragmentação obtidos através de vários métodos analíticos, como Análise de Deconvolução de Espectros de Compostos somente para MS1 ( CSA ), aquisição dependente de dados ( DDA ) e vários métodos de aquisição independente de dados ( DIA ), incluindo MS e , Fragmentação de íons All-Ion (AIF) e análises SWATH-MS. O objetivo do pacote IDSL.CSA é ajudar a simplificar o processo de análise de dados e melhorar a anotação geral da estrutura química nos campos da metabolômica e exposômica.
Start na planilha de parâmetros IDSL.CSA. install.packages("IDSL.CSA")
Antes de processar seus dados de espectrometria de massa ( MZXML , MZML , NETCDF ) usando o fluxo de trabalho IDSL.CSA, os dados de espectrometria de massa devem ser processados usando o fluxo de trabalho IDSL.IPA para adquirir informações cromatográficas dos picos ( M/Z-RT ). Quando as informações cromatográficas de listas de picos alinhadas individuais e agregadas foram geradas usando o fluxo de trabalho IDSL.IPA, faça o download da planilha de parâmetros IDSL.CSA e selecione os parâmetros de acordo e use esta planilha como entrada para o fluxo de trabalho IDSL.CSA:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Siga estas etapas para um estudo de caso rápido (n = 33) ST002263, que possui dados de orbitrap de hibrides de HF exatos q coletados nos modos hilic-esi-pos/neg.
Processar dados de espectrometria de massa bruta e informações cromatográficas usando o método descrito para idsl.ipa
A análise de espectros compostos requer 39 parâmetros distribuídos em 5 seções separadas para uma análise em escala completa. Para este estudo, use os valores de parâmetros padrão apresentados na planilha de parâmetros IDSL.CSA. Em seguida, forneça informações para
2.1. Selecione Sim para o Param0001 na guia Start para processar apenas o fluxo de trabalho da CSA .
2.2. CSA0005 para o endereço de localização de dados do HRMS (Dados do HRMS de nível MS1)
2.3. CSA0008 para o endereço do diretório peaklists gerado pelo IDSL.ipa Workflow
2.4. CSA0009 para o endereço do diretório peak_alignment gerado pelo IDSL.ipa Workflow
2.5. CSA0011 para localização da saída (arquivos .msp e EICs)
2.6. Você também pode aumentar o número de threads de processamento usando CSA0004 de acordo com o seu poder computacional
Execute este comando no console ou terminal rstudio r/r/
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Você pode analisar os resultados no endereço que você forneceu para o CSA0011 .
4.1. Csa_msp inclui arquivo .msp
4.2. CSA_ADDUCT_ANNOTATION INCLUI PAIXAS COM POTUÇÃO INFORMAÇÕES DE ADUTUCA
4.3. Peak_alignment_subset inclui subconjuntos de tabelas de pico alinhadas para os principais íons em cada cluster CSA
4.4. alinhed_specectra_table inclui informações para a agregação de CSA na tabela alinhada
[1] FAKOURI BAYGI, S., KUMAR, Y. BARUPAL, DK IDSL.CSA: Análise de espectros compostos para anotação química de conjuntos de dados de metabolômica não direcionados. Analytical Chemistry , 2023 , 95 (25) , 9480-9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, DK IDSL. O IPA caracteriza o espaço químico orgânico em conjuntos de dados não direcionados de LC/HRMS. Journal of Proteome Research , 2022 , 21 (6) , 1485-1494.