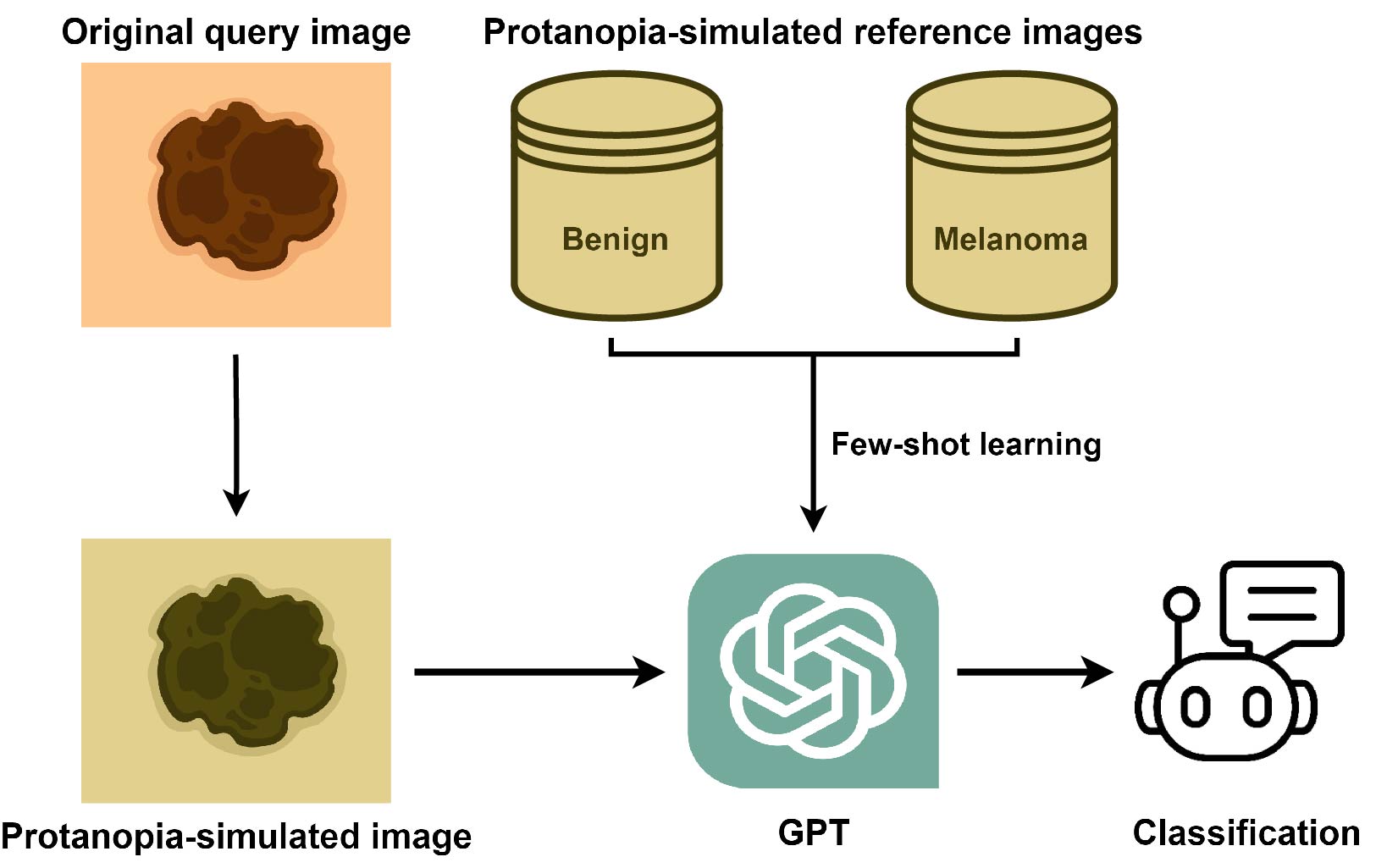
Este repositório contém código e dados para executar a classificação dermovoscópica com deficiência de deficiência de cor com o GPT-4V.
Leia o artigo
Encontre a resposta detalhada do modelo GPT Form Mendeley Data
Numpy
Pil
Daltonlens
JSON
Openai
base64
Pandas
. ├── data # Contains subdirectories for processed data │ ├── all │ ├── all_resized │ ├── all_resized_brettel_protan_1 │ ├── all_resized_brettel_deutan_1 │ ├── all_resized_brettel_tritan_1 │ ├── bn │ ├── bn_resized │ ├── bn_resized_label │ ├── bn_resized_label_brettel_protan_1 │ ├── bn_resized_label_brettel_deutan_1 │ ├── bn_resized_label_brettel_tritan_1 │ ├── mm │ ├── mm_resized │ ├── mm_resized_label │ ├── mm_resized_label_brettel_protan_1 │ ├── mm_resized_label_brettel_deutan_1 │ ├── mm_resized_label_brettel_tritan_1 │ └── selected_images.xlsx # Image names ├── RAW # Contains raw data downloaded from ISCI Archive ├── result # Results from running API_calling will be stored here │ ├── 2_shot_brettel_protan_1 │ │ ├──rep 1 │ │ └── ... │ ├── 2_shot_brettel_deutan_1 │ └── ... ├── CVD_classification_GPT.py # Call OpenAI API for classification ├── CVD_convertor.py # Convert original image to CVD simulated image ├── data_labeling.py # Add label to the image for reference ├── data_resizing.py # Resize the data with the original aspect ratio ├── data_selection.py # Select data from RAW └── README.md
Todas as imagens dermoscópicas são baixadas do Arquivo ISIC.
Este projeto é apenas para fins de pesquisa acadêmica. O código deste repositório é liberado sob a licença do MIT. Se você usar os dados fornecidos, cite o arquivo ISIC.S
Para simulação CVD, escolhemos o pacote Daltonlens-Python. Ele abordou uma variedade de algoritmos atualmente disponíveis da simulação de daltonismo. O blog do autor também vale a pena ler.
Selecionamos o Brettel et al. 1997 como o método de simulação CVD e defina a gravidade em 1, que pode ser ajustada com o código fornecido conforme necessário.
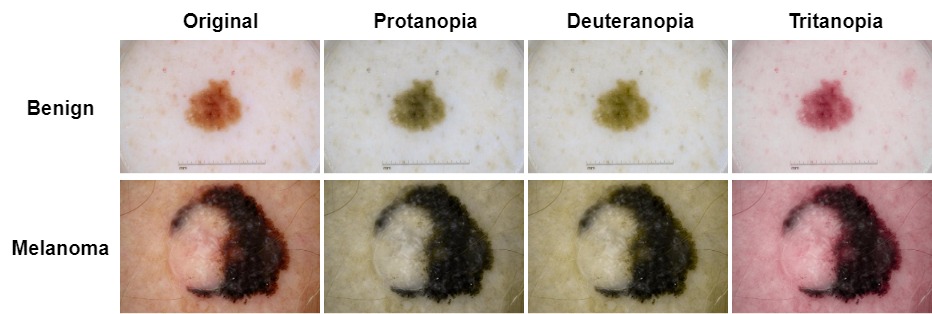
Exemplo benigno: ISIC_0012656
Exemplo de melanoma: ISIC_0046725
Baixe os dados brutos do Arquivo ISIC
Selecione dados:
Run Python data_selection.py -[Opções]
Dados do processo:
Run Python Data_Resizeing.py -[Opções]
Execute o Python data_labeling.py -[Opções]
Converter dados:
Run Python cvd_convertor.py -[Opções]
Ligue para a API:
Run Python CVD_Classification_GPT.Py -[Opções]
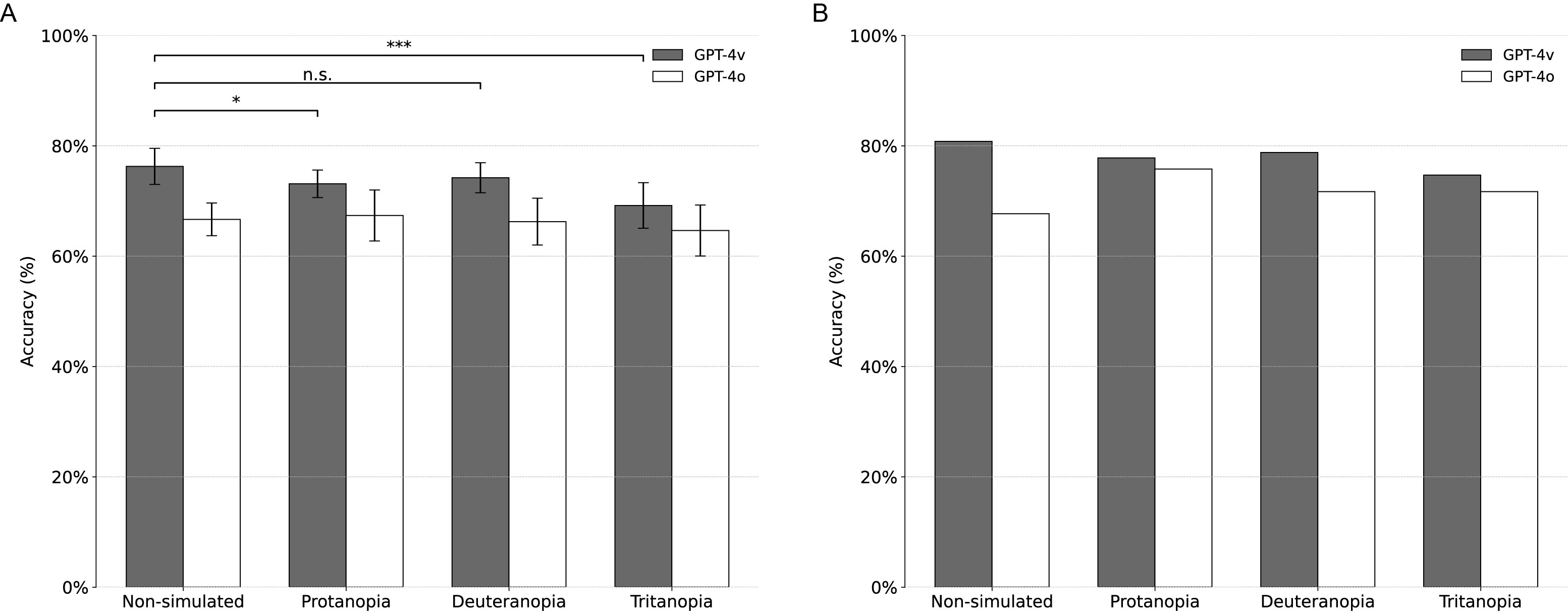
(A) Precursistas médias de classificação de GPT-4V para imagens não simuladas e simuladas por CVD (protanopia, deuteranopia, tritanopia). Barras de erro: desvios padrão. *: p <0,05; **: p <0,01 (teste t; duas caudas). NS: Não é significativo. Todas as experiências foram em dez repetições. (B) Precisão de classificação do GPT-4V após a aplicação da estratégia de consenso nas dez repetições para cada consulta de imagem. GPT-4O incluído para comparação.
O GPT-4V adaptou sua interpretação sobre as descobertas baseadas em cores para diferentes simulações de CVD. Especificamente, não mencionou cores vermelha ou rosa nas simulações de protanope e deuteranope, mas associou essas cores a uma previsão de melanoma em imagens não simuladas. Nas simulações de tritanopia-caracterizadas por tons rosados predominantes-o GPT-4V não é mais tratado rosa como uma característica de melanoma.
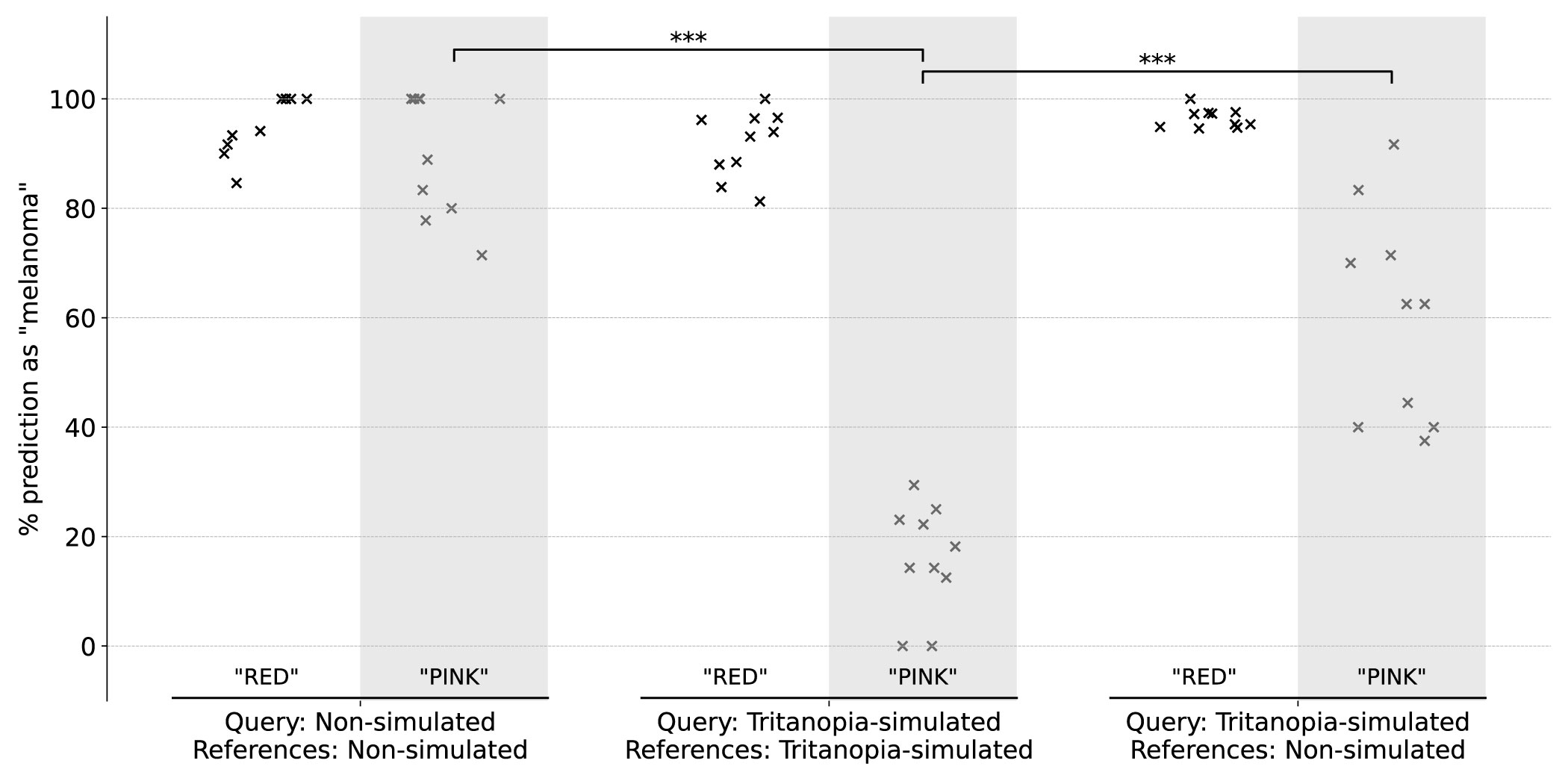
Para cada uma das condições não simuladas e simuladas de tritanopia, dois grupos de consultas de imagem foram identificados com base nas explicações do GPT-4V para suas previsões. O primeiro grupo, rotulado como “vermelho”, inclui descrições em que a cor vermelha é mencionada para descrever a imagem de consulta, independentemente da cor rosa. O segundo grupo, rotulado como "rosa", compreende descrições mencionando exclusivamente rosa sem vermelho. O eixo y exibe a porcentagem de previsões de "melanoma" de cada replicação. Por padrão, as imagens de consulta e referência não foram simuladas ou simuladas para a mesma condição de CVD. Um teste de ablação (as duas últimas colunas) foi realizado para a simulação de tritanopia, amostrando as referências de imagens não simuladas. ***: p <0,001 (teste t; duas caudas).