Temos o prazer de anunciar que o tamanho do índice no disco caiu 8 vezes e na memória 4 vezes devido à mudança para apenas um tipo de FM-Index (2bit.64 em vez de 2bit.64 e 8bit.32) e 8x compressão de matriz de sufixo. Por exemplo, para o genoma humano, o tamanho do índice no disco caiu para ~ 10 GB de ~ 80 GB e a pegada de memória cai para ~ 10 GB de ~ 40 GB. Há uma redução substancial no tempo de IO do índice devido à redução e quase nenhum impacto de desempenho no mapeamento de leitura. Devido a essa mudança na estrutura do índice (no comprometimento nº 4B59796, 10 de outubro de 2020), você precisará reconstruir o índice.
Adicionado sinalizador MC no arquivo SAM de saída no Commit A591E22. A saída deve corresponder à versão original do BWA-MEM 0.7.17.
Até o comando E0AC59E, temos um submódulo Git SalfestringLib. Para obtê -lo, use -Recursivo durante a clonagem ou use "Git Submodule Init" e "Git Submodule Atualize" em um repositório já clonado (veja abaixo para obter mais detalhes).
# Use binários pré-compilados (recomendados) Curl -l https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.2.1/bwa-mem2-2.2.1_x64-linux.tar.bz2 | Tar JXF - BWA-MEM2-2.2.1_X64-Linux/BWA-MEM2 ÍNDICE REF.FA bwa-mem2-2.2.1_x64-linux/bwa-mem2 mem ref.fa read1.fq read2.fq> out.sam# compilar da fonte (não recomendado para usuários em geral)# Get the Sourcegit clone-Recursive https: // github.com/bwa-mem2/bwa-mem2cd bwa-mem2# clone orgit https://github.com/bwa-mem2/bwa-mem2cd bwa-mem2 Git submodule init Git Submodule Atualização# Compile e Runmake ./bwa-mem2
A ferramenta BWA-MEM2 é a próxima versão do algoritmo BWA-MEM no BWA. Produz alinhamento idêntico ao BWA e é ~ 1,3-3.1x mais rápido, dependendo do caixa de uso, do conjunto de dados e da máquina em execução.
O BWA original foi desenvolvido por Heng Li (@LH3). O aprimoramento do desempenho no BWA-MEM2 foi realizado principalmente pelo Vasimuddin MD (@yuk12) e Sanchit misra (@sanchit-misra) do Laboratório de Computação Paralelo, Intel. O BWA-MEM2 é distribuído sob a licença do MIT.
Para usuários em geral, é recomendável usar os binários pré -compilados na página de liberação. Esses binários foram compilados com o compilador Intel e correm mais rápido que os binários compilados por GCC. Os binários pré -compilados também apoiam indiretamente a expedição da CPU. O binário bwa-mem2 pode escolher automaticamente a implementação mais eficiente com base no conjunto de instruções SIMD disponível na máquina em execução. Os binários pré -compilados foram gerados em uma máquina CentOS7 usando a seguinte linha de comando:
Faça CXX = ICPC multi
O uso é exatamente o mesmo que a ferramenta original do BWA MEM. Aqui está uma breve sinopse. Run ./bwa-mem2 para comandos disponíveis.
# Indexação da sequência de referência (requer memória de 28N GB em que n é o tamanho da sequência de referência) ../ Índice BWA-MEM2 [-P Prefixo] <in.fastA> Onde <in.fastA> é o caminho para referência ao arquivo de sequência fastha e <fix> é o prefixo dos nomes dos arquivos que armazenam o índice resultante. O padrão está in.FASTA. # Mapping # run "./bwa-mem2 Mem" para obter todas as opções./bwa-mem2 Mem -t <Mum_Threads> <Prefix> <reads.fq/fa>> out.sam Onde <fix> é o prefixo especificado ao criar o índice ou o caminho para o arquivo fasta de referência, caso nenhum prefixo tenha sido fornecido.
Conjuntos de dados:
Genoma de referência: Human_G1K_V37.FASTA
| Alias | Fonte do conjunto de dados | No. de leituras | Leia o comprimento |
|---|---|---|---|
| D1 | Broad Institute | 2 x 2,5m bp | 151BP |
| D2 | SRA: SRR7733443 | 2 x 2,5m bp | 151BP |
| D3 | SRA: SRR9932168 | 2 x 2,5m bp | 151BP |
| D4 | SRA: SRX6999918 | 2 x 2,5m bp | 151BP |
Detalhes da máquina:
Processador: Intel (R) Xeon (R) 8280 CPU @ 2.70GHz
OS: CentOS Linux Release 7.6.1810
Memória: 100 GB
Seguimos as etapas abaixo para coletar os resultados do desempenho:
A. Etapas de download de dados:
Faça o download do SRA Toolkit de https://trace.ncbi.nlm.nih.gov/traces/sra/sra.cgi?view=software#header-global
TAR XFZV SRATOOLKIT.2.10.5-CENTOS_LINUX64.TAR.GZ
Download D2: Sratoolkit.2.10.5-CENTOS_LINUX64/BIN/FASTQ-DUMP-SPLIT-FILS SRR77733443
Download D3: Sratoolkit.2.10.5-CENTOS_LINUX64/BIN/FASTQ-DUMP-SPLIT-FILES SRR9932168
Download D4: Sratoolkit.2.10.5-CENTOS_LINUX64/BIN/FASTQ-DUMP-SPLIT-FILES SRX6999918
B. Etapas de alinhamento:
Git clone https://github.com/bwa-mem2/bwa-mem2.git
CD BWA-MEM2
make CXX=icpc (usando o Intel C/C ++ Compiler)
ou make (usando o compilador GCC)
./bwa-mem2 Índice <ref.fa>
./bwa-mem2 mem [-t <#threads>] <ref.fa> <in_1.fastq> [<In_2.fastq>]> <output.sam>
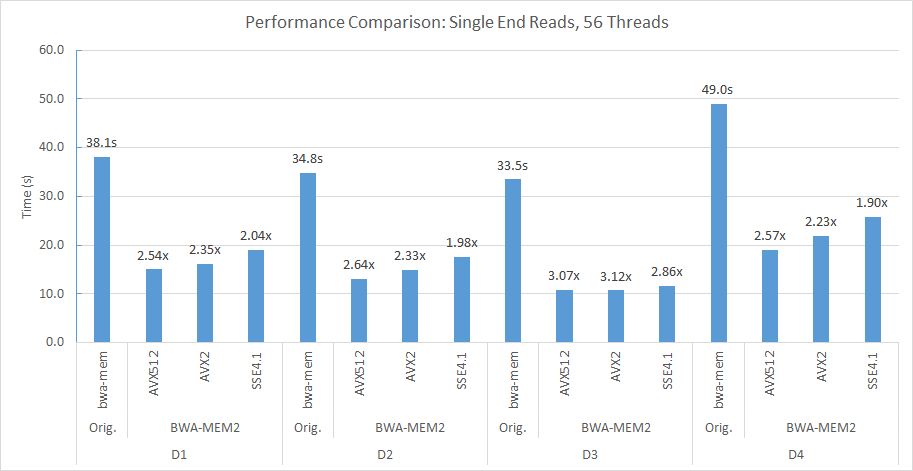
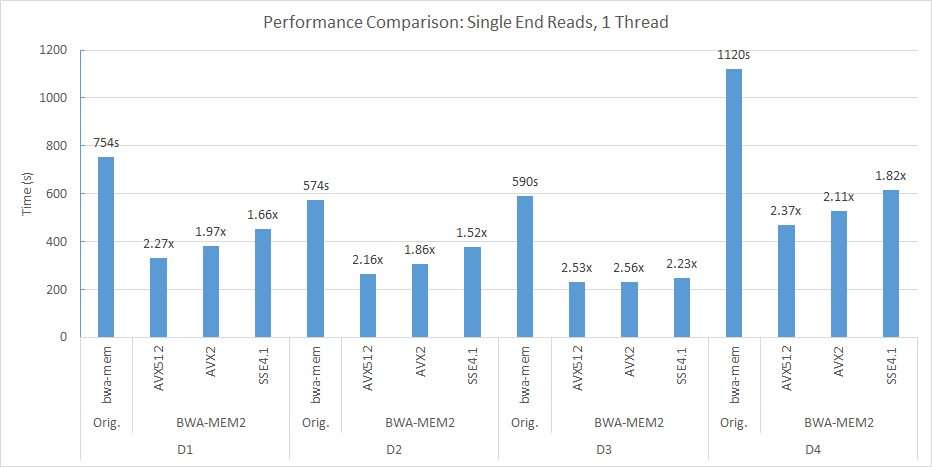
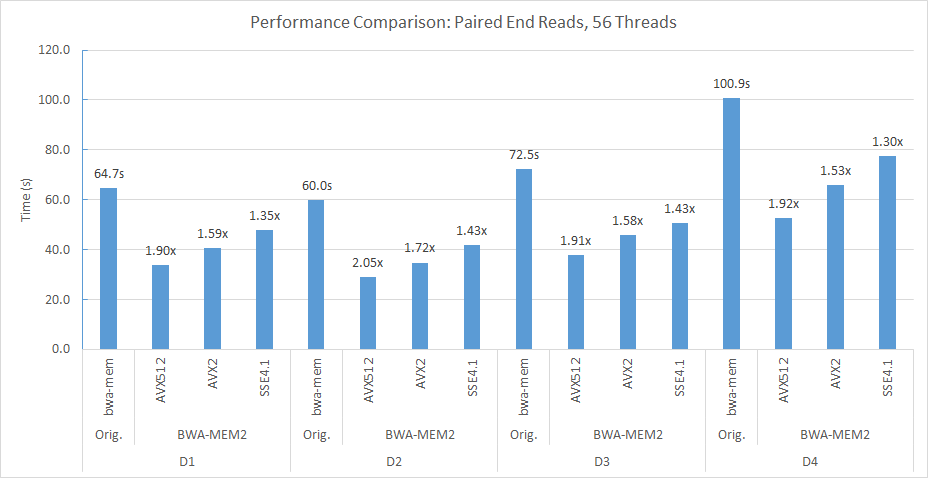
Por exemplo, em nosso soquete duplo (56 threads cada) e o nó de computação de NUMA duplo, usamos a seguinte linha de comando para alinhar D2 ao genoma de referência Human_G1K_V37.FASTA.
numactl -m 0 -C 0-27,56-83 ./bwa-mem2 index human_g1k_v37.fasta numactl -m 0 -C 0-27,56-83 ./bwa-mem2 mem -t 56 human_g1k_v37.fasta SRR7733443_1.fastq SRR7733443_2.fastq > d2_align.sam

O BWA-MEM2-LISA é uma versão acelerada do BWA-MEM2, onde aplicamos os índices instruídos à fase de semeadura. A filial BWA-MEM2-LISA contém o código-fonte da implementação. A seguir estão os recursos do BWA-MEM2-LISA:
Exatamente a mesma saída que BWA-MEM2.
Todas as linhas de comando para criar um índice e o mapeamento de leitura são exatamente iguais ao BWA-MEM2.
O BWA-MEM2-LISA acelera a fase de semeadura (um dos principais gargalos no BWA-MEM2) em até 4,5x em comparação com o BWA-MEM2.
A pegada de memória do índice BWA-MEM2-LISA é de ~ 120 GB para o genoma humano.
O código está presente na filial bwa-mem2-lisa: https://github.com/bwa-mem2/bwa-mem2/tree/bwa-mem2-lisa
A filial ERT do repositório BWA-MEM2 contém a base de código da aceleração baseada na árvore da radix enuerada de BWA-MEM2. O código ERT é construído na parte superior do BWA-MEM2 (graças ao trabalho árduo de @arun-sub). A seguir, são apresentados os destaques da ferramenta BWA-MEM2 baseada em ERT:
Exatamente a mesma saída que o BWA-MEM (2)
A ferramenta possui dois sinalizadores adicionais para permitir o uso da solução ERT (para criação e mapeamento de índices), caso contrário, é executado no modo baunilha BWA-MEM2
Ele usa 1 sinalizador adicional para criar índice ERT (diferente do índice BWA-MEM2) e 1 sinalizador adicional para usar esse índice ERT (consulte o ReadMe of Ert Branch)
A solução ERT é 10% -30% mais rápida (testada na configuração da máquina acima) em comparação com a baunilha BWA -MEM2 -os usuários são aconselhados a usar a opção -K 1000000 para ver as acelerações
A impressão do pé da memória do índice ERT é ~ 60 GB
O código está presente no ramo do ERT: https://github.com/bwa-mem2/bwa-mem2/tree/ert
Vasimuddin MD, Sanchit Misra, Heng Li, Srinivas Aluru. Aceleração eficiente de arquitetura de arquitetura do BWA-MEM para sistemas multicore. Simpósio de processamento paralelo e distribuído do IEEE (IPDPS), 2019. 10.1109/ipdps.2019.00041