
Isling é uma ferramenta para detectar a integração viral ou vetorial em leituras de extremidade pareada. Por favor, leia nosso artigo para obter detalhes completos.
Se você tem conda e snakemake , para executar com os dados de teste (incluídos) localmente:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
Se você tem snakemake e singularity instalados, pode usar:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
Como alternativa, se você instalou o Docker, no macOS, você pode executar:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
Isso usará o arquivo de configuração e os dados dentro do contêiner, e os resultados aparecerão em uma pasta chamada out seu diretório de trabalho atual. No Linux, você precisará executar esse comando como root e, no Windows, precisará ajustar a sintaxe Bind -Mount ( -v de argumento).
Os dados de entrada (leituras e referências de host e viral) são especificados ni um arquivo de configuração - para seus próprios dados, você precisará modificar o arquivo de configuração de exemplo ( test/config/test.yml ) para apontar para seus próprios dados. Consulte configfile.md para obter mais informações sobre o formato do arquivo de configuração.
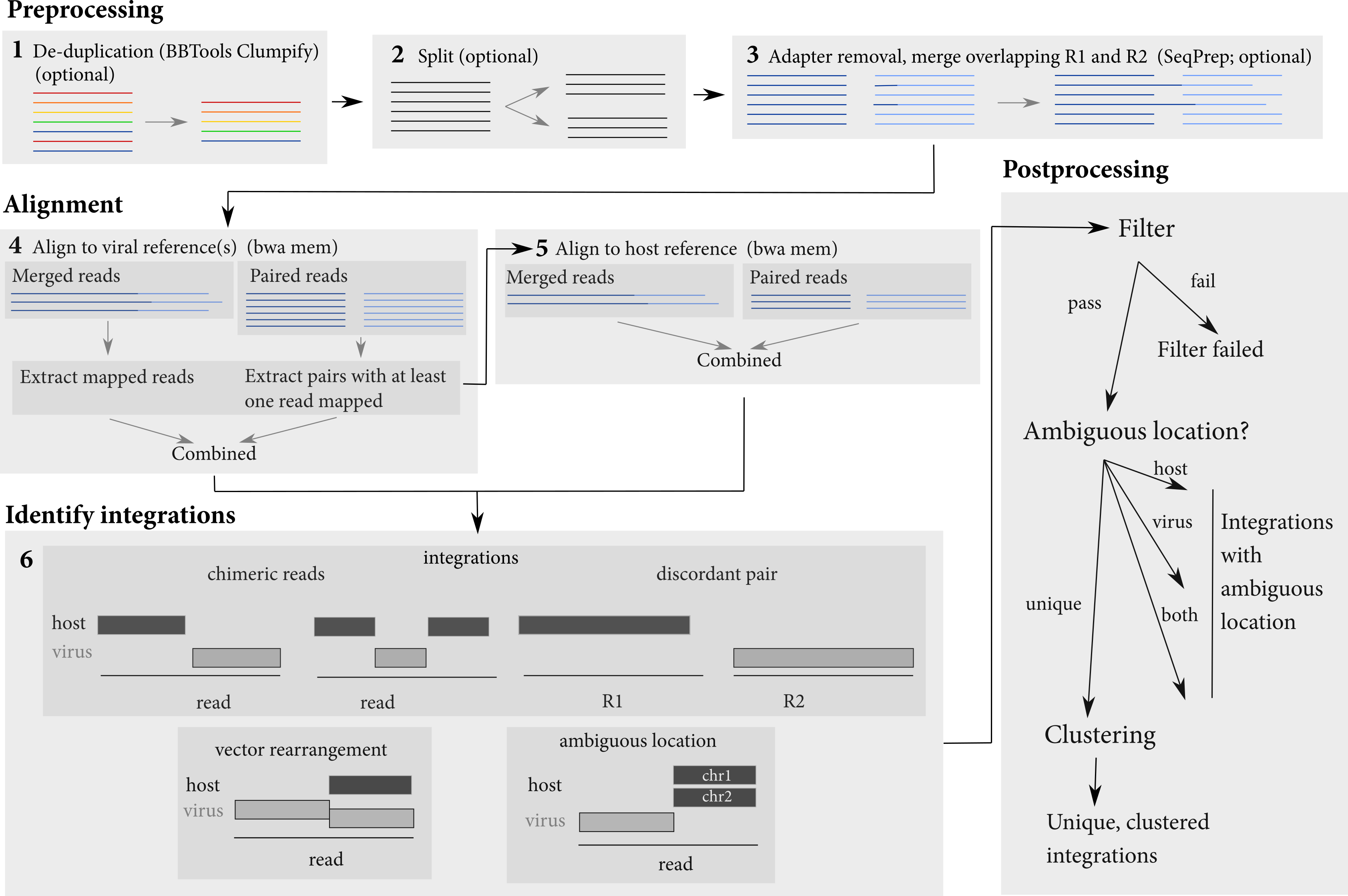
O pipeline executa várias etapas para identificar sites de integração. São necessários conjuntos de dados de entrada que consistem em arquivos FastQ ou BAM. Ele faz algum pré-processamento das leituras (mesclando leituras sobrepostas, opcional) e as alinham a um host e uma sequência viral. As leituras são alinhadas primeiro às seqüências virais e, em seguida, as leituras alinhadas são extraídas e alinhadas ao host. Esses alinhamentos são usados para identificar integrações virais.
Isling requer snakemake e singularity (recomendado) ou conda para fornecer dependências. São necessários ou acima do Python, python da versão 3.5 ou acima, pandas devem ser instalados automaticamente se a instalação snakemake com conda .
Alternativley, use a versão do Docker, que contém isling e todas as dependências.
As entradas necessárias são o arquivo de configuração, que especifica o host e as referências virais/vetoriais, e são necessárias leituras. Especifique todas as entradas em um arquivo de configuração. Atualmente, Isling funciona apenas para leituras de extremidade pareada.
Consulte o arquivo configfile.md para obter uma descrição do formato deste arquivo de configuração.
Isling Saídas Sites de integração em um formato separado por TAB no diretório de saída especificado no arquivo de configuração.
Na pasta de saída, uma pasta é criada para cada conjunto de dados no arquivo de configuração e, para cada conjunto de dados, as integrações podem ser encontradas no diretório ints . Haverá um conjunto de arquivos de saída para cada amostra.
Para cada amostra, existem vários arquivos de saída, que podem ser interessantes para casos de uso específicos.
<sample>.<host>.<virus>.integrations.txt : Informações sobre todas<sample>.<host>.<virus>.integrations.post.txt : Informações sobre junções detectadas que passaram todos os filtros<sample>.<host>.<virus>.integrations.post.unique.txt : informações sobre junções detectadas que passaram todos os filtros e têm uma localização inequívoca no host e no vetor/vírus<sample>.<host>.<virus>.integrations.post.unique.merged.txt : o resultado da mesclagem de junções ou junções sobrepostas com as mesmas coordenadas no host e no vírus/vetor. A mesclagem é realizada apenas com as junções de integração que têm uma localização inequívoca no host e no vírus/vetor.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : informações sobre junções detectadas que passaram todos os filtros e têm um vetor/vírus de localização inequívoca, mas localização ambígua no host<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : informações sobre junções detectadas que passaram todos os filtros e têm um hospedeiro de localização inequívoco, mas localização ambígua no vetor/vírus<sample>.<host>.<virus>.integrations.post.both_ambig.txt : informações sobre junções detectadas que passaram todos os filtros e têm uma localização ambígua no host e no vetor/vírus Os arquivos de saída fornecem a localização das integrações identificadas e suas propriedades. As coordenadas para junções de integração são especificadas em termos de suas bases ambíguas. Ou seja, muitas vezes há uma lacuna ou sobreposição entre o host e as partes virais de uma leitura: 
Como a localização da integração não pode ser determinada exclusivamente neste caso, o isling produz as coordenadas dessas bases no genoma do host e do vetor/vírus como a localização da integração.
Com exceção do arquivo de saída de cluster mesclado, todos os arquivos contêm estas colunas:
gap ou overlap para uma leitura quimierica (host e peças virais identificadas na mesma leitura), uma junção é clean se não tiver bases ambíguas. Como alternativa, se a jucntion foi identificada em um par discordante, o OverlapType é discordanthv ) ou vírus/host ( vh )+ ou -bwa ) do alinhamento do hostbwa ) do Alinhamento ViralOverlapType for gapchimeric , um par discordant ou é uma short integração na qual ambas as junções foram observadas na mesma leiturabwa )bwa )'xxx' Para reproduzir as figuras no manuscrito isling, consulte o ReadMe no diretório benchmarking .