Papel: https://arxiv.org/abs/2310.00149
Autores: Hao Liu, Jiarui Feng, Lecheng Kong, Ningyue Liang, Dacheng Tao, Yixin Chen, Muhan Zhang
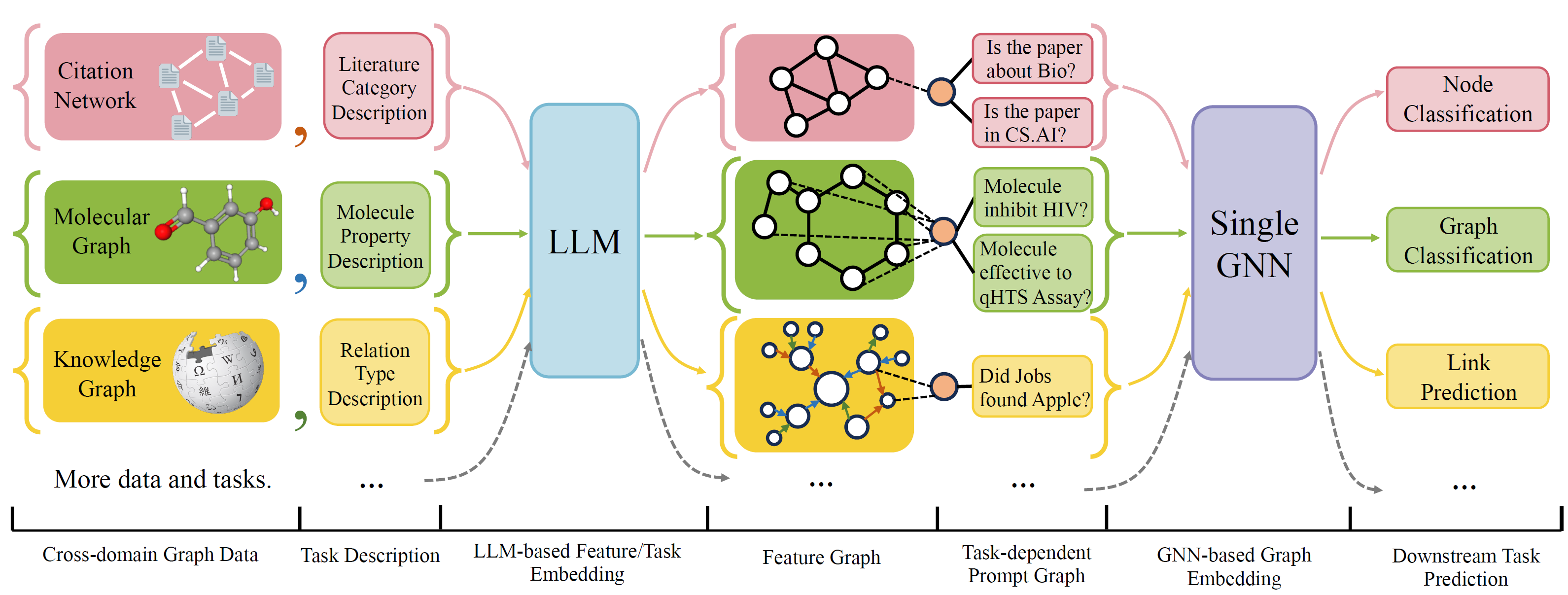
OFA é uma estrutura geral de classificação de gráficos que pode resolver uma ampla gama de tarefas de classificação de gráficos com um único modelo e um único conjunto de parâmetros. As tarefas são domínios cruzados (por exemplo, rede de citação, gráfico molecular, ...) e tarefas cruzadas (por exemplo, poucos tiro, zero tiro, nível de gráfico, nó, ...)
Ofa usa linguagens naturais para descrever todos os gráficos e usar um LLM para incorporar toda a descrição no mesmo espaço de incorporação, que permite o treinamento de domínio cruzado usando um único modelo.
Ofa propõe um paradiagm de que todas as informações da tarefa são convertidas para solicitar gráficos. Portanto, o modelo subsequente é capaz de ler as informações das tarefas e prever o alvo Rellavent de acordo, sem precisar ajustar os parâmetros e a arquitetura do modelo. Portanto, um único modelo pode ser uma tarefa cruzada.
OFA CURADO Uma lista de conjuntos de dados de gráficos de fontes e domínios diferentes e descreva nós/arestas nos gráficos com um protocolo de descritas sistemáticas. Agradecemos a trabalhos anteriores, incluindo OGB, GIMLET, MoleculeNet, Graphllm e Villmow por fornecer maravilhosos dados de gráfico bruto/texto que possibilitem nosso trabalho.
O One foi submetido a uma grande revisão, onde limpamos o código e corrigimos vários bugs relatados. As principais atualizações são:
Se você usou anteriormente nosso repositório, puxe e exclua os antigos arquivos de recurso/texto gerados e regenere. Desculpe pela inconveniência.
Para instalar o requisito para o projeto usando o CONDA:
conda env create -f environment.yml
Para experimentos conjuntos de ponta a ponta em todos os conjuntos de dados coletados, execute
python run_cdm.py --override e2e_all_config.yaml
Todos os argumentos podem ser alterados por valores separados pelo espaço, como
python run_cdm.py --override e2e_all_config.yaml num_layers 7 batch_size 512 dropout 0.15 JK none
Os usuários podem modificar a variável task_names em ./e2e_all_config.yaml para controlar quais conjuntos de dados estão incluídos durante o treinamento. O comprimento de task_names , d_multiple e d_min_ratio devem ser os mesmos. Eles também podem ser especificados nos argumentos da linha de comando por valores separados por vírgula.
por exemplo
python run_cdm.py task_names cora_link,arxiv d_multiple 1,1 d_min_ratio 1,1
OFA-IND pode ser especificado por
python run_cdm.py task_names cora_link d_multiple 1 d_min_ratio 1
Para executar os experimentos de poucos e zero tiro
python run_cdm.py --override lr_all_config.yaml
Definimos configurações para cada tarefa, cada configuração de tarefa contém várias configurações de conjuntos de dados.
As configurações de tarefas são armazenadas em ./configs/task_config.yaml . Uma tarefa geralmente consiste em várias divisões de conjuntos de dados (não necessariamente os mesmos conjuntos de dados). Por exemplo, uma tarefa regular de classificação de nó Cora de ponta a ponta fará com que a divisão de trem do conjunto de dados CORA como o conjunto de dados de trem, a divisão válida do conjunto de dados CORA como um dos conjuntos de dados válidos e da mesma forma para a divisão de teste. Você também pode ter mais validação/teste especificando a divisão do trem da CORA como um dos conjuntos de dados de validação/teste. Especificamente, uma configuração de tarefa se parece
arxiv :
eval_pool_mode : mean
dataset : arxiv # dataset name
eval_set_constructs :
- stage : train # a task should have one and only one train stage dataset
split_name : train
- stage : valid
split_name : valid
dataset : cora # replace the default dataset for zero-shot tasks
- stage : valid
split_name : valid
- stage : test
split_name : test
- stage : test
split_name : train # test the train split As configurações do conjunto de dados são armazenadas em ./configs/task_config.yaml . Uma configuração de conjunto de dados define como um conjunto de dados é construído. Especificamente,
arxiv :
task_level : e2e_node
preprocess : null # name of the preprocess function defined in task_constructor.py
construct : ConstructNodeCls # name of the dataset construction function defined in task_constructor.py
args : # additional arguments to construct function
walk_length : null
single_prompt_edge : True
eval_metric : acc # evaluation metric
eval_func : classification_func # evaluation function that process model output and batch to input to evaluator
eval_mode : max # evaluation mode (min/max)
dataset_name : arxiv # name of the OFAPygDataset
dataset_splitter : ArxivSplitter # splitting function defined in task_constructor.py
process_label_func : process_pth_label # name of process label function that transform original label to the binary labels
num_classes : 40 Se você estiver implementando um conjunto de dados como CORA/PubMed/Arxiv, recomendamos adicionar um diretório de seus dados $ personalizada_data $ sob dados/single_graph/$ personalizou_data $ e implementar gen_data.py no diretório, você pode usar dados/cora/gen_data. Py como exemplo.
Após a construção dos dados, você precisa registrar seu nome de conjunto de dados aqui e implementar um divisor como aqui. Se você estiver executando tarefas de tiro zero/poucos anos, poderá construir uma divisão Zero-Shot/Few-Shot aqui também.
Por fim, registre uma entrada de configuração em configs/data_config.yaml. Por exemplo, para classificação de nó de ponta a ponta
$data_name$ :
<< : *E2E-node
dataset_name : $data_name$
dataset_splitter : $splitter$
process_label_func : ... # usually processs_pth_label should work
num_classes : $number of classes$Process_label_func converte o rótulo de destino no rótulo binário e transforma a incorporação da classe Se a tarefa for zero/pouca tiro, onde o número de nó de classe não for corrigido. Uma lista de processos favoráveis ao Process_label_func está aqui. Ele assume todas as classes incorporando e o rótulo correto. A saída é uma tupla: (etiqueta, class_node_embedding, etiqueta binária/única).
Se você deseja mais flexibilidade, adicionar conjuntos de dados personalizados requer a implementação de uma subclasse personalizada de OfApyGDataset. Um modelo está aqui:
class CustomizedOFADataset ( OFAPygDataset ):
def gen_data ( self ):
"""
Returns a tuple of the following format
(data, text, extra)
data: a list of Pyg Data, if you only have a one large graph, you should still wrap it with the list.
text: a list of list of texts. e.g. [node_text, edge_text, label_text] this is will be converted to pooled vector representation.
extra: any extra data (e.g. split information) you want to save.
"""
def add_text_emb ( self , data_list , text_emb ):
"""
This function assigns generated embedding to member variables of the graph
data_list: data list returned in self.gen_data.
text_emb: list of torch text tensor corresponding to the returned text in self.gen_data. text_emb[0] = llm_encode(text[0])
"""
data_list [ 0 ]. node_text_feat = ... # corresponding node features
data_list [ 0 ]. edge_text_feat = ... # corresponding edge features
data_list [ 0 ]. class_node_text_feat = ... # class node features
data_list [ 0 ]. prompt_edge_text_feat = ... # edge features used in prompt node
data_list [ 0 ]. noi_node_text_feat = ... # noi node features, refer to the paper for the definition
return self . collate ( data_list )
def get_idx_split ( self ):
"""
Return the split information required to split the dataset, this optional, you can further split the dataset in task_constructor.py
"""
def get_task_map ( self ):
"""
Because a dataset can have multiple different tasks that requires different prompt/class text embedding. This function returns a task map that maps a task name to the desired text embedding. Specifically, a task map is of the following format.
prompt_text_map = {task_name1: {"noi_node_text_feat": ["noi_node_text_feat", [$Index in data[0].noi_node_text_feat$]],
"class_node_text_feat": ["class_node_text_feat",
[$Index in data[0].class_node_text_feat$]],
"prompt_edge_text_feat": ["prompt_edge_text_feat", [$Index in data[0].prompt_edge_text_feat$]]},
task_name2: similar to task_name 1}
Please refer to examples in data/ for details.
"""
return self . side_data [ - 1 ]
def get_edge_list ( self , mode = "e2e" ):
"""
Defines how to construct prompt graph
f2n: noi nodes to noi prompt node
n2f: noi prompt node to noi nodes
n2c: noi prompt node to class nodes
c2n: class nodes to noi prompt node
For different task/mode you might want to use different prompt graph construction, you can do so by returning a dictionary. For example
{"f2n":[1,0], "n2c":[2,0]} means you only want f2n and n2c edges, f2n edges have edge type 1, and its text embedding feature is data[0].prompt_edge_text_feat[0]
"""
if mode == "e2e_link" :
return { "f2n" : [ 1 , 0 ], "n2f" : [ 3 , 0 ], "n2c" : [ 2 , 0 ], "c2n" : [ 4 , 0 ]}
elif mode == "lr_link" :
return { "f2n" : [ 1 , 0 ], "n2f" : [ 3 , 0 ]}