Это официальный репозиторий «Одна модель, чтобы управлять всеми: к универсальной сегментации медицинских изображений с текстовыми подсказками».
Это универсальная модель сегментации, дополненная знаниями, основанная на беспрецедентном сборе данных (72 общедоступных набора 3D-медицинских данных сегментации), которая может сегментировать 497 классов из 3 различных методов (МР, КТ, ПЭТ) и 8 областей человеческого тела, подсказанных текстом (анатомическими терминология).
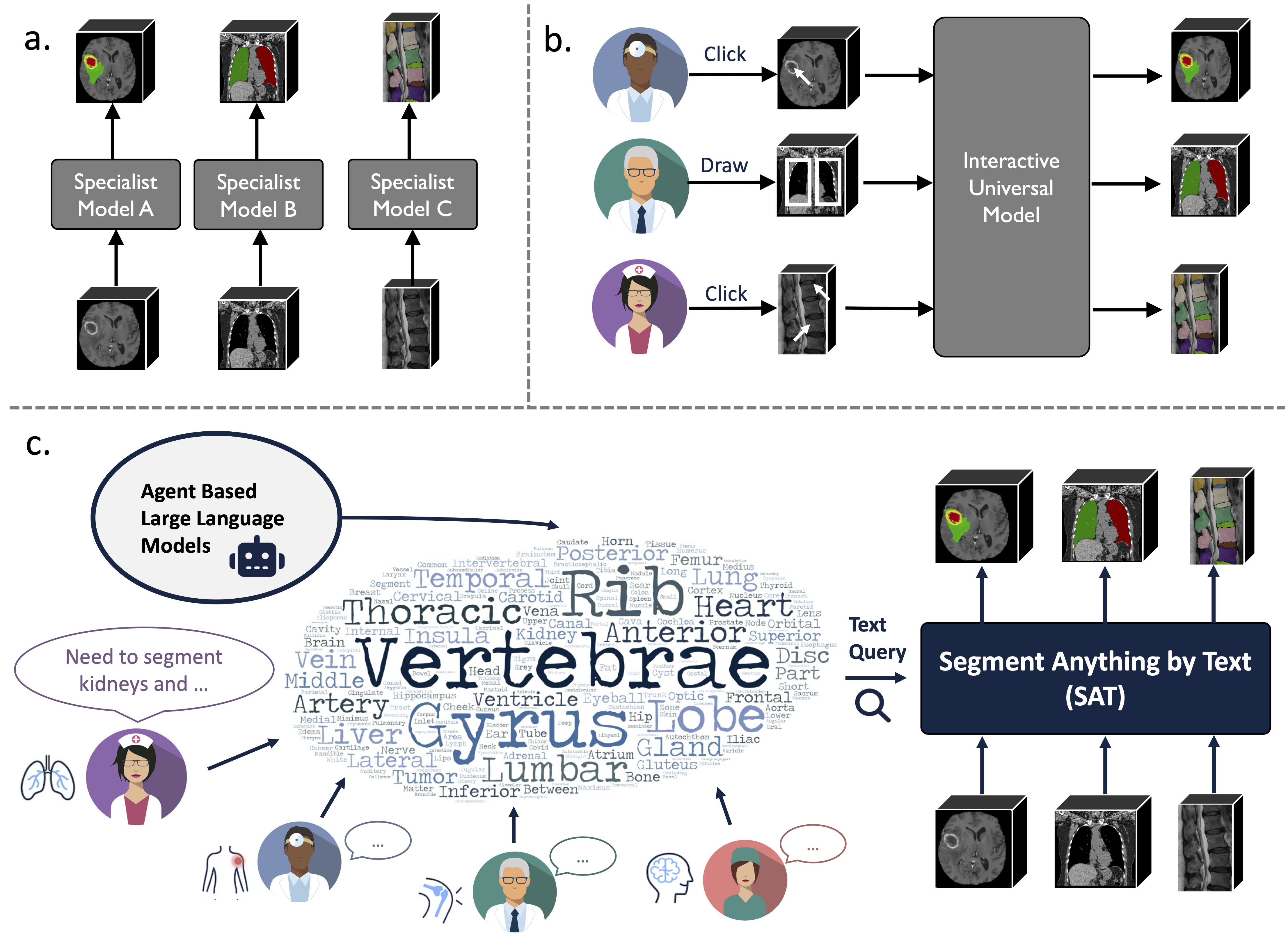
Это может быть мощным и более эффективным средством, чем обучение и внедрение ряда специализированных моделей. Узнайте больше на нашем сайте или в газете.
2024.08 ? На основе SAT и больших языковых моделей мы создаем комплексный, крупномасштабный и регионально ориентированный набор данных для интерпретации 3D-КТ грудной клетки. Он содержит сегментацию на уровне органов по 196 категориям и многоуровневые отчеты, где каждое предложение основано на соответствующей сегментации. Проверьте это на обнимающем лице.
2024.06 ? Мы выпустили код для создания SAT-DS , коллекции из 72 общедоступных наборов данных сегментации, содержащих более 22 тысяч 3D-изображений, 302 тысяч масок сегментации и 497 классов из 3 различных методов (МРТ, КТ, ПЭТ) и 8 областей человеческого тела, на основе которых мы строим САТ. Мы также предлагаем ярлыки для загрузки наборов данных 42/72, которые предварительно обработаны и упакованы нами для вашего удобства и готовы к немедленному использованию после загрузки и извлечения. Подробности проверьте в этом репо.
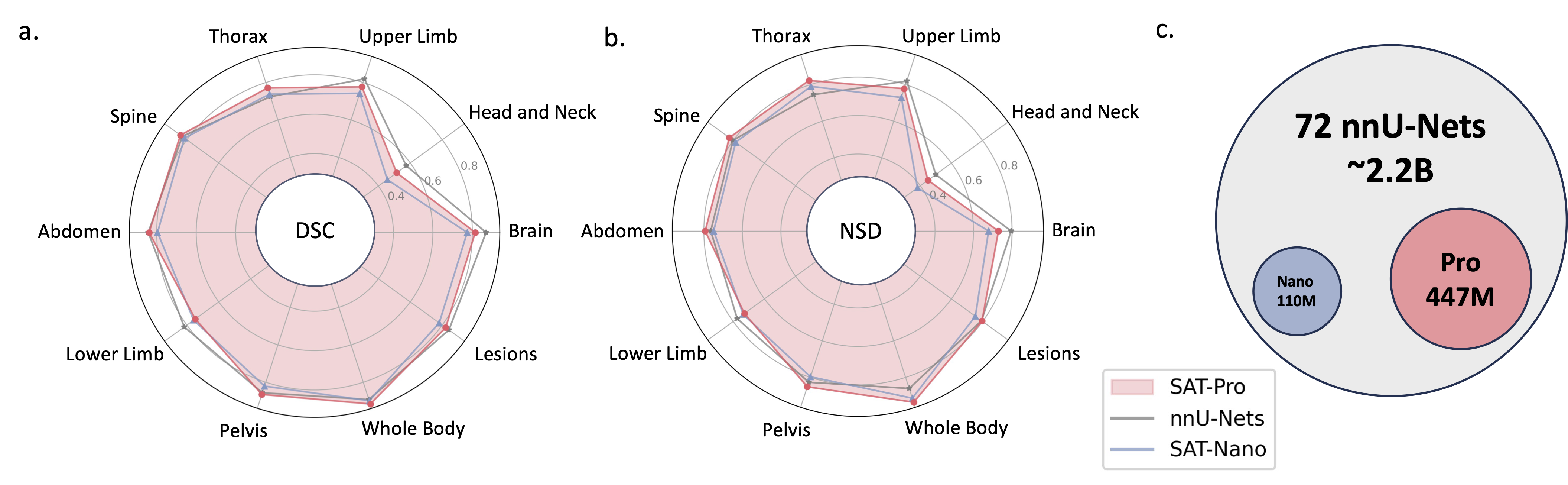
2024.05 ? Мы обучаем новую версию SAT с моделью большего размера ( SAT-Pro ) и большим количеством наборов данных ( 72 ), и теперь она поддерживает 497 классов! Мы также обновляем SAT-Nano и выпускаем несколько вариантов SAT-Nano, основанных на различных визуальных основах (U-Mamba и SwinUNETR) и текстовых кодировщиках (MedCPT и BERT-Base). Более подробную информацию об этом обновлении можно найти в нашей новой статье.
Реализация U-Net опирается на настроенную версию динамических сетевых архитектур, для ее установки:
cd model
pip install -e dynamic-network-architectures-main
Еще несколько ключевых требований:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
Вам также необходимо установить mamba_ssm , если вам нужен вариант SAT-Nano U-Mamba.
С1. Создайте среду, следуя requirements.txt .
С2. Загрузите контрольную точку SAT и Text Encoder с сайта HuggingFace.
С3. Подготовьте данные в файле JSONL. Проверьте демо в data/inference_demo/demo.jsonl .
image (путь к изображению), labe (имя целей сегментации), dataset (к какому набору данных принадлежит образец) и modality (КТ, МРТ или домашнее животное) необходимы для каждого сегментируемого образца. Модальности и классы, поддерживаемые SAT, можно найти в Таблице 12 документа.
orientation_code (ориентация) по умолчанию — RAS , что подходит для большинства изображений в аксиальной плоскости. Для изображений в сагиттальной плоскости (например, исследование позвоночника) установите значение ASR . Входное изображение должно иметь форму H,W,D Наш код обработки данных нормализует входное изображение с точки зрения ориентации, интенсивности, интервала и т. д. Два успешно обработанных изображения можно найти в demoprocessed_data . Убедитесь, что нормализация выполнена правильно, чтобы гарантировать производительность SAT.
С4. Запустите вывод с помощью SAT-Pro?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d — это размер пакета патчей входного изображения, который необходимо настроить в зависимости от памяти графического процессора (см. таблицу ниже); --max_queries рекомендуется устанавливать больше, чем классы в наборе данных вывода, если только память вашего графического процессора не очень ограничена;
| Модель | пакетный размер_3d | Память графического процессора |
|---|---|---|
| САТ-Про | 1 | ~ 34 ГБ |
| САТ-Про | 2 | ~ 62 ГБ |
| САТ-Нано | 1 | ~ 24 ГБ |
| САТ-Нано | 2 | ~ 36 ГБ |
С5. Проверьте --rcd_dir для выходных данных. Результаты организованы по наборам данных. Для каждого случая будут найдены входное изображение, агрегированный результат сегментации и папка, содержащая сегментации каждого класса. Все выходные данные сохраняются в виде файлов nifiti. Вы можете визуализировать их с помощью ITK-SNAP.
Если вы хотите использовать SAT-Nano, обученный на 72 наборах данных, просто измените --vision_backbone на «UNET» и соответственно измените --checkpoint и --text_encoder_checkpoint .
Для других вариантов SAT-Nano (обученных на 49 наборах данных):
UNET-Ours: установите --vision_backbone 'UNET' и --text_encoder 'ours' ;
UNET-CPT: установите --vision_backbone 'UNET' и --text_encoder 'medcpt' ;
UNET-BB: установите --vision_backbone 'UNET' и --text_encoder 'basebert' ;
UMamba-CPT: установите --vision_backbone 'UMamba' и --text_encoder 'medcpt' ;
SwinUNETR-CPT: установите --vision_backbone 'SwinUNETR' и --text_encoder 'medcpt' ;
Некоторая подготовка перед началом тренировки:
sh/ чтобы начать процесс обучения. Возьмем, к примеру, SAT-Pro: sbatch sh/train_sat_pro.sh
Это также требует создания тестовых данных после этого репозитория. Вы можете обратиться к сценарию slurm sh/evaluate_sat_pro.sh чтобы начать процесс оценки:
sbatch sh/evaluate_sat_pro.sh
Если вы используете этот код для своего исследования или проекта, укажите:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}