Набор молекулярных и вычислительных рабочих процессов для пространственного тотального секвенирования РНК (STRS)
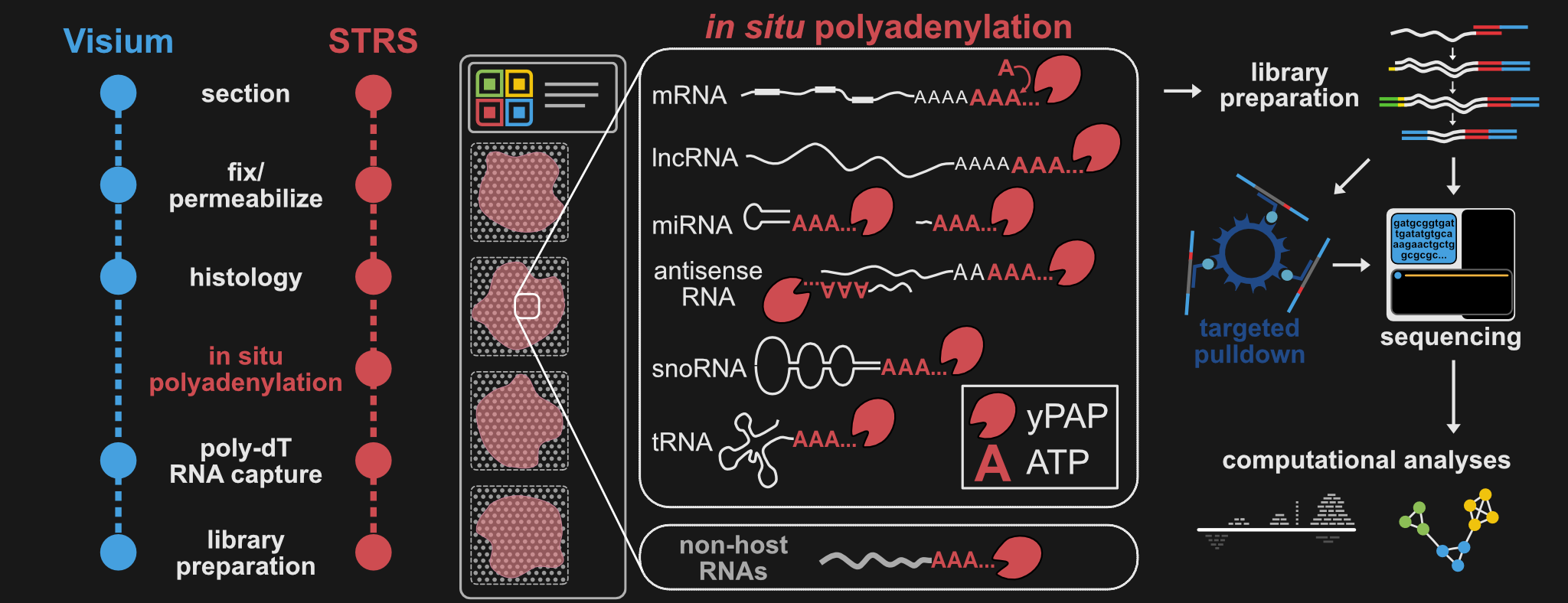
В этом репозитории есть все протоколы, конвейеры и сценарии, которые вам понадобятся для запуска и анализа STRS практически на любом образце!
txg_snake — разрабатываемым конвейером для библиотек 10x, особенно для наборов данных STRS!МакКеллар и др., Природная биотехнология , 2022 г.
ссылка на препринт bioRxiv
manuscripts : файлы препринта и последующей рукописи в формате .pdf (надеюсь!)
protocols : документы Microsoft Word и .pdf протоколов, используемых здесь.
pipelines : рабочие процессы Snakemake, использованные в нашем исследовании. К ним относятся конвейеры выравнивания для данных STRS (kallisto, STARsolo и miRge3.0), небольших данных RNAseq (STAR и miRge3.0), SmartSeqTotal (kallisto) и VASAdrop (kallisto).
scripts : весь остальной код, который мы использовали! В основном содержит сценарии R и служебные функции, используемые в нашем пространственном анализе.
references : информация и сценарии по эталонным геномам и аннотациям, использованным в этом исследовании.
resources : различные метаданные, списки генов и другая информация, которую мы использовали для анализа наших данных, для получения более подробной информации см. файлы README в каждом подкаталоге.
Наборы данных STRS: GSE200481
Данные SkM/сердечной малой РНКсек: GSE200480
Данные одноядерной общей РНКсек (клеточная линия C2C12): GSE209780
Данные Атласа малых РНК (Исакова и др., ПНАС , 2020): GSE119661
Данные SmartSeqTotal (Исакова и др., ПНАН , 2021): GSE151334
Данные VASAdrop (Салмен и др., Nature Biotechnology , 2022 г.): GSE176588.
Ивейн Де Вламинк ([email protected])
Бенджамин Д. Косгроув ([email protected])
Дэвид В. МакКеллар ([email protected])