Этот проект включает в себя различные методы выборки населения и оценку различных методов. Мы включаем множество ситуаций, которые могут привести к смещению оценки уровня инфекции на основе выборки, в том числе случаи отсутствия ответа, частота ложноположительных/отрицательных результатов, возможность профиля передачи для пациентов в период заражения. Основываясь на модели EpiABM, этот пакет также может вывести лучший метод отбора проб, запустив моделирование передачи заболевания, чтобы увидеть ошибку прогнозирования каждого метода отбора проб.
EpiOS пока недоступен в PyPI, но модуль можно установить локально. Каталог следует сначала загрузить на ваш локальный компьютер, а затем его можно установить с помощью команды:
pip install -e .Мы также рекомендуем вам установить модель EpiABM для генерации данных моделирования заражения. Сначала вы можете загрузить pyEpiabm в любое место на вашем компьютере, а затем установить его с помощью команды:
pip install -e path/to/pyEpiabm Доступ к документации можно получить с помощью значка docs выше.
Вот диаграмма классов UML для нашего проекта: 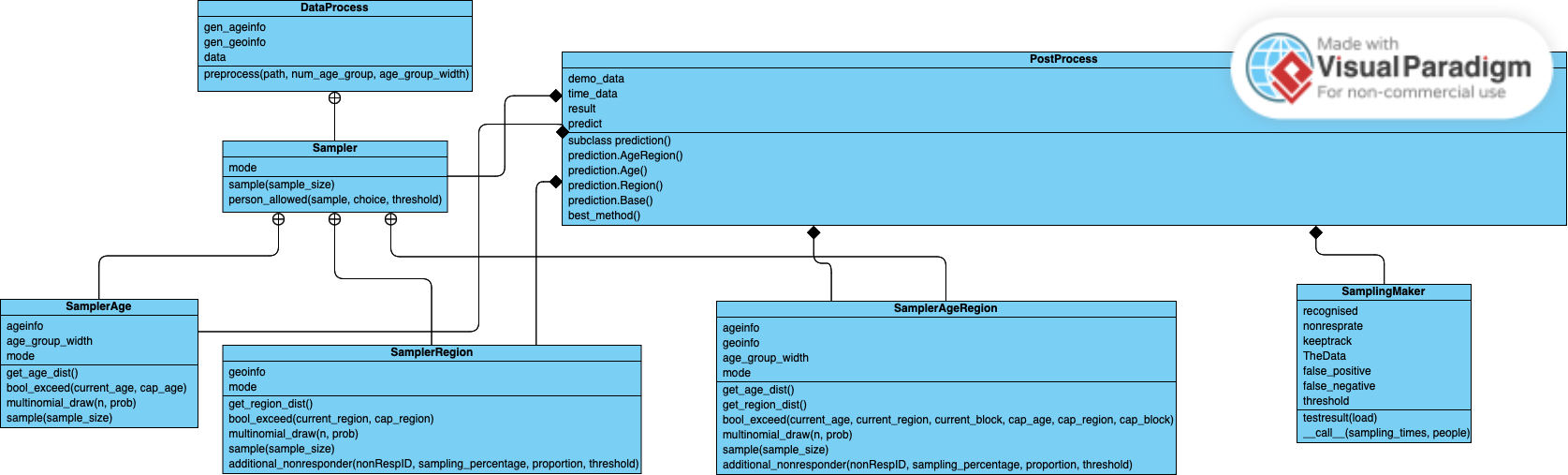
Файл params.py содержит все параметры, необходимые для этой модели. Кроме того, файлы во input папке являются примерами временных файлов, созданных в ходе предварительной обработки данных. Он будет использоваться классами сэмплеров. Параметр data_store_path в каждом классе сэмплера — это путь для хранения этих файлов.
PostProcess для создания графиков Во-первых, вам необходимо определить новый объект PostProcess и ввести demodata демографических данных и timedata заражения, сгенерированные из pyEpiabm. Во-вторых, вы можете использовать PostProcess.predict для прогнозирования на основе различных методов выборки. Вы можете напрямую вызвать метод выборки, который хотите использовать в качестве метода; затем укажите моменты времени для выборки и размер выборки. Здесь мы будем использовать AgeRegion в качестве метода выборки, [0, 1, 2, 3, 4, 5] в качестве временных точек для выборки и 3 для обозначения размера выборки. Наконец, вы можете указать, хотите ли вы учитывать тех, кто не ответил, и хотите ли вы сравнить свои результаты с истинными данными, указав параметры non_responder и comparison .
В примере кода вы можете увидеть следующее:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)Теперь ваша фигура будет сохранена по указанному пути!
PostProcess чтобы выбрать лучший метод выборки Во-первых, вам необходимо определить новый объект PostProcess и ввести demodata демографических данных и timedata заражения, сгенерированные из pyEpiabm. Во-вторых, вы можете использовать PostProcess.best_method для сравнения производительности различных методов выборки. Вы можете предоставить методы, которые хотите сравнить; затем укажите интервалы выборки и размер выборки. В-третьих, вы можете указать, хотите ли вы учитывать неответивших и хотите ли сравнивать свои результаты с истинными данными, указав параметры non_responder и comparison . Кроме того, поскольку методы выборки являются стохастическими, вы можете указать количество выполняемых итераций, чтобы получить среднюю производительность. Более того, для ускорения можно включить parallel_computation . Наконец, вы можете включить hyperparameter_autotune , чтобы автоматически находить наилучшую комбинацию гиперпараметров.
В примере кода вы можете увидеть следующее:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed