
Этот репозиторий является официальной реализацией CELL-E 2: перевод белков в изображения и обратно с помощью двунаправленного преобразователя текста в изображение.
Создайте виртуальную среду и установите необходимые пакеты с помощью:
pip install -r requirements.txt
Далее установите torch = 2.0.0 с соответствующей версией CUDA.
Модели доступны на Hugging Face.
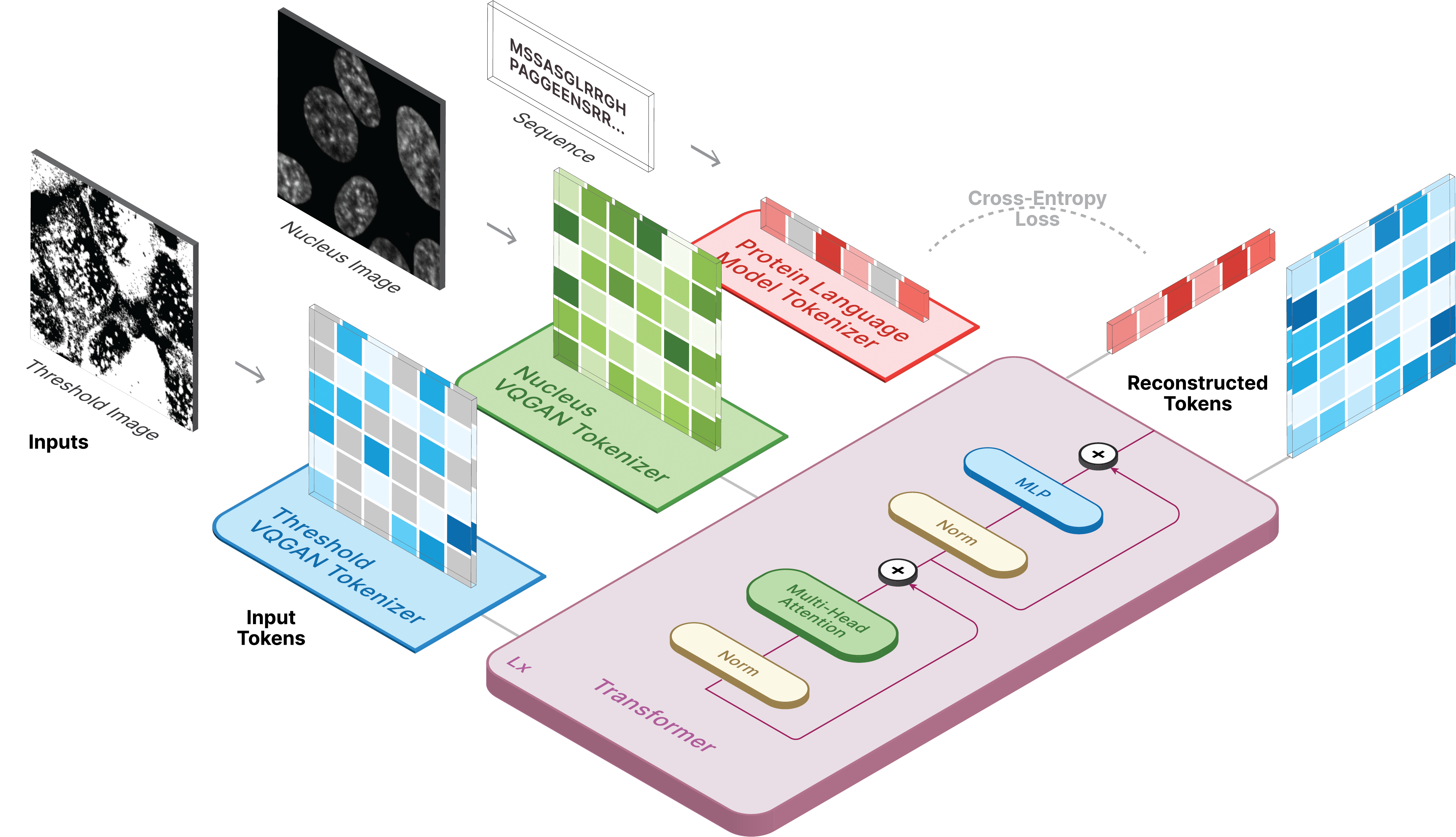
У нас также есть два места, где вы можете делать прогнозы на основе своих собственных данных!
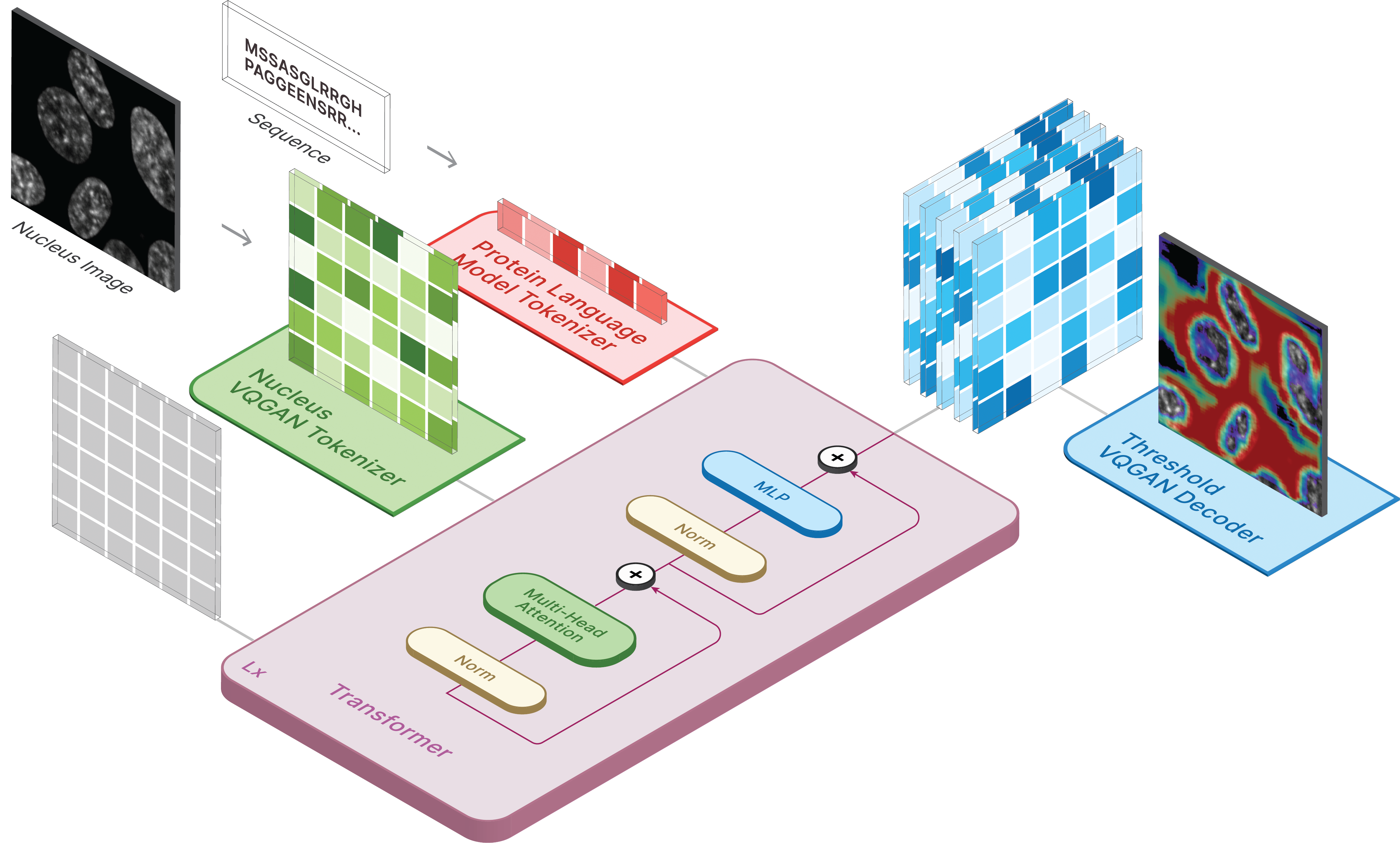
Чтобы создавать изображения, установите сохраненную модель в качестве ckpt_path. Этот метод может быть нестабильным, поэтому обратитесь к Demo.ipynb , чтобы увидеть другой способ загрузки.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )Обучение CELL-E происходит в 3 этапа:
Если вы используете изображение порога белка, установите threshold: True для набора данных.
Мы используем слегка модифицированную версию кода taming-transformers.
Для обучения запустите следующий скрипт:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
Дополнительные флаги, такие как --gpus , можно найти в исходном репозитории.
Мы предоставляем сценарии для загрузки изображений Human Protein Atlas и OpenCell в папке сценариев. data_csv необходим для загрузчика данных. Вы должны создать CSV-файл, который содержит столбцы nucleus_image_path , protein_image_path , metadata_path , split (train или val) и sequence (необязательно). Предполагается, что этот файл находится в той же папке общих data , что и файлы изображений и метаданных.
Метаданные — это JSON, который должен сопровождать каждую последовательность белка. Если последовательность не отображается в data_csv , она должна появиться в metadata.json с ключом с именем protein_sequence .
Добавление сюда дополнительной информации может быть полезно для запроса отдельных белков. Их можно получить с помощью retrieve_metadata , который создает переменную self.metadata внутри объекта набора данных.
Для обучения запустите следующий скрипт:
python celle_main.py --base configs/celle.yaml -t True
Укажите --gpus в том же формате, что и VQGAN.
CELL-E содержит следующие опции:
ckpt_path : Возобновить предыдущее обучение CELL-E 2. Сохраненная модель с помощью state_dictvqgan_model_path : сохраненная модель изображения белка (с state_dict) для кодировщика изображения белка.vqgan_config_path : сохраненная модель изображения белка в формате yaml.condition_model_path : Сохраненная модель состояния (ядра) (с state_dict) для кодировщика изображений белка.condition_config_path : Ямл модели сохраненного условия (ядра)num_images : 1, если используется только кодировщик изображения белка, 2, если включен кодировщик условного изображения.image_key : nucleus , target или thresholddim : Размер встраивания языковой модели.num_text_tokens : общее количество токенов в языковой модели (33 для ESM-2)text_seq_len : Общее количество рассматриваемых аминокислот.depth : глубина модели трансформатора, чем глубже, тем лучше за счет видеопамяти.heads : количество голов, используемых при многоголовом внимании.dim_head : размер головы вниманияattn_dropout : Уровень потери внимания во время обученияff_dropout : Частота отсева с прямой связью при обученииloss_img_weight : взвешивание, применяемое к реконструкции изображения. вес текста = 1loss_text_weight : взвешивание, применяемое для реконструкции условного изображения.stable : Нормальные веса (для случаев взрывающихся градиентов)learning_rate : Скорость обучения для оптимизатора Адама.monitor : параметр, используемый для сохранения моделей. Пожалуйста, сообщите нам, если вы решите использовать наш код для какой-либо части вашего исследования.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}