Репозиторий, содержащий данные и код для генотипирования гуара ( Cyamopsis тетрагонолоба (L.) Taub.) и проекта GWAS. Краткое описание содержимого репозитория:
Скрипт make_consensus_vcf.py использовался для создания окончательного набора данных вариантов с использованием необработанных вызовов вариантов GATK-HC, NGSEP и TASSEL 5, сценарий make_snp_stats.py использовался с теми же входными данными для изучения статистики качества вызовов вариантов.
Блокнот callset_refinement_filtering.ipynb использовался для запуска контроля качества вариантов и создания окончательного отфильтрованного набора данных общих вариантов.
Блокнот genetic_analysis.ipynb использовался для выполнения большинства генетических анализов, включая PCA, анализ неравновесия по сцеплению и анализ ассоциаций на основе обобщенной линейной модели.
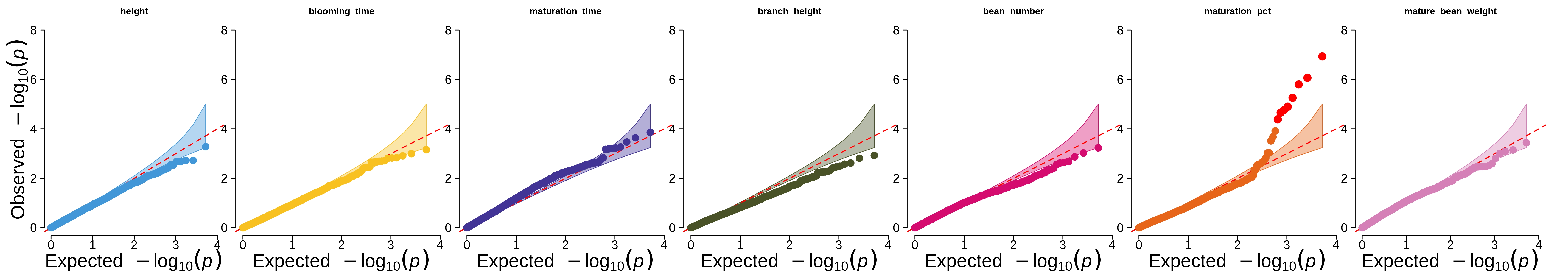
Скрипт prepare_farmcpu.py использовался для форматирования входных данных для инструмента FarmCPU (график QQ результирующих значений p показан в статье и ниже).
Файл admix_file_create.py использовался для предварительной обработки генотипов для анализа ADMIXTURE (частично это было сделано с использованием Hail в genetic_analysis.ipynb ).
Скрипт guar_stat.R использовался для статистического анализа окончательных данных, проведения общегеномного анализа ассоциаций с использованием инструмента FarmCPU и построения основных показателей.
Скрипт parse_vcf_fcpu.py использовался для форматирования окончательной таблицы генотипов SNP, доступной в виде файла genotypes.xlsx в этом репозитории.