Этот пакет предоставляет обученные модели U-net для сегментации легких. На данный момент доступны четыре модели:
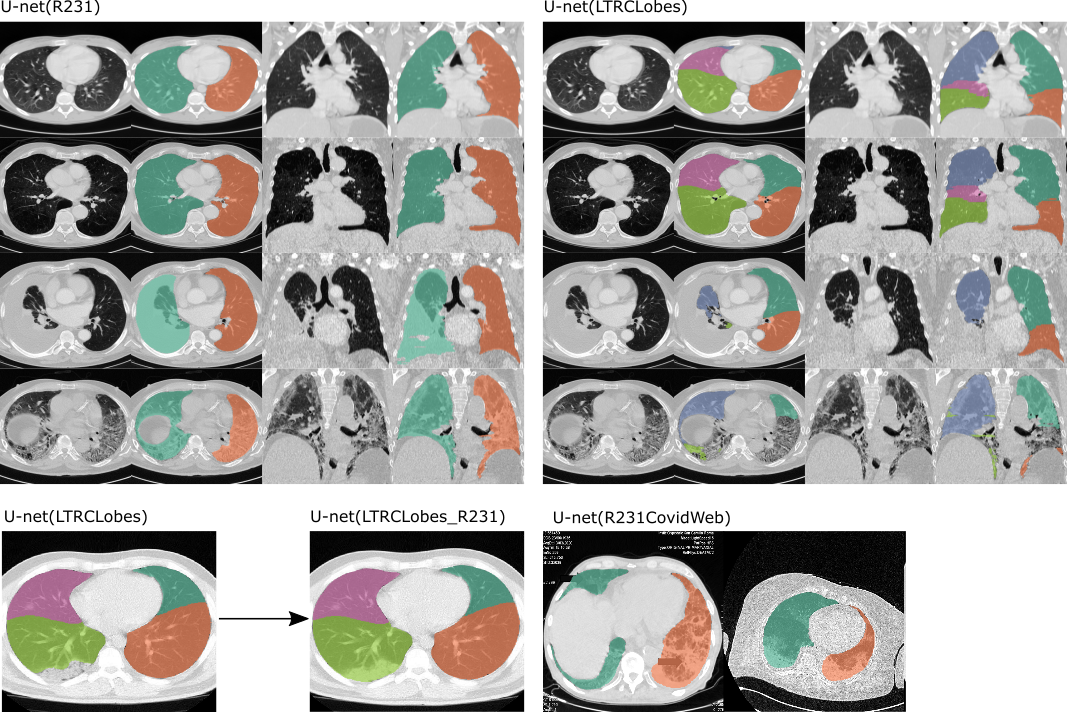
U-net(R231): Эта модель была обучена на большом и разнообразном наборе данных, который охватывает широкий диапазон визуальных переменных. Модель выполняет сегментацию на отдельные срезы, извлекает правое и левое легкое отдельно, включая воздушные карманы, опухоли и выпот. Трахея не будет включена в сегментацию легких. https://doi.org/10.1186/s41747-020-00173-2
U-net(LTRCLobes): эта модель была обучена на подмножестве набора данных LTRC. Модель выполняет сегментацию отдельных долей легких, но ее эффективность ограничена, когда присутствуют плотные патологии или когда трещины не видны на каждом срезе.
U-net(LTRCLobes_R231): будет запущена модель R231 и LTRCLobes и объединены результаты. Ложноотрицательные результаты из LTRCLobes будут заполнены предсказаниями R231 и сопоставлены с меткой соседа. Ложные срабатывания от LTRCLobes будут удалены. Процесс слияния требует больших вычислительных ресурсов и, в зависимости от данных и результатов, может занять до нескольких минут на каждый том.
U-net (R231CovidWeb)
Примеры применения двух моделей . Слева: U-сетка (R231) позволяет различать левое и правое легкое и включает очень плотные области, такие как выпот (третий ряд), опухоль или тяжелый фиброз (четвертый ряд). Справа: U-net(LTRLobes) различает доли легких, но не включает очень плотные области. LTRCLobes_R231 объединит результаты LTRCLobes и R231. R231CovidWeb обучен на дополнительных данных о COVID-19.
Семантика вывода :
Две модели этикеток (слева-справа):
1 = Правое легкое
2 = левое легкое
Пять моделей этикеток (доли легких):
1 = левая верхняя доля
2 = левая нижняя доля
3 = правая верхняя доля
4 = правая средняя доля
5 = правая нижняя доля
Более интересные исследования данных КТ легких можно найти на веб-сайте нашей исследовательской группы: https://www.cir.meduniwien.ac.at/research/lung/
Если вы используете этот код или одну из обученных моделей в своей работе, обратитесь к:
Хофманнингер Дж., Прайер Ф., Пан Дж. и др. Автоматическая сегментация легких при рутинной визуализации — это, прежде всего, проблема разнообразия данных, а не методологическая проблема. Eur Radiol Exp 4, 50 (2020). https://doi.org/10.1186/s41747-020-00173-2
Этот документ содержит подробное описание используемого набора данных, тщательную оценку модели U-net (R231) и сравнение с эталонными методами.
pip install lungmask
или
pip install git+https://github.com/JoHof/lungmask
В Windows, в зависимости от ваших настроек, может потребоваться заранее установить Torch: https://pytorch.org
Время выполнения вывода с поддержкой только ЦП и графического процессора сильно различается. При использовании графического процессора обработка тома занимает всего несколько секунд, при использовании только процессора — несколько минут. Чтобы использовать графический процессор, убедитесь, что ваша установка факела поддерживает CUDA. В случае ошибок cuda из-за нехватки памяти уменьшите размер пакета до 1 с помощью необязательного аргумента --batchsize 1
lungmask INPUT OUTPUT
Если INPUT указывает на файл, файл будет обработан. Если INPUT указывает на каталог, в каталоге будет выполняться поиск серий DICOM. Самый большой найденный объем (с точки зрения количества вокселей) будет использоваться для расчета маски легких. OUTPUT — имя выходного файла. Поддерживаются все форматы ITK.
Выберите модель:
По умолчанию будет использоваться U-net (R231). Однако вы можете указать альтернативную модель, например LTRCLobes...
lungmask INPUT OUTPUT --modelname LTRCLobes
Для дополнительных опций введите:
lungmask -h
from lungmask import LMInferer
import SimpleITK as sitk
inferer = LMInferer()
input_image = sitk.ReadImage(INPUT)
segmentation = inferer.apply(input_image) # default model is U-net(R231)
input_image должен быть объектом SimpleITK.
Загрузите альтернативную модель следующим образом:
inferer = LMInferer(modelname="R231CovidWeb")
Чтобы использовать возможность объединения моделей (например, LTRCLobes_R231), используйте:
inferer = LMInferer(modelname='LTRCLobes', fillmodel='R231')
Начиная с версии 0.2.9, массивы numpy поддерживаются в качестве входных томов. В этом режиме предполагается, что входной массив numpy имеет следующий формат для каждой оси:
Модель работает только на полных срезах. На обрабатываемом срезе должно быть видно все легкое, а для сегментации легкое должно быть окружено тканью. Однако модель вполне устойчива к случаям с обрезанным полем зрения, пока легкое окружено тканью.
lungmask INPUT OUTPUT --modelname R231CovidWeb
Обычная модель U-net (R231) очень хорошо подходит для компьютерной томографии COVID-19. Однако коллекции срезов и отчетов о случаях заболевания из Интернета часто обрезаются, аннотируются или кодируются в обычных форматах изображений, так что исходные значения единиц Хаунсфилда (HU) можно лишь оценить. Данные обучения модели U-net(R231CovidWeb) были дополнены срезами COVID-19, которые были сопоставлены с обычными форматами изображений в HU. Данные были собраны и подготовлены компанией MedSeg (http://medicalsegmentation.com/covid19/). Хотя обычная U-сеть (R231) показала очень хорошие результаты для этих изображений, могут быть случаи, когда эта модель даст немного улучшенную сегментацию. Обратите внимание, что при использовании изображений из Интернета вам необходимо сопоставить изображения обратно с HU. В этом сообщении блога описывается, как это можно сделать. Альтернативно вы можете установить тег --noHU . 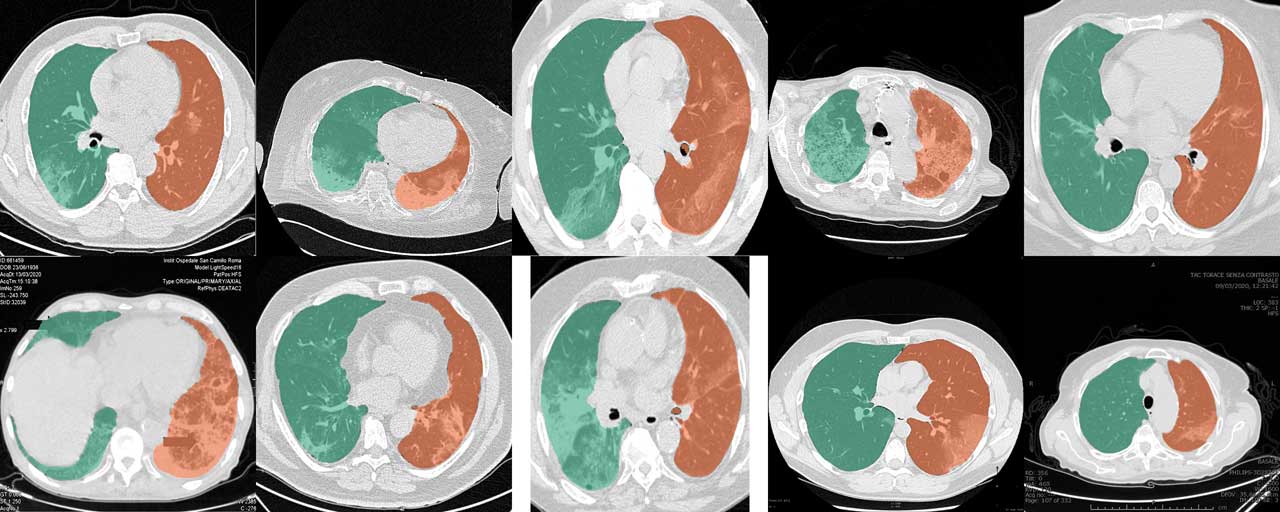
Эта функция доступна только в версиях от 0.2.5 до 0.2.14. Начиная с версии 0.2.5 эти изображения поддерживаются. Используйте тег --noHU , если вы обрабатываете изображения, не закодированные в HU. Имейте в виду, что модели обучались на правильных компьютерных томограммах, закодированных в HU. Результаты для обрезанных, аннотированных изображений со сдвигом очень высокой и очень низкой интенсивности могут быть не очень надежными. При использовании тега --noHU можно обрабатывать только отдельные фрагменты.