MedSegDiff — платформа на основе диффузионно-вероятностной модели (DPM) для сегментации медицинских изображений. Алгоритм разработан в нашей статье MedSegDiff: Сегментация медицинских изображений с помощью диффузионной вероятностной модели и MedSegDiff-V2: Сегментация медицинских изображений на основе диффузии с преобразователем.
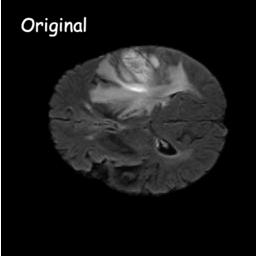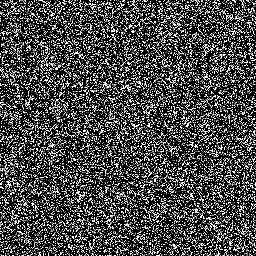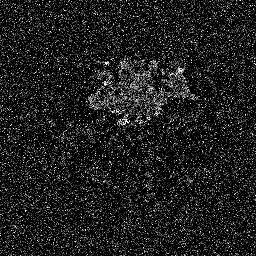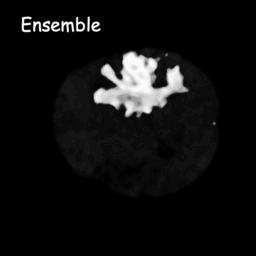 Модели диффузии работают путем уничтожения обучающих данных путем последовательного добавления гауссовского шума, а затем обучения восстановлению данных путем обращения этого процесса зашумления вспять. После обучения мы можем использовать модель диффузии для генерации данных, просто пропуская случайно выбранный шум через обученный процесс шумоподавления. В этом проекте мы расширяем эту идею на сегментацию медицинских изображений. Мы используем исходное изображение в качестве условия и генерируем несколько карт сегментации из случайных шумов, а затем выполняем их ансамбль для получения окончательного результата. Этот подход учитывает неопределенность медицинских изображений и превосходит предыдущие методы по нескольким критериям.
Модели диффузии работают путем уничтожения обучающих данных путем последовательного добавления гауссовского шума, а затем обучения восстановлению данных путем обращения этого процесса зашумления вспять. После обучения мы можем использовать модель диффузии для генерации данных, просто пропуская случайно выбранный шум через обученный процесс шумоподавления. В этом проекте мы расширяем эту идею на сегментацию медицинских изображений. Мы используем исходное изображение в качестве условия и генерируем несколько карт сегментации из случайных шумов, а затем выполняем их ансамбль для получения окончательного результата. Этот подход учитывает неопределенность медицинских изображений и превосходит предыдущие методы по нескольким критериям.
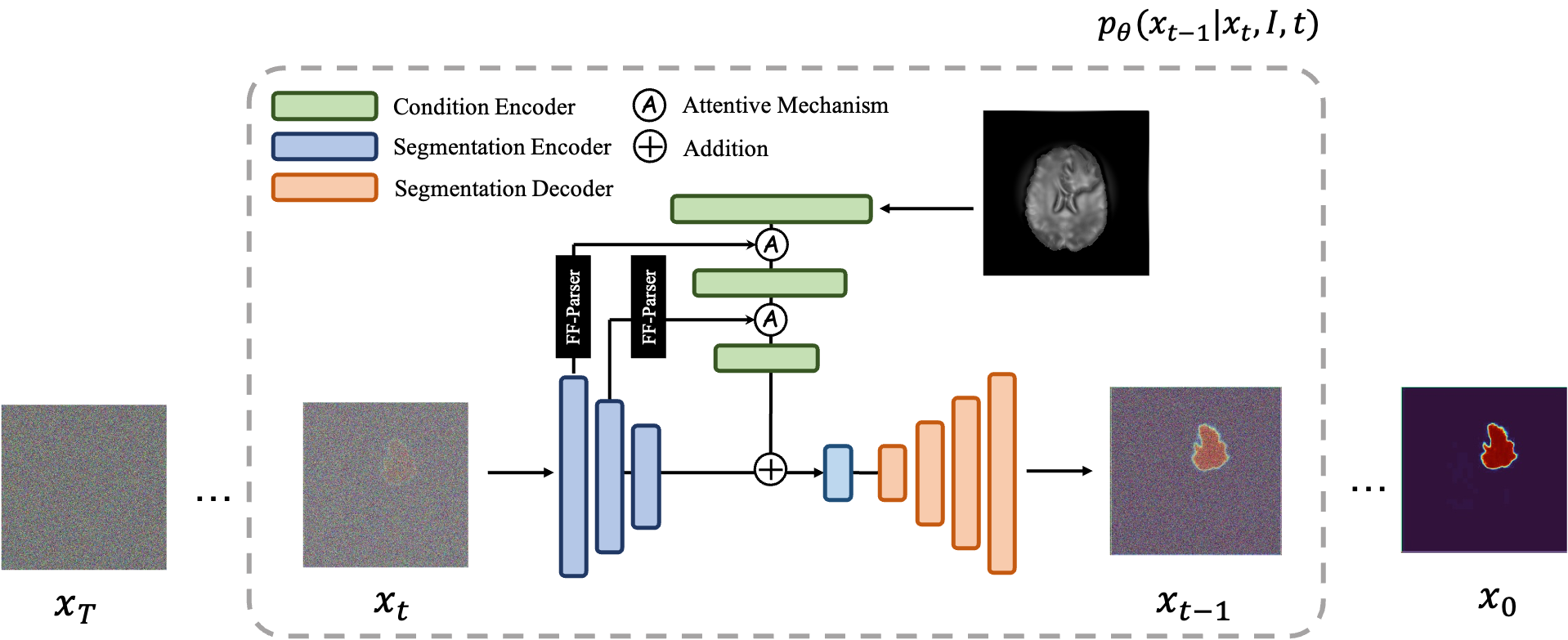 | 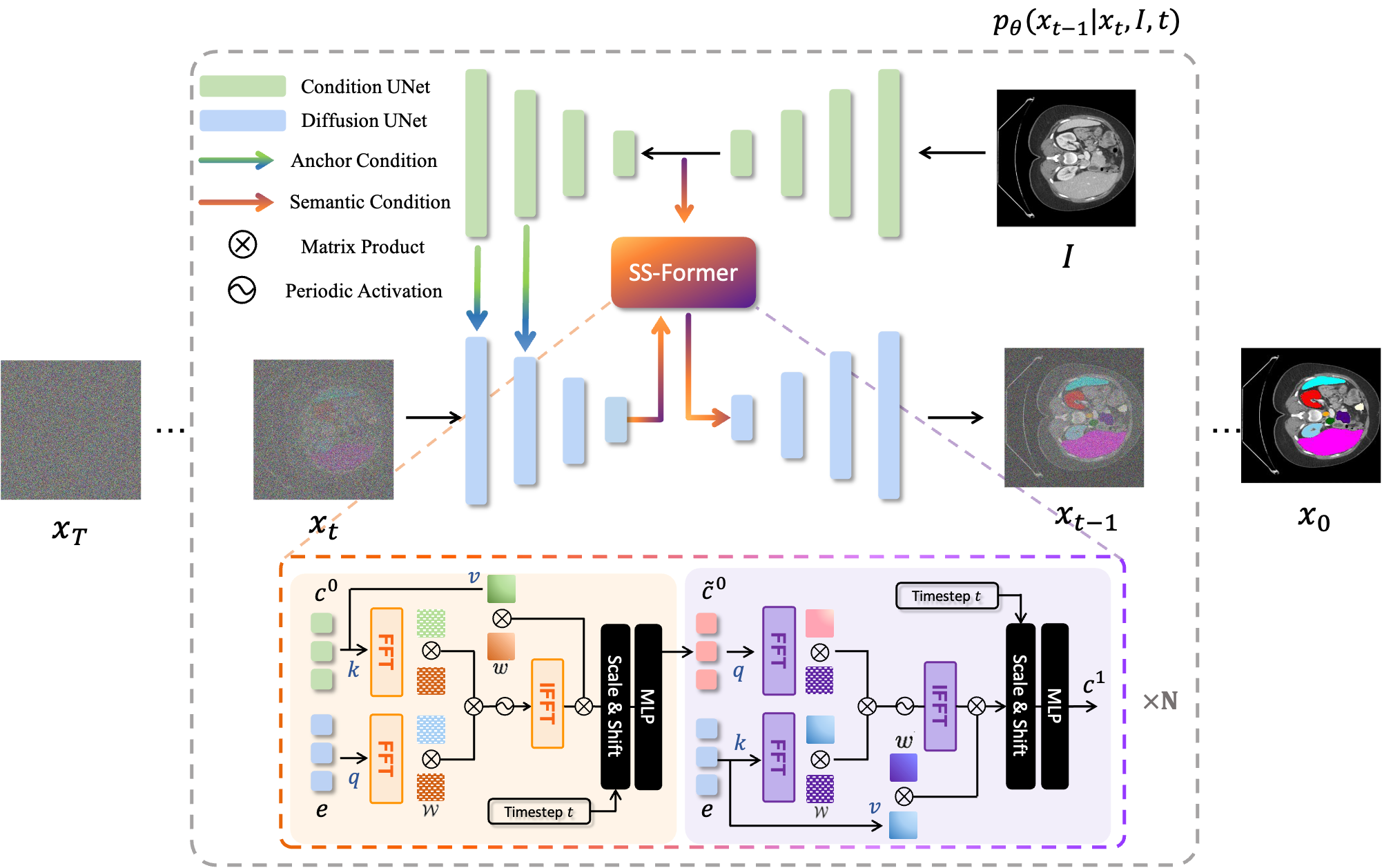 |
|---|---|
| МедСегДифф-V1 | MedSegDiff-V2 |
--dpm_solver True .python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images* pip install -r requirement.txt
data
| ----ISIC
| ----Test
| | | ISBI2016_ISIC_Part1_Test_GroundTruth.csv
| | |
| | ----ISBI2016_ISIC_Part1_Test_Data
| | | ISIC_0000003.jpg
| | | .....
| | |
| | ----ISBI2016_ISIC_Part1_Test_GroundTruth
| | ISIC_0000003_Segmentation.png
| | | .....
| |
| ----Train
| | ISBI2016_ISIC_Part1_Training_GroundTruth.csv
| |
| ----ISBI2016_ISIC_Part1_Training_Data
| | ISIC_0000000.jpg
| | .....
| |
| ----ISBI2016_ISIC_Part1_Training_GroundTruth
| | ISIC_0000000_Segmentation.png
| | .....
Для обучения запустите: python scripts/segmentation_train.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Для выборки запустите: python scripts/segmentation_sample.py --data_name ISIC --data_dir *input data direction* --out_dir *output data direction* --model_path *saved model* --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
Для оценки запустите python scripts/segmentation_env.py --inp_pth *folder you save prediction images* --out_pth *folder you save ground truth images*
По умолчанию образцы будут сохранены в папке ./results/
data
└───training
│ └───slice0001
│ │ brats_train_001_t1_123_w.nii.gz
│ │ brats_train_001_t2_123_w.nii.gz
│ │ brats_train_001_flair_123_w.nii.gz
│ │ brats_train_001_t1ce_123_w.nii.gz
│ │ brats_train_001_seg_123_w.nii.gz
│ └───slice0002
│ │ ...
└───testing
│ └───slice1000
│ │ ...
│ └───slice1001
│ │ ...
Для обучения запустите: python scripts/segmentation_train.py --data_dir (where you put data folder)/data/training --out_dir output data direction --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --lr 1e-4 --batch_size 8
Для выборки запустите: python scripts/segmentation_sample.py --data_dir (where you put data folder)/data/testing --out_dir output data direction --model_path saved model --image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16 --diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False --num_ensemble 5
...
Запустить MedSegDiff для других наборов данных просто. Просто напишите еще один файл загрузчика данных после ./guided_diffusion/isicloader.py или ./guided_diffusion/bratsloader.py . Добро пожаловать в открытые вопросы, если у вас возникнут какие-либо проблемы. Мы будем признательны, если вы предоставите свои расширения набора данных. В отличие от естественных изображений, медицинские изображения сильно различаются в зависимости от разных задач. Расширение обобщения метода требует усилий каждого.
Чтобы обучить точную модель, например, MedSegDiff-B в статье, установите гиперпараметры модели следующим образом:
--image_size 256 --num_channels 128 --class_cond False --num_res_blocks 2 --num_heads 1 --learn_sigma True --use_scale_shift_norm False --attention_resolutions 16
диффузионные гиперпараметры:
--diffusion_steps 1000 --noise_schedule linear --rescale_learned_sigmas False --rescale_timesteps False
Чтобы ускорить выборку:
--diffusion_steps 50 --dpm_solver True
работать на нескольких графических процессорах:
--multi-gpu 0,1,2 (for example)
обучающие гиперпараметры, такие как:
--lr 5e-5 --batch_size 8
и установите --num_ensemble 5 при выборке.
Выполнение около 100 000 шагов в процессе обучения будет сходиться на большинстве наборов данных. Обратите внимание: хотя на большинстве последующих этапов потери не уменьшатся, качество результатов все равно улучшается. Подобный процесс наблюдается и в других приложениях DPM, например в создании изображений. Надеюсь, кто-нибудь умный скажет мне, почему?
Вскоре я опубликую его производительность при меньшем размере пакета (подходящем для работы на графическом процессоре с 24 ГБ) для сравнения?
Настройка для раскрытия всего его потенциала (MedSegDiff++):
--image_size 256 --num_channels 512 --class_cond False --num_res_blocks 12 --num_heads 8 --learn_sigma True --use_scale_shift_norm True --attention_resolutions 24
Затем обучите его с размером пакета --batch_size 64 и выберите номер ансамбля --num_ensemble 25 .
Приглашаем вас внести свой вклад в MedSegDiff. Любой метод может улучшить производительность или ускорить алгоритм. Я пишу MedSegDiff V2 с целью публикации в журналах Nature/CVPR. Я рад указать участников в качестве своих соавторов?
Код много скопирован из openai/improved-diffusion, WuJunde/ MrPrism, WuJunde/ DiagnosisFirst, LuChengTHU/dpm-solver, JuliaWolleb/Diffusion-based-Segmentation, hojonathanho/diffusion,guide-diffusion, bigmb/Unet-Segmentation-Pytorch-Nest -оф-Унец, nnUnet, lucidrains/vit-pytorch
Пожалуйста, процитируйте
@inproceedings{wu2023medsegdiff,
title={MedSegDiff: Medical Image Segmentation with Diffusion Probabilistic Model},
author={Wu, Junde and FU, RAO and Fang, Huihui and Zhang, Yu and Yang, Yehui and Xiong, Haoyi and Liu, Huiying and Xu, Yanwu},
booktitle={Medical Imaging with Deep Learning},
year={2023}
}
@article{wu2023medsegdiff,
title={MedSegDiff-V2: Diffusion based Medical Image Segmentation with Transformer},
author={Wu, Junde and Ji, Wei and Fu, Huazhu and Xu, Min and Jin, Yueming and Xu, Yanwu}
journal={arXiv preprint arXiv:2301.11798},
year={2023}
}
https://ko-fi.com/jundewu