
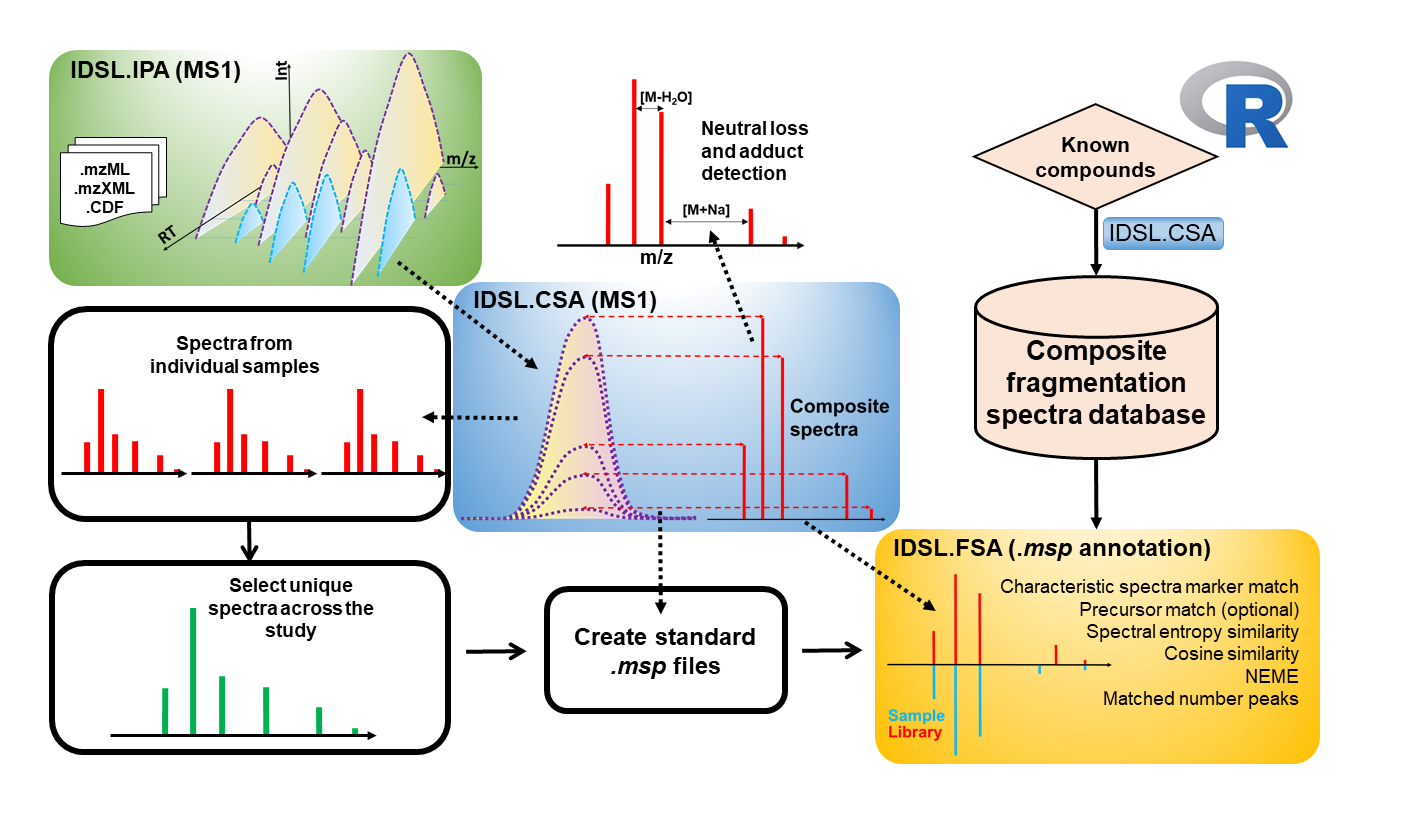
Композитный анализ спектра (IDSL.CSA) R для анализа данных масс -спектрометрии был разработан в интегрированной лаборатории науки о данных для метаболомики и экспосомики (idsl.me) . Этот пакет может использоваться для деконволюции спектров фрагментации, полученных с помощью различных аналитических методов, таких как анализ деконволюции (CSA ) только для MS1 ( CSA ), данные о сборе, зависимом , Все-ионная фрагментация (AIF) и анализы Swath-MS. Цель пакета IDSL.CSA - помочь в оптимизации процесса анализа данных и улучшении общей аннотации химической структуры в областях метаболомики и экспозимики.
Start на таблице параметров IDSL.CSA. install.packages("IDSL.CSA")
Перед обработкой данных масс-спектрометрии ( MZXML , MZML , NETCDF ) с использованием рабочего процесса IDSL.CSA, данные масс-спектрометрии должны обрабатываться с использованием рабочего процесса IDSL.IPA для получения хроматографической информации о пиках ( M/Z-RT ). Когда хроматографическая информация индивидуальных и агрегированных выравниваемых пиковых списков была сгенерирована с использованием рабочего процесса idsl.ipa, загрузите электронный таблица параметров idsl.csa и выберите параметры соответственно, а затем используйте эту таблицу в качестве ввода для рабочего потока idsl.csa:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Следуйте этим шагам для быстрого тематического исследования (n = 33) ST002263, который имеет точные данные Thermo Q-гибридных орбит-орбит, собранные в режимах Hilic-ESI-POS/NEG.
Обработать необработанную масс -спектрометрию и хроматографическая информация с использованием метода, описанного для idsl.ipa
Анализ композитных спектров требует 39 параметров, распределенных по 5 отдельным разделам для полномасштабного анализа. Для этого исследования используйте значения параметров по умолчанию, представленные в электронной таблице IDSL.CSA. Далее предоставьте информацию для
2.1. Выберите «Да» для Param0001 на вкладке Start , чтобы обработать только рабочий процесс CSA .
2.2. CSA0005 для адреса местоположения данных HRMS (данные HRMS уровня MS1)
2.3. CSA0008 для адреса каталога peaklists , сгенерированного рабочим процессом IDSL.IPA
2.4. CSA0009 для адреса каталога peak_alignment , сгенерированного рабочим процессом IDSL.IPA
2.5. CSA0011 для местоположения вывода (файлы и EICS .msp)
2.6 Вы также можете увеличить количество потоков обработки, используя CSA0004 в соответствии с вашей вычислительной мощностью
Запустите эту команду в консоли или терминале R/Rstudio:
library(IDSL.CSA)
IDSL.CSA_workflow("Address of the CSA parameter spreadsheet")
Вы можете анализировать результаты по адресу, который вы предоставили для CSA0011 .
4.1. CSA_MSP включает файл .msp
4.2. CSA_ADDUCT_ANNOTATION включает в себя списки с потенциальной информацией о аддукте
4.3. PEACK_ALINMENT_SUBSET включает в себя подмножества выровненных пиковых таблиц для основных ионов в каждом кластере CSA
4.4. Aligned_spectra_table включает информацию для агрегации CSA на выровненной таблице
[1] Fakouri Baygi, S., Kumar, Y. Barupal, DK idsl.csa: анализ композитных спектров для химической аннотации наборов данных о нецелевых метаболомиках. Аналитическая химия , 2023 , 95 (25) , 9480–9487.
[2] Fakouri Baygi, S., Kumar, Y. Barupal, Dk Idsl. IPA характеризует органическое химическое пространство в нецелевых наборах данных LC/HRMS. Journal of Proteome Research , 2022 , 21 (6) , 1485-1494.