
Isling-это инструмент для обнаружения вирусной или векторной интеграции в парных чтениях. Пожалуйста, прочитайте нашу газету для полной информации.
Если у вас установлены conda и snakemake , чтобы запустить с (включенными) тестовыми данными локально:
git clone https://github.com/aehrc/isling.git && cd isling
snakemake --configfile test/config/test.yml --cores <cores> --use-conda
Если у вас установлен snakemake и singularity , вы можете использовать вместо этого:
snakemake --configfile test/config/test.yml --cores <cores> --use-singularity
В качестве альтернативы, если у вас установлен Docker, на MacOS вы можете запустить:
docker run --rm -it -v"$(pwd)"/out:/opt/isling/out szsctt/isling:latest snakemake --configfile test/config/test.yml --cores 1
Это будет использовать файл конфигурации и данные внутри контейнера, и результаты будут отображаться в папке, out в вашем текущем рабочем каталоге. На Linux вам нужно будет запустить эту команду в качестве корня, а в Windows вам нужно будет настроить синтаксис Bind -Mount (аргумент -v ).
Входные данные (чтения и хост и вирусные ссылки) указаны NI A Config File - Для ваших собственных данных вам необходимо изменить пример файла config ( test/config/test.yml ), чтобы указывать на ваши собственные данные. См configfile.md для получения дополнительной информации о формате файла конфигурации.
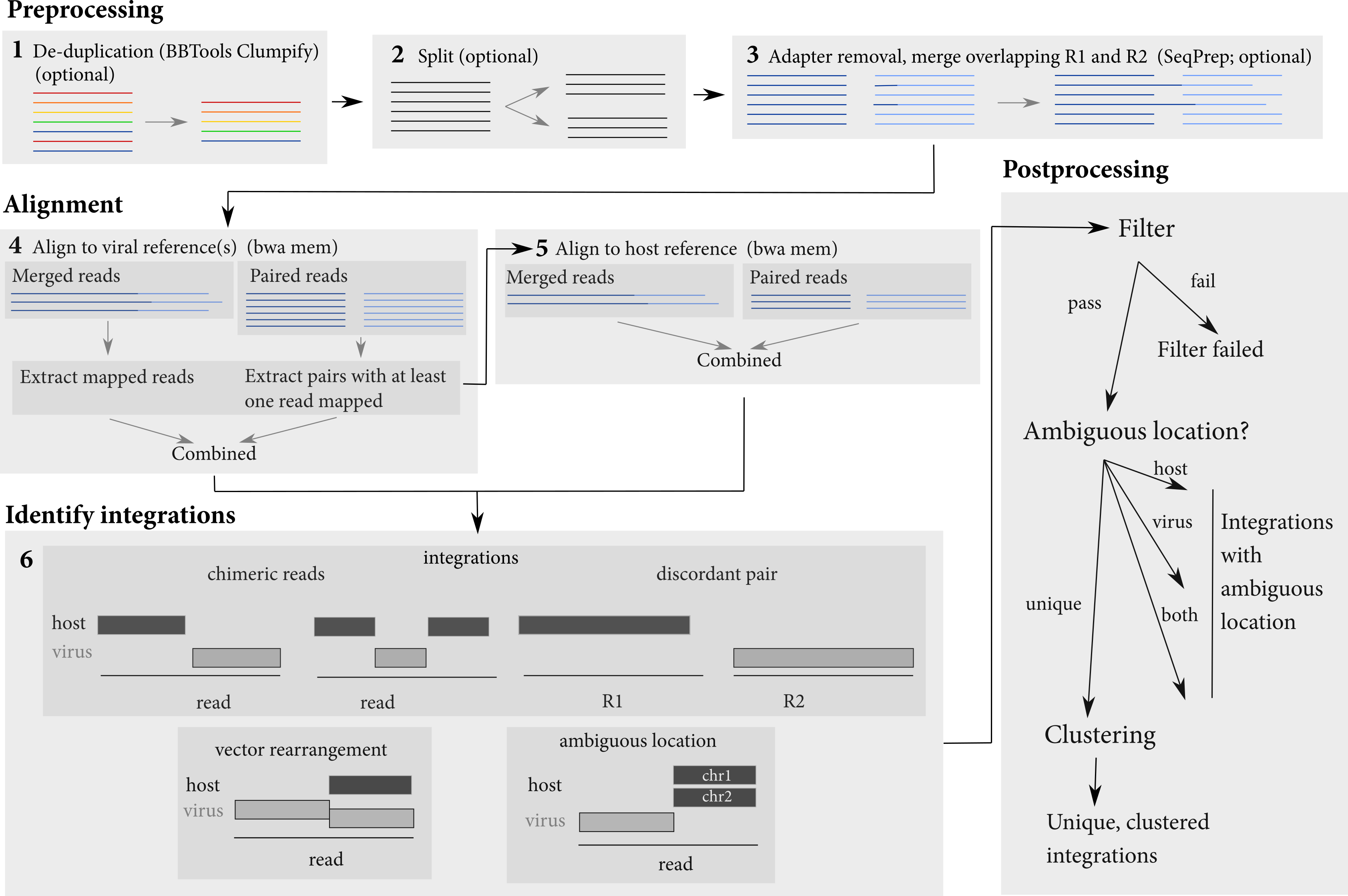
Трубопровод выполняет несколько шагов, чтобы идентифицировать сайты интеграции. Он принимает в качестве входных наборов данных, состоящих из файлов FASTQ или файлов BAM. Он выполняет некоторую предварительную обработку чтения (слияние перекрывающихся чтений, необязательно), а затем выравнивает их как к хозяину, так и с вирусной последовательности. Чтения сначала выровнены с вирусной последовательности, а затем выровненные чтения извлекаются и выровнены с хостом. Эти выравнивания используются для выявления вирусной интеграции.
Isling требует snakemake и singularity (рекомендуется) или conda для снабжения зависимостей. Добавление, python версии 3.5 или выше требуется и pandas (они должны быть автоматически установлены при установке snakemake с conda .
Alternativley, используйте версию Docker, которая содержит Isling и все зависимости.
Требуемые входы - это файл конфигурации, который определяет хост и вирусные/векторные ссылки, и требуются чтения. Укажите все входы в файле конфигурации. Isling в настоящее время работает только для парных чтений.
См. File configfile.md для описания формата этого файла конфигурации.
Сайты интеграции ISLING в формате, разделенном в TAB, в выходном каталоге, указанном в файле конфигурации.
В выходной папке одна папка создается для каждого набора данных в файле конфигурации, и для каждого набора данных интеграции можно найти в каталоге ints . Для каждого образца будет один набор выходных файлов.
Для каждого образца существует ряд выходных файлов, которые могут представлять интерес для конкретных случаев использования.
<sample>.<host>.<virus>.integrations.txt : Информация обо всех обнаруженных соединениях, независимо от того, проходили ли они фильтры или нет<sample>.<host>.<virus>.integrations.post.txt : Информация о обнаруженных соединениях, которые проходили все фильтры<sample>.<host>.<virus>.integrations.post.unique.txt : Информация о обнаруженных соединениях, которые проходили все фильтры и имеют однозначное место как на хосте, так и на вектор/вирус<sample>.<host>.<virus>.integrations.post.unique.merged.txt : результат объединения перекрывающихся соединений или соединений с одинаковыми координатами как у хоста, так и в вирусе/векторе. Слияние проводится только с помощью интеграционных соединений, которые имеют однозначное место как в хосте, так и в вирусе/векторе.<sample>.<host>.<virus>.integrations.post.host_ambig.txt : Информация о обнаруженных соединениях, которые прошли все фильтры и имеют однозначный вектор местоположения/вирус, но неоднозначное место на хосте<sample>.<host>.<virus>.integrations.post.virus_ambig.txt : Информация о обнаруженных соединениях, которые проходили все фильтры и имеют однозначный хост местоположения, но неоднозначное местоположение в векторе/вирусе<sample>.<host>.<virus>.integrations.post.both_ambig.txt : Информация о обнаруженных соединениях, которые проходили все фильтры и имеют неоднозначное место как на хосте, так и на векторе/вирусе Выходные файлы дают местоположение идентифицированных интеграций и их свойства. Координаты для интеграционных соединений указаны с точки зрения их неоднозначных оснований. То есть часто существует разрыв или совпадение между хостом и вирусными частями чтения: 
Поскольку в этом случае местоположение интеграции не может быть однозначно определено, Isling выводит координаты этих оснований в геноме хозяина и вектора/вируса в качестве местоположения интеграции.
За исключением выходного файла объединенного кластера, все файлы содержат эти столбцы:
gap или overlap для химиерического чтения (хост и вирусные части, идентифицированные на одном и том же чтении), соединение является clean если у него нет неоднозначных оснований. В качестве альтернативы, если Jucntion был идентифицирован в диссонирующей паре, OverlapType является discordanthv ), либо вируса/хозяина ( vh )+ , либо -bwa ) выравнивания хозяинаbwa ) вирусного выравниванияOverlapType - это gapchimeric чтении, discordant пара или это short интеграция, при которой оба соединения наблюдались в одном и том же чтенииbwa )bwa )'xxx' Чтобы воспроизвести фигуры в рукописи Isling, см. Readme в benchmarking .