麻省理工学院(MIT)的研究人员发布了名为Boltz-1的强大开源人工智能模型,该模型在生物分子结构预测方面达到了与谷歌DeepMind的AlphaFold3相同的精度。这一突破性进展有望显着加速生物医学研究和药物开发进程,为全球科研人员提供一个高效且易于使用的工具。 Boltz-1的完全开源特性,打破了以往模型的封闭性,鼓励了全球范围内的合作与创新,这对于推动科学发现和技术进步具有深远意义。
麻省理工学院(MIT)科学家们最近发布了一款强大的开源人工智能模型,名为Boltz-1。这一创新有望显着加速生物医学研究和药物开发。
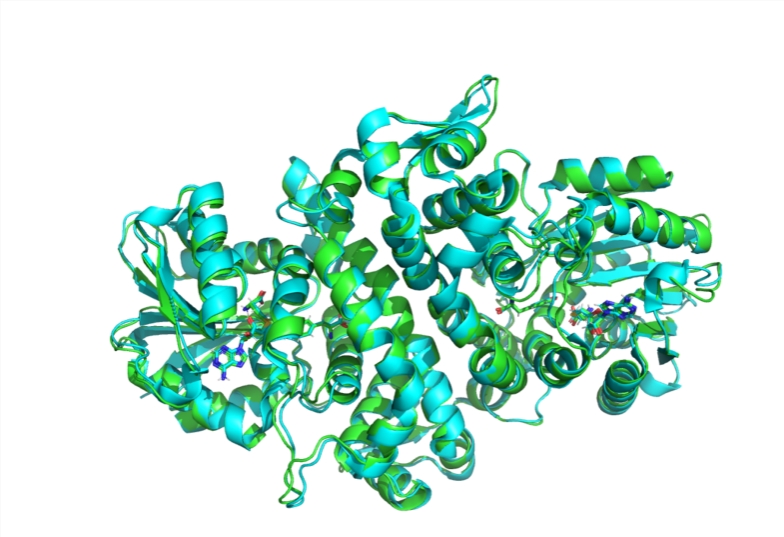
Boltz-1是首个完全开源的模型,能够在生物分子结构预测方面达到与谷歌DeepMind 的AlphaFold3相同的先进水平。该模型的开发团队来自MIT Jameel 机器学习健康诊所,主要由研究生杰里米・沃尔文德和加布里埃尔・科尔索领导,合作团队还包括MIT 的研究员萨罗・帕萨罗以及电气工程与计算机科学教授瑞吉娜・巴兹利和汤米・亚卡拉。
在12月5日的发布会上,沃尔文德和科尔索表示,他们的最终目标是促进全球合作,加速科学发现,并提供一个稳健的平台以推进生物分子建模。科尔索提到,“我们希望这能成为社区的起点”,并强调“Boltz-1” 而非“Boltz” 的命名意图在于鼓励社区的参与。
蛋白质在几乎所有生物过程中都起着关键作用,而蛋白质的形状与其功能密切相关,因此了解蛋白质的结构对于设计新药或工程化具有特定功能的新蛋白质至关重要。由于蛋白质长链氨基酸折叠成三维结构的过程极其复杂,准确预测其结构一直是科学界面临的重大挑战。
DeepMind 的AlphaFold2通过机器学习快速预测3D 蛋白质结构,精确度高得让实验科学家难以分辨。 AlphaFold3则在此基础上进行改进,采用了生成式AI 模型,但由于它并非完全开源,受到科学界的批评。因此,MIT 的研究团队着手开发Boltz-1,遵循AlphaFold3的基本思路,并在此基础上进行改进,提升模型的准确性和预测效率。
研究团队耗时四个月,进行了多次实验,克服了在蛋白质数据银行中遇到的模糊性和异质性问题。最终,他们的实验表明Boltz-1在复杂生物分子结构预测方面达到了与AlphaFold3相同的精度。
研究人员计划继续提升Boltz-1的性能,并缩短预测时间。他们还邀请研究者在GitHub 上试用Boltz-1,并通过Slack 频道与其他用户交流。研究团队希望Boltz-1能够促进更广泛的合作,激发社区的创造性应用。
项目:https://jclinic.mit.edu/democratizing-science-boltz-1/
划重点:
Boltz-1是首个完全开源的生物分子结构预测模型,达到与AlphaFold3相同的性能。
该模型的开发旨在促进全球合作,推动生物医学研究与药物开发。
MIT 团队希望通过Boltz-1简化蛋白质结构预测,使更多科研人员能够使用这一强大工具。
Boltz-1的开源特性,无疑将加速全球生物医学研究的步伐,为疾病治疗和药物研发带来新的希望。 MIT团队的努力为科学界树立了良好的榜样,也为未来AI技术在生命科学领域的应用提供了新的方向和可能性。期待Boltz-1能为人类健康事业做出更大贡献。