هذا هو المستودع الرسمي لـ "نموذج واحد يحكمهم جميعًا: نحو تقسيم عالمي للصور الطبية مع مطالبات نصية"
إنه نموذج تجزئة عالمي معزز بالمعرفة مبني على مجموعة بيانات غير مسبوقة (72 مجموعة بيانات تجزئة طبية ثلاثية الأبعاد عامة)، والتي يمكنها تقسيم 497 فئة من 3 طرق مختلفة (MR، CT، PET) و8 مناطق في الجسم البشري، مدفوعة بالنص (التشريحي). مصطلحات).
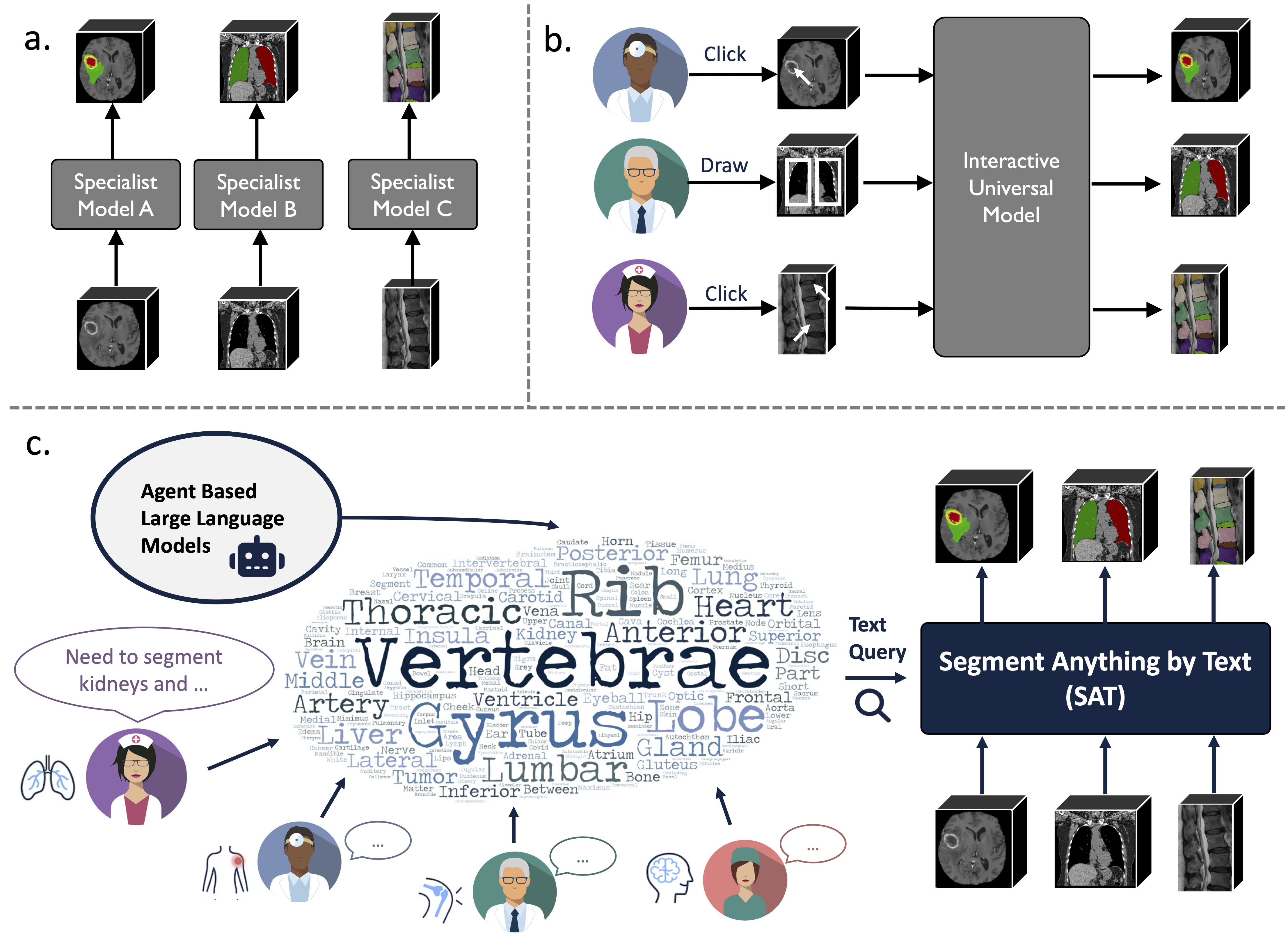
يمكن أن يكون قويًا وأكثر كفاءة من التدريب ونشر سلسلة من النماذج المتخصصة. العثور على المزيد على موقعنا أو ورقة.
2024.08 ؟ استنادًا إلى نماذج SAT والنماذج اللغوية الكبيرة، نقوم ببناء مجموعة بيانات شاملة وواسعة النطاق وموجهة للمنطقة لتفسير التصوير المقطعي المحوسب للصدر ثلاثي الأبعاد. ويحتوي على تجزئة على مستوى العضو لـ 196 فئة، وتقارير متعددة التفاصيل، حيث يتم ربط كل جملة بالتجزئة المقابلة. التحقق من ذلك على المعانقة.
2024.06 ؟ لقد أصدرنا التعليمات البرمجية لبناء SAT-DS ، وهي مجموعة مكونة من 72 مجموعة بيانات تجزئة عامة، تحتوي على أكثر من 22 ألف صورة ثلاثية الأبعاد و302 ألف قناع تجزئة و497 فئة من 3 طرق مختلفة (التصوير بالرنين المغناطيسي، والتصوير المقطعي المحوسب، والتصوير المقطعي بالإصدار البوزيتروني) و8 مناطق في الجسم البشري، والتي بناءً عليها نحن نبني SAT. كما نقدم أيضًا روابط تنزيل مختصرة لمجموعات البيانات 42/72، والتي تتم معالجتها مسبقًا وتعبئتها بواسطتنا لراحتك، وتكون جاهزة للاستخدام الفوري عند التنزيل والاستخراج. تحقق من هذا الريبو للحصول على التفاصيل.
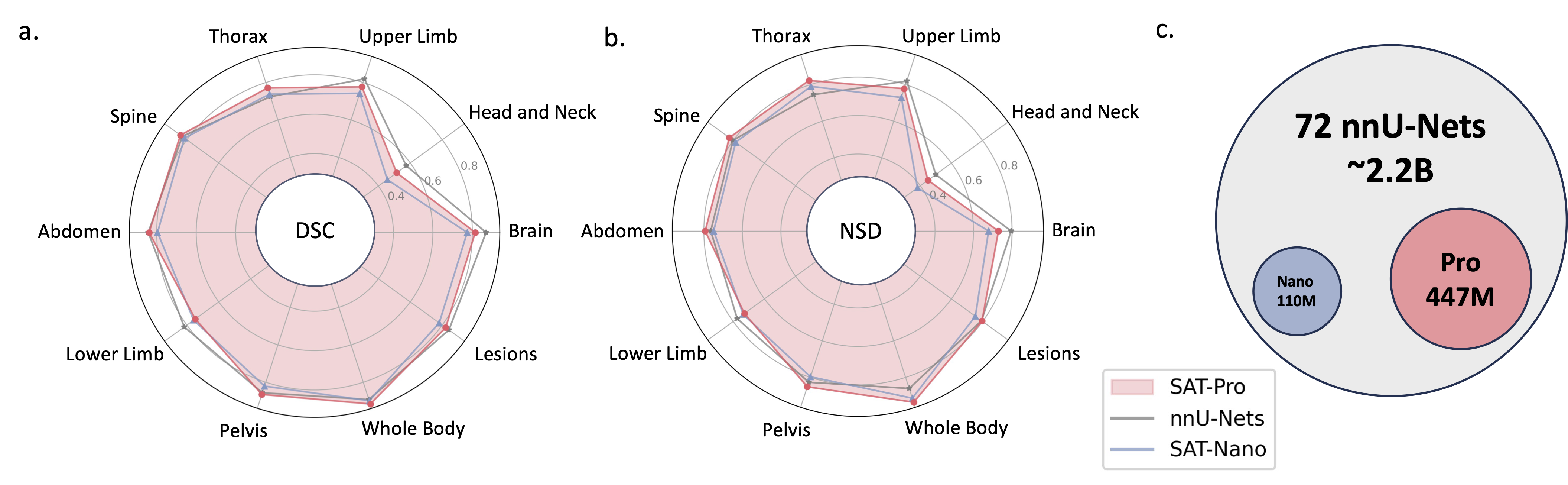
2024.05 ؟ نقوم بتدريب إصدار جديد من SAT بحجم نموذج أكبر ( SAT-Pro ) والمزيد من مجموعات البيانات ( 72 )، وهو يدعم 497 فصلًا الآن! نقوم أيضًا بتجديد SAT-Nano، وإصدار بعض المتغيرات من SAT-Nano، استنادًا إلى أعمدة بصرية مختلفة (U-Mamba وSwinUNETR) وأجهزة تشفير النصوص (MedCPT وBERT-Base). لمزيد من التفاصيل حول هذا التحديث، راجع ورقتنا الجديدة.
يعتمد تنفيذ U-Net على نسخة مخصصة من معماريات الشبكة الديناميكية، لتثبيتها:
cd model
pip install -e dynamic-network-architectures-main
بعض المتطلبات الرئيسية الأخرى:
torch>=1.10.0
numpy==1.21.5
monai==1.1.0
transformers==4.21.3
nibabel==4.0.2
einops==0.6.1
positional_encodings==6.0.1
تحتاج أيضًا إلى تثبيت mamba_ssm إذا كنت تريد إصدار U-Mamba من SAT-Nano
S1. قم ببناء البيئة باتباع requirements.txt .txt.
S2. قم بتنزيل نقطة تفتيش SAT وText Encoder من Huggingface.
S3. قم بإعداد البيانات في ملف jsonl. تحقق من العرض التوضيحي في data/inference_demo/demo.jsonl .
هناك حاجة إلى image (المسار إلى الصورة)، labe (اسم أهداف التجزئة)، dataset (مجموعة البيانات التي تنتمي إليها العينة) modality (التصوير المقطعي، أو التصوير بالرنين المغناطيسي أو الحيوانات الأليفة) لكل عينة لتقسيمها. يمكن العثور على الأساليب والفئات التي يدعمها اختبار SAT في الجدول 12 من الورقة.
orientation_code (الاتجاه) هو RAS بشكل افتراضي، والذي يناسب معظم الصور في المستوى المحوري. بالنسبة للصور في المستوى السهمي (على سبيل المثال، فحص العمود الفقري)، اضبط هذا على ASR . يجب أن تكون صورة الإدخال بالشكل H,W,D وسيقوم كود معالجة البيانات الخاص بنا بتطبيع صورة الإدخال من حيث الاتجاه والكثافة والتباعد وما إلى ذلك. يمكن العثور على صورتين تمت معالجتهما بنجاح في demoprocessed_data ، تأكد من إجراء التسوية بشكل صحيح لضمان أداء SAT.
S4. ابدأ الاستدلال باستخدام SAT-Pro ?:
torchrun
--nproc_per_node=1
--master_port 1234
inference.py
--rcd_dir 'demo/inference_demo/results'
--datasets_jsonl 'demo/inference_demo/demo.jsonl'
--vision_backbone 'UNET-L'
--checkpoint 'path to SAT-Pro checkpoint'
--text_encoder 'ours'
--text_encoder_checkpoint 'path to Text encoder checkpoint'
--max_queries 256
--batchsize_3d 2
--batchsize_3d هو حجم مجموعة تصحيحات الصور المدخلة، ويجب تعديلها بناءً على ذاكرة وحدة معالجة الرسومات (راجع الجدول أدناه)؛ - يوصى بتعيين --max_queries أكبر من الفئات الموجودة في مجموعة بيانات الاستدلال، ما لم تكن ذاكرة وحدة معالجة الرسومات الخاصة بك محدودة للغاية؛
| نموذج | Batchsize_3D | ذاكرة وحدة معالجة الرسومات |
|---|---|---|
| سات برو | 1 | ~ 34 جيجابايت |
| سات برو | 2 | ~ 62 جيجابايت |
| سات نانو | 1 | ~ 24 جيجابايت |
| سات نانو | 2 | ~ 36 جيجابايت |
S5. تحقق من --rcd_dir للمخرجات. يتم تنظيم النتائج حسب مجموعات البيانات. لكل حالة، سيتم العثور على صورة الإدخال ونتيجة التقسيم المجمعة ومجلد يحتوي على تجزئة كل فئة. يتم تخزين جميع المخرجات كملفات nifiti. يمكنك تصورها باستخدام ITK-SNAP.
إذا كنت تريد استخدام SAT-Nano الذي تم تدريبه على 72 مجموعة بيانات، فما عليك سوى تعديل --vision_backbone إلى "UNET"، وتغيير --checkpoint و- --text_encoder_checkpoint وفقًا لذلك.
بالنسبة لمتغيرات SAT-Nano الأخرى (المدربة على 49 مجموعة بيانات):
UNET-خاصتنا: set --vision_backbone 'UNET' و-- --text_encoder 'ours' ؛
UNET-CPT: تعيين --vision_backbone 'UNET' و-- --text_encoder 'medcpt' ؛
UNET-BB: تعيين --vision_backbone 'UNET' و- --text_encoder 'basebert' ؛
UMamba-CPT: set --vision_backbone 'UMamba' و-- --text_encoder 'medcpt' ؛
SwinUNETR-CPT: set --vision_backbone 'SwinUNETR' و-- --text_encoder 'medcpt' ؛
بعض الاستعدادات قبل البدء بالتدريب:
sh/ لبدء عملية التدريب. خذ سات برو على سبيل المثال: sbatch sh/train_sat_pro.sh
يتطلب هذا أيضًا إنشاء بيانات اختبار بعد هذا الريبو. يمكنك الرجوع إلى البرنامج النصي slurm sh/evaluate_sat_pro.sh لبدء عملية التقييم:
sbatch sh/evaluate_sat_pro.sh
إذا كنت تستخدم هذا الرمز لبحثك أو مشروعك، يرجى ذكر:
@arxiv{zhao2023model,
title={One Model to Rule them All: Towards Universal Segmentation for Medical Images with Text Prompt},
author={Ziheng Zhao and Yao Zhang and Chaoyi Wu and Xiaoman Zhang and Ya Zhang and Yanfeng Wang and Weidi Xie},
year={2023},
journal={arXiv preprint arXiv:2312.17183},
}