مجموعة من مسارات العمل الجزيئية والحسابية لتسلسل الحمض النووي الريبي (RNA) المكاني الإجمالي (STRS)
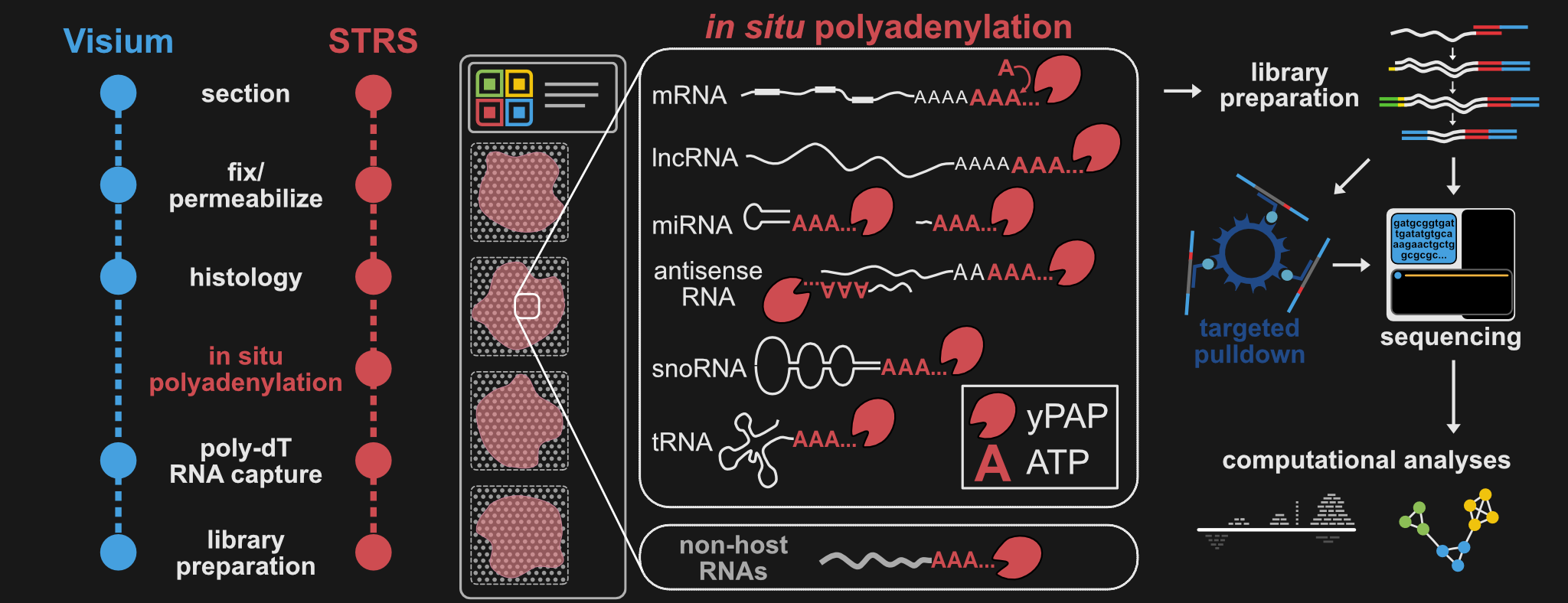
يحتوي هذا المستودع على جميع البروتوكولات وخطوط الأنابيب والبرامج النصية التي ستحتاجها لتشغيل وتحليل STRS على أي عينة تقريبًا!
txg_snake ، وهو مسار عمل قيد التنفيذ لمكتبات 10x، وخاصة مجموعات بيانات STRS!ماكيلار وآخرون، التكنولوجيا الحيوية الطبيعية ، 2022
رابط الطباعة المسبق لـ bioRxiv
manuscripts : ملفات .pdf للطبعة الأولية والمخطوطة اللاحقة (نأمل!)
protocols : مستندات Microsoft Word و.pdf الخاصة بالبروتوكولات المستخدمة هنا
pipelines : سير عمل Snakemake المستخدم في دراستنا. وتشمل هذه خطوط أنابيب المحاذاة لبيانات STRS (kallisto، STARsolo، & miRge3.0)، وبيانات RNAseq الصغيرة (STAR & miRge3.0)، وSmartSeqTotal (kallisto)، وVASAdrop (kallisto).
scripts : جميع التعليمات البرمجية الأخرى التي استخدمناها! يحتوي في الغالب على نصوص R ووظائف الأداة المساعدة المستخدمة في تحليلاتنا المكانية.
references : معلومات ونصوص عن الجينومات المرجعية والشروح المستخدمة في هذه الدراسة
resources : بيانات التعريف المتنوعة، وقوائم الجينات، والمعلومات الأخرى التي استخدمناها لتحليل بياناتنا ، راجع ملفات README في كل دليل فرعي لمزيد من التفاصيل
مجموعات بيانات STRS: GSE200481
بيانات RNAseq الصغيرة SkM/القلب: GSE200480
إجمالي بيانات RNAseq أحادية النواة (خط الخلية C2C12): GSE209780
بيانات أطلس الحمض النووي الريبي الصغيرة (Isakova et al, PNAS , 2020): GSE119661
بيانات SmartSeqTotal (Isakova et al, PNAS , 2021): GSE151334
بيانات VASAdrop (Salmen et al, Nature Biotechnology , 2022): GSE176588
إيوين دي فلامينك ([email protected])
بنيامين د. كوسجروف ([email protected])
ديفيد دبليو ماكيلار ([email protected])