يتكون هذا المشروع من طرق مختلفة لأخذ عينات من السكان وتقييم الطرق المختلفة. قمنا بتضمين الكثير من المواقف التي قد تسبب تحيزًا في تقدير مستوى الإصابة بناءً على العينة، بما في ذلك غير المستجيبين، ومعدل الإيجابية/السلبية الكاذبة، وقدرة ملف انتقال العدوى للمرضى خلال فترة إصابتهم. استنادًا إلى نموذج EpiABM، يمكن لهذه الحزمة أيضًا إخراج أفضل طريقة لأخذ العينات عن طريق تشغيل عمليات محاكاة لانتقال المرض لمعرفة خطأ التنبؤ لكل طريقة لأخذ العينات.
EpiOS ليس متاحًا بعد على PyPI، ولكن يمكن تثبيت الوحدة محليًا. يجب أولاً تنزيل الدليل على جهازك المحلي، ومن ثم يمكن تثبيته باستخدام الأمر:
pip install -e .نوصيك أيضًا بتثبيت نموذج EpiABM لإنشاء بيانات محاكاة الإصابة. يمكنك أولاً تنزيل pyEpiabm إلى أي مكان على جهازك، ومن ثم يمكن تثبيته باستخدام الأمر:
pip install -e path/to/pyEpiabm يمكن الوصول إلى الوثائق عبر شارة docs المذكورة أعلاه.
فيما يلي رسم تخطيطي لفئة UML لمشروعنا: 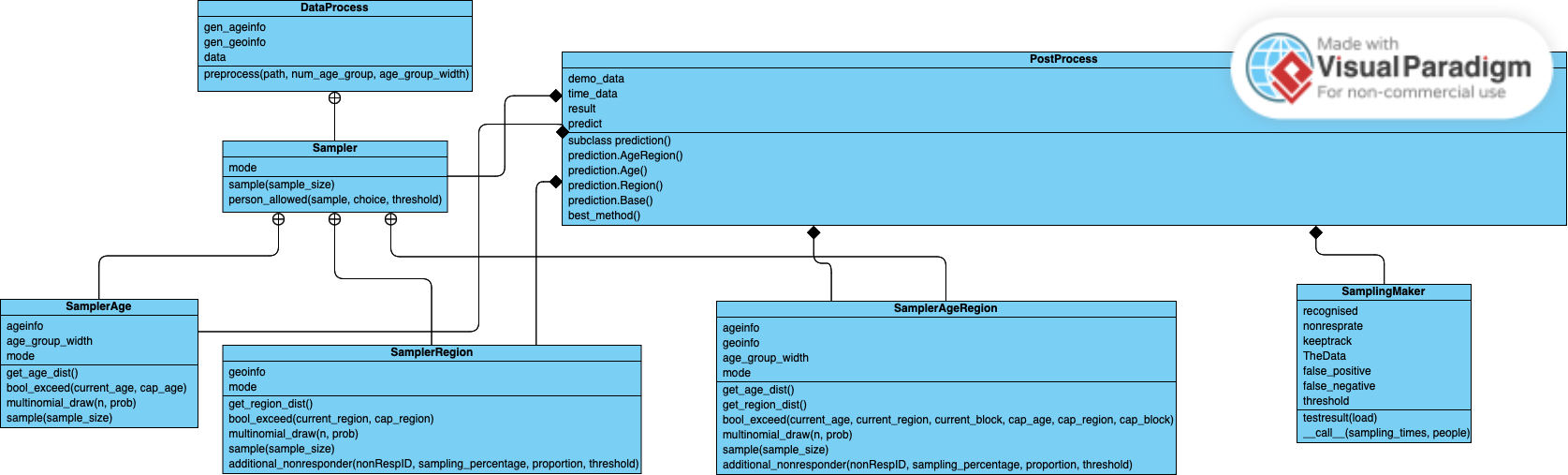
يتضمن الملف params.py جميع المعلمات المطلوبة في هذا النموذج. بالإضافة إلى ذلك، فإن الملفات الموجودة في مجلد input هي أمثلة على الملفات المؤقتة التي تم إنشاؤها أثناء المعالجة المسبقة للبيانات. سيتم استخدامه من قبل فئات العينات. المعلمة data_store_path في كل فئة من فئات العينات هي المسار لتخزين هذه الملفات.
PostProcess لإنشاء المؤامرات أولاً، تحتاج إلى تحديد كائن PostProcess جديد وإدخال البيانات demodata للبيانات الديموغرافية وبيانات timedata الإصابة التي تم إنشاؤها من pyEpiabm. ثانيًا، يمكنك استخدام PostProcess.predict لإجراء التنبؤ بناءً على طرق أخذ العينات المختلفة. يمكنك استدعاء طريقة أخذ العينات التي تريد استخدامها كطريقة مباشرة؛ ثم حدد النقاط الزمنية للعينة وحجم العينة. هنا، سوف نستخدم AgeRegion كطريقة لأخذ العينات، [0, 1, 2, 3, 4, 5] كنقاط زمنية لأخذ العينات و 3 لتكون حجم العينة. وأخيرًا، يمكنك تحديد ما إذا كنت تريد اعتبار غير المستجيبين وما إذا كنت تريد مقارنة نتائجك بالبيانات الحقيقية عن طريق تحديد المعلمة non_responder comparison .
على سبيل المثال التعليمات البرمجية، يمكنك رؤية ما يلي:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
res , diff = postprocess . predict . AgeRegion (
time_sample = [ 0 , 1 , 2 , 3 , 4 , 5 ], sample_size = 3 ,
non_responders = False ,
comparison = True ,
gen_plot = True ,
saving_path_sampling = 'path/to/save/sampled/predicted/infection/plot' ,
saving_path_compare = 'path/to/save/comparison/plot'
)الآن، سيتم حفظ الرقم الخاص بك في المسار المحدد!
PostProcess لتحديد أفضل طريقة لأخذ العينات أولاً، تحتاج إلى تحديد كائن PostProcess جديد وإدخال البيانات demodata للبيانات الديموغرافية وبيانات timedata الإصابة التي تم إنشاؤها من pyEpiabm. ثانيًا، يمكنك استخدام PostProcess.best_method لمقارنة أداء طرق أخذ العينات المختلفة. يمكنك توفير الطرق التي تريد مقارنتها؛ ثم حدد فترات أخذ العينات للعينة وحجم العينة. ثالثًا، يمكنك تحديد ما إذا كنت تريد اعتبار غير المستجيبين وما إذا كنت تريد مقارنة نتائجك بالبيانات الحقيقية عن طريق تحديد المعلمة non_responder comparison . بالإضافة إلى ذلك، نظرًا لأن طرق أخذ العينات عشوائية، يمكنك تحديد عدد التكرارات التي يتم تشغيلها للحصول على متوسط الأداء. علاوة على ذلك، يمكن تشغيل parallel_computation لتسريعها. وأخيرًا، يمكنك تشغيل hyperparameter_autotune للعثور تلقائيًا على أفضل مجموعة من المعلمات الفائقة.
على سبيل المثال التعليمات البرمجية، يمكنك رؤية ما يلي:
python import epios
postprocess = epios . PostProcess ( time_data = timedata , demo_data = demodata )
# Define the input keywards for finding the best method
best_method_kwargs = {
'age_group_width_range' : [ 14 , 17 , 20 ]
}
# Suppose we want to compare among methods Age-Random, Base-Same,
# Base-Random, Region-Random and AgeRegion-Random
# And suppose we want to turn on the parallel computation to speed up
if __name__ == '__main__' :
# This 'if' statement can be omitted when not using parallel computation
postprocess . best_method (
methods = [
'Age' ,
'Base-Same' ,
'Base-Random' ,
'Region-Random' ,
'AgeRegion-Random'
],
sample_size = 3 ,
hyperparameter_autotune = True ,
non_responder = False ,
sampling_interval = 7 ,
iteration = 1 ,
# When considering non-responders, input the following line
# non_resp_rate=0.1,
metric = 'mean' ,
parallel_computation = True ,
** best_method_kwargs
)
# Then the output will be printed