يرجى العلم أن هذا المستودع مخصص لأغراض الأرشيف فقط. لم يعد يتم الاحتفاظ بوظائف ANCOM المستقلة في هذا المستودع. يُنصح المستخدم بشدة باستخدام وظائف ancom أو ancombc في حزمة ANCOMBC R الخاصة بنا. للحصول على تقارير الأخطاء، يرجى الذهاب إلى مستودع ANCOMBC.
يطبق الكود الحالي ANCOM في مجموعات البيانات المقطعية والطولية مع السماح باستخدام المتغيرات المشتركة. يجب تضمين المكتبات التالية لتشغيل كود R:
library( nlme )
library( tidyverse )
library( compositions )
source( " programs/ancom.R " )لقد اعتمدنا منهجية ANCOM-II كخطوة معالجة مسبقة للتعامل مع أنواع مختلفة من الأصفار قبل إجراء تحليل الوفرة التفاضلية.
feature_table_pre_process(feature_table, meta_data, sample_var, group_var = NULL, out_cut = 0.05, zero_cut = 0.90, lib_cut, neg_lb)feature_table : إطار بيانات أو مصفوفة تمثل جدول OTU/SV المرصود مع الأصناف في الصفوف ( rownames ) والعينات في الأعمدة ( colnames ). لاحظ أن هذا هو جدول الوفرة المطلقة ، لا تقم بتحويله إلى جدول الوفرة النسبية (حيث إجماليات الأعمدة تساوي 1).meta_data : إطار البيانات أو المصفوفة لجميع المتغيرات والمتغيرات المشتركة محل الاهتمام.sample_var : الحرف. اسم العمود الذي يقوم بتخزين معرفات العينات.group_var : الحرف. اسم مؤشر المجموعة. مطلوب group_var للكشف عن الأصفار الهيكلية والقيم المتطرفة. للحصول على تعريفات الأصفار المختلفة (الصفر الهيكلي، والصفر الخارجي، وصفر أخذ العينات)، يرجى الرجوع إلى ANCOM-II.out_cut الكسر العددي بين 0 و1. بالنسبة لكل تصنيف، سيتم اكتشاف الملاحظات ذات نسبة توزيع الخليط الأقل من out_cut كأصفار خارجية؛ بينما سيتم اكتشاف الملاحظات التي تكون فيها نسبة توزيع الخليط أكبر من 1 - out_cut كقيم خارجية.zero_cut : الكسر العددي بين 0 و1. لا يتم تضمين الأصناف التي تحتوي على نسبة أصفار أكبر من zero_cut في التحليل.lib_cut : رقمي. لا يتم تضمين العينات ذات حجم المكتبة الأقل من lib_cut في التحليل.neg_lb : منطقي. يشير TRUE إلى أنه سيتم تصنيف الصنف على أنه صفر هيكلي في المجموعة التجريبية المقابلة باستخدام الحد الأدنى المقارب له. وبشكل أكثر تحديدًا، يشير neg_lb = TRUE إلى أنك تستخدم كلا المعيارين المذكورين في القسم 3.2 من ANCOM-II لاكتشاف الأصفار الهيكلية؛ وبخلاف ذلك، neg_lb = FALSE سوف يستخدم فقط المعادلة 1 في القسم 3.2 من ANCOM-II للإعلان عن الأصفار الهيكلية. feature_table : إطار بيانات لجدول OTU المعالج مسبقًا.meta_data : إطار بيانات للبيانات الوصفية المعالجة مسبقًا.structure_zeros : تتكون المصفوفة من 0 و1s مع 1 مما يشير إلى أن الصنف تم تحديده على أنه صفر هيكلي في المجموعة المقابلة.ANCOM(feature_table, meta_data, struc_zero, main_var, p_adj_method, alpha, adj_formula, rand_formula, lme_control)feature_table : إطار بيانات يمثل جدول OTU/SV مع الأصناف في الصفوف ( rownames ) والعينات في الأعمدة ( colnames ). يمكن أن تكون قيمة الإخراج من feature_table_pre_process . لاحظ أن هذا هو جدول الوفرة المطلقة ، لا تقم بتحويله إلى جدول الوفرة النسبية (حيث إجماليات الأعمدة تساوي 1).meta_data : إطار بيانات المتغيرات. يمكن أن تكون قيمة الإخراج من feature_table_pre_process .struc_zero : تتكون المصفوفة من 0 و1s مع 1 مما يشير إلى أن الصنف تم تحديده على أنه صفر هيكلي في المجموعة المقابلة. يمكن أن تكون قيمة الإخراج من feature_table_pre_process .main_var : الحرف. اسم المتغير الرئيسي للفائدة. يدعم ANCOM v2.1 حاليًا التصنيف main_var .p_adjust_method : الحرف. تحديد طريقة ضبط القيم الاحتمالية للمقارنات المتعددة. الافتراضي هو "BH" (إجراء Benjamini-Hochberg).alpha : مستوى الأهمية. الافتراضي هو 0.05.adj_formula : سلسلة أحرف تمثل صيغة التعديل (انظر المثال).rand_formula : سلسلة أحرف تمثل صيغة التأثيرات العشوائية في lme . لمزيد من التفاصيل، راجع ?lme .lme_control : قائمة تحدد قيم التحكم لملاءمة lme. لمزيد من التفاصيل، راجع ?lmeControl . 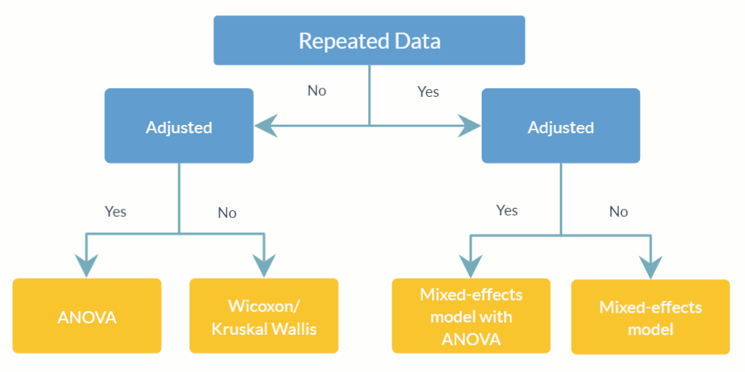
out : إطار بيانات يحتوي على إحصائية W لكل تصنيف والأعمدة اللاحقة التي تعد مؤشرات منطقية لما إذا كان OTU أو الصنف متوافرًا بشكل تفاضلي ضمن سلسلة من القطع (0.9 و0.8 و0.7 و0.6). يتم استخدام detected_0.7 بشكل شائع. ومع ذلك، يمكنك اختيار detected_0.8 أو detected_0.9 إذا كنت تريد أن تكون أكثر تحفظًا في نتائجك (FDR أصغر)، أو استخدام detected_0.6 إذا كنت ترغب في استكشاف المزيد من الاكتشافات (قوة أكبر)
fig : كائن ggplot لمؤامرة البركان.
library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_moving_pics.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig library( readr )
library( tidyverse )
otu_data = read_tsv( " data/moving-pics-table.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/moving-pics-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = SampleID )
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = NULL
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 1000 ; neg_lb = FALSE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " Subject " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " ReportedAntibioticUsage " ; rand_formula = NULL ; lme_control = NULL
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . نود هنا أن نعرف ما إذا كانت هناك بعض الأصفار الهيكلية عبر مستويات delivery المختلفة library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = NULL ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )
write_csv( res $ out , " outputs/res_ecam.csv " )
# Step 3: Volcano Plot
# Number of taxa except structural zeros
n_taxa = ifelse(is.null( struc_zero ), nrow( feature_table ), sum(apply( struc_zero , 1 , sum ) == 0 ))
# Cutoff values for declaring differentially abundant taxa
cut_off = c( 0.9 * ( n_taxa - 1 ), 0.8 * ( n_taxa - 1 ), 0.7 * ( n_taxa - 1 ), 0.6 * ( n_taxa - 1 ))
names( cut_off ) = c( " detected_0.9 " , " detected_0.8 " , " detected_0.7 " , " detected_0.6 " )
# Annotation data
dat_ann = data.frame ( x = min( res $ fig $ data $ x ), y = cut_off [ " detected_0.7 " ], label = " W[0.7] " )
fig = res $ fig +
geom_hline( yintercept = cut_off [ " detected_0.7 " ], linetype = " dashed " ) +
geom_text( data = dat_ann , aes( x = x , y = y , label = label ),
size = 4 , vjust = - 0.5 , hjust = 0 , color = " orange " , parse = TRUE )
fig group_var . نود هنا أن نعرف ما إذا كانت هناك بعض الأصفار الهيكلية عبر مستويات delivery المختلفة library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ 1 | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )group_var . نود هنا أن نعرف ما إذا كانت هناك بعض الأصفار الهيكلية عبر مستويات delivery المختلفة library( readr )
library( tidyverse )
otu_data = read_tsv( " data/ecam-table-taxa.tsv " , skip = 1 )
otu_id = otu_data $ `feature-id`
otu_data = data.frame ( otu_data [, - 1 ], check.names = FALSE )
rownames( otu_data ) = otu_id
meta_data = read_tsv( " data/ecam-sample-metadata.tsv " )[ - 1 , ]
meta_data = meta_data % > %
rename( Sample.ID = `#SampleID` ) % > %
mutate( month = as.numeric( month ))
source( " programs/ancom.R " )
# Step 1: Data preprocessing
feature_table = otu_data ; sample_var = " Sample.ID " ; group_var = " delivery "
out_cut = 0.05 ; zero_cut = 0.90 ; lib_cut = 0 ; neg_lb = TRUE
prepro = feature_table_pre_process( feature_table , meta_data , sample_var , group_var ,
out_cut , zero_cut , lib_cut , neg_lb )
feature_table = prepro $ feature_table # Preprocessed feature table
meta_data = prepro $ meta_data # Preprocessed metadata
struc_zero = prepro $ structure_zeros # Structural zero info
# Step 2: ANCOM
main_var = " delivery " ; p_adj_method = " BH " ; alpha = 0.05
adj_formula = " month " ; rand_formula = " ~ month | studyid "
lme_control = list ( maxIter = 100 , msMaxIter = 100 , opt = " optim " )
res = ANCOM( feature_table , meta_data , struc_zero , main_var , p_adj_method ,
alpha , adj_formula , rand_formula , lme_control )