
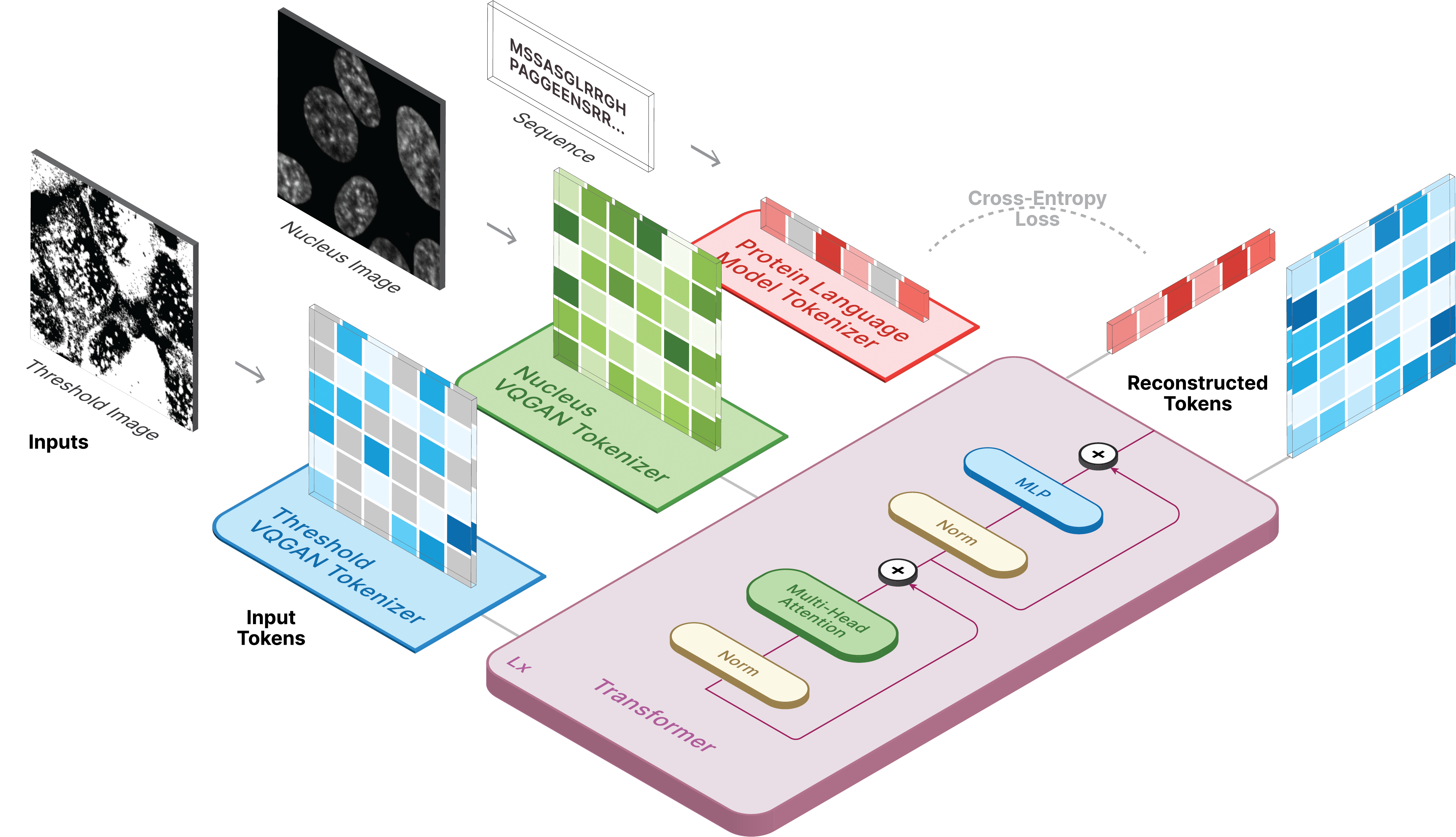
يعد هذا المستودع التطبيق الرسمي لـ CELL-E 2: ترجمة البروتينات إلى صور وإعادتها باستخدام محول ثنائي الاتجاه لتحويل النص إلى صورة.
إنشاء بيئة افتراضية وتثبيت الحزم المطلوبة عبر:
pip install -r requirements.txt
بعد ذلك، قم بتثبيت torch = 2.0.0 بإصدار CUDA المناسب
النماذج متوفرة على Hugging Face.
لدينا أيضًا مساحتان متاحتان حيث يمكنك تشغيل التنبؤات على بياناتك الخاصة!
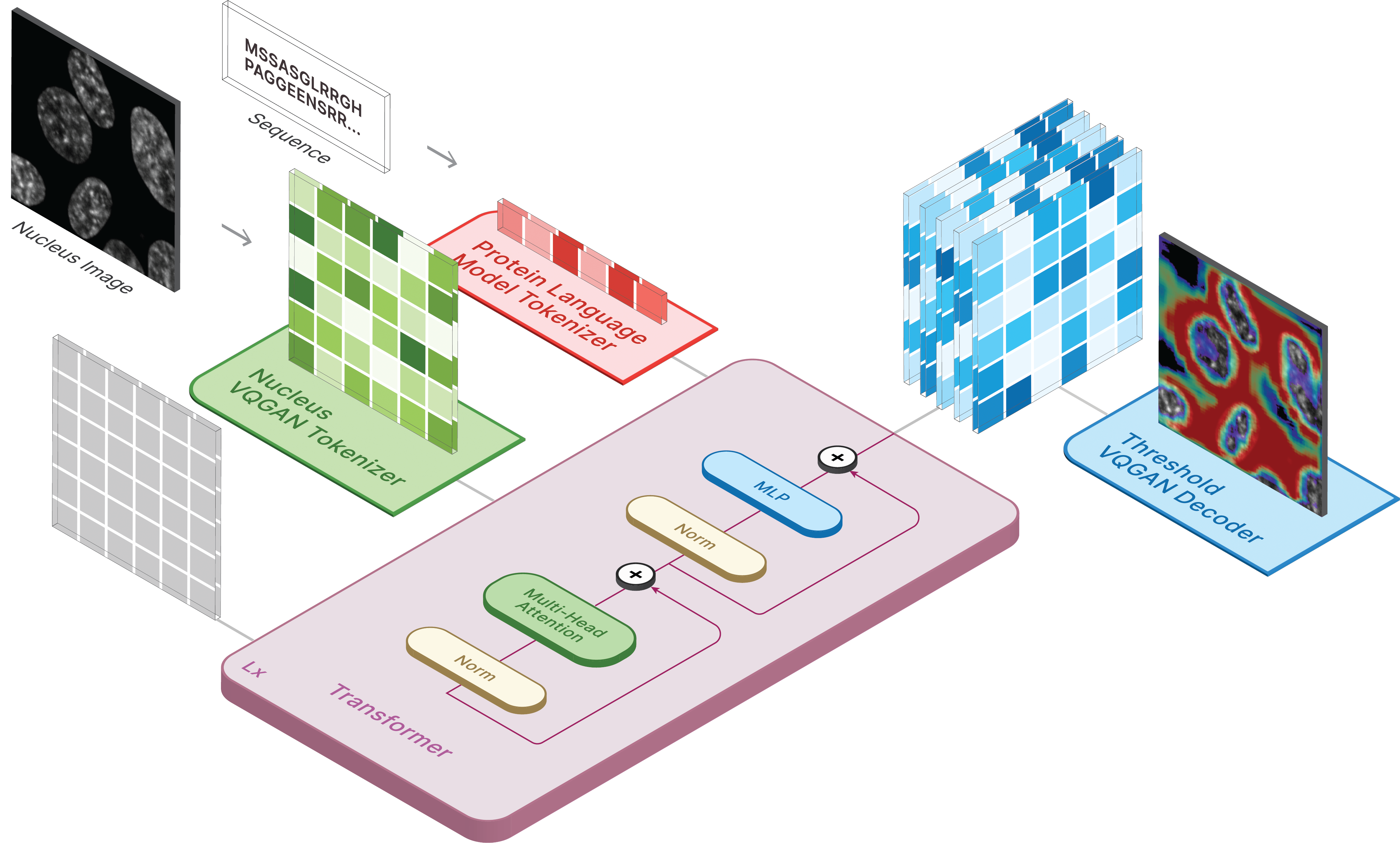
لإنشاء صور، قم بتعيين النموذج المحفوظ على أنه ckpt_path. يمكن أن تكون هذه الطريقة غير مستقرة، لذا راجع Demo.ipynb لمعرفة طريقة أخرى للتحميل.
from omegaconf import OmegaConf
from celle_main import instantiate_from_config
configs = OmegaConf . load ( configs / celle . yaml );
model = instantiate_from_config ( configs . model ). to ( device );
model . sample ( text = sequence ,
condition = nucleus ,
return_logits = True ,
progress = True ) model . sample_text ( condition = nucleus ,
image = image ,
return_logits = True ,
progress = True )يتم التدريب على CELL-E على ثلاث مراحل:
في حالة استخدام صورة عتبة البروتين، قم بتعيين threshold: True لمجموعة البيانات.
نحن نستخدم نسخة معدلة قليلاً من كود ترويض المحولات.
للتدريب، قم بتشغيل البرنامج النصي التالي:
python celle_taming_main.py --base configs/threshold_vqgan.yaml -t True
يرجى الرجوع إلى الريبو الأصلي للحصول على علامات إضافية، مثل --gpus .
نحن نقدم برامج نصية لتنزيل صور Human Protein Atlas وOpenCell في مجلد البرامج النصية. هناك حاجة إلى data_csv لمحمل البيانات. يجب عليك إنشاء ملف CSV يحتوي على الأعمدة nucleus_image_path و protein_image_path و metadata_path و split (train أو val) sequence (اختياري). من المفترض أن هذا الملف موجود داخل نفس مجلد data العامة مثل ملفات الصور والبيانات التعريفية.
البيانات الوصفية عبارة عن ملف JSON يجب أن يصاحب كل تسلسل بروتيني. إذا لم يظهر التسلسل في data_csv ، فيجب أن يظهر في metadata.json مع المفتاح المسمى protein_sequence .
يمكن أن تكون إضافة المزيد من المعلومات هنا مفيدة للاستعلام عن البروتينات الفردية. يمكن استرجاعها عن طريق retrieve_metadata ، الذي يقوم بإنشاء متغير self.metadata داخل كائن مجموعة البيانات.
للتدريب، قم بتشغيل البرنامج النصي التالي:
python celle_main.py --base configs/celle.yaml -t True
حدد --gpus بنفس تنسيق VQGAN.
يحتوي CELL-E على الخيارات التالية:
ckpt_path : استئناف تدريب CELL-E 2 السابق. النموذج المحفوظ باستخدامstate_dictvqgan_model_path : نموذج صورة البروتين المحفوظ (مع State_dict) لتشفير صورة البروتينvqgan_config_path : نموذج صورة البروتين المحفوظ yamlcondition_model_path : نموذج الحالة (النواة) المحفوظ (مع State_dict) لتشفير صورة البروتينcondition_config_path : نموذج الحالة المحفوظة (النواة) yamlnum_images : 1 في حالة استخدام أداة تشفير صور البروتين فقط، و2 في حالة تضمين أداة تشفير الصور الشرطيةimage_key : nucleus أو target أو thresholddim : أبعاد تضمين نموذج اللغةnum_text_tokens : إجمالي عدد الرموز المميزة في نموذج اللغة (33 لـ ESM-2)text_seq_len : العدد الإجمالي للأحماض الأمينية التي تم النظر فيهاdepth : عادة ما يكون عمق نموذج المحول، الأعمق أفضل على حساب VRAMheads : عدد الرؤوس المستخدمة في الاهتمام متعدد الرؤوسdim_head : حجم رؤوس الانتباهattn_dropout : معدل ترك الانتباه في التدريبff_dropout : معدل التسرب من التغذية إلى الأمام في التدريبloss_img_weight : الوزن المطبق على إعادة بناء الصورة. وزن النص = 1loss_text_weight : يتم تطبيق الترجيح على إعادة بناء الصورة الشرطية.stable : الأوزان القياسية (عند حدوث تدرجات متفجرة)learning_rate : معدل التعلم لمحسن آدمmonitor : Param يستخدم لحفظ النماذج يرجى ذكرنا إذا قررت استخدام الكود الخاص بنا في أي جزء من بحثك.
@inproceedings{
anonymous2023translating,
title={CELL-E 2: Translating Proteins to Pictures and Back with a Bidirectional Text-to-Image Transformer},
author={Emaad Khwaja, Yun S. Song, Aaron Agarunov, and Bo Huang},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=YSMLVffl5u}
}